|
| 细菌全基因组从头测序(Bacterial Whole Genome de novo Sequencing),是指对基因组序列未知或没有近缘物种基因组信息的某个物种,构建不同插入片段长度的基因组文库并进行序列测定,利用生物信息学方法进行组装、基因预测和功能注释,从而获得细菌的全基因组序列图谱。
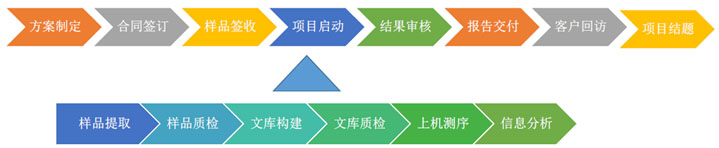
关键词:细菌基因组;第二代高通量测序;第三代单分子测序;从头测序;进化;环境适应性 服务流程
测序策略 采用第三代单分子测序平台为主、第二代高通量测序平台为辅的测序策略获得细菌的全基因组序列图谱。
样品要求
测序平台
生物信息分析 提供 34 项基本分析内容;根据客户需求定制个性化分析方案
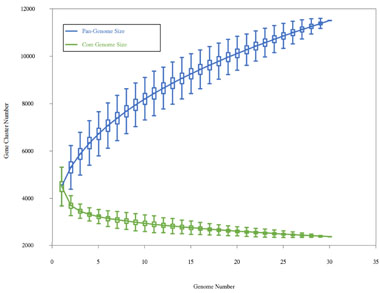
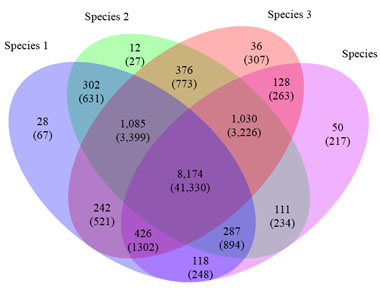
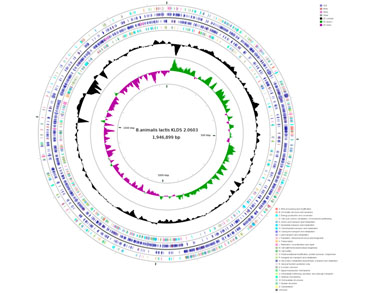
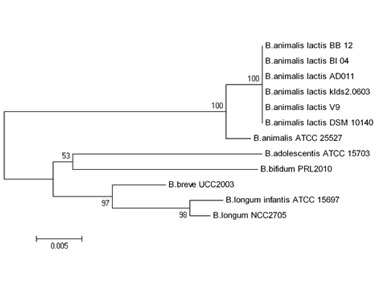
A+B: 精细图和完成图标准分析内容; C: 高级信息分析内容。 结果展示
项目交付
研究思路 对于细菌基因组从头测序,文献中的热点研究主题包括:
Q & A 细菌完成图仅用第三代单分子测序平台是否可行? 不可行。小质粒(20 kb)的片段有可能在建库过程中丢失;染色体的部分区域也有可能会因为取样的概率问题或样品的降解问题而没有被测序到。 第三代单分子测序平台的单碱基准确率低,如何保证拼接结果的准确性? 第三代单分子测序平台测序数据的单碱基准确率仅为 87~92%。通过三个步骤来确保拼接结果的准确性:在拼接前,利用第三代单分子测序序列间的 Overlap 对测序产生的数据进行校正;拼接后,利用第三代单分子测序序列对拼接的结果进行校正;第二步校正后,进一步利用高质量的第二代高通量测序数据对拼接结果进行校正。通过这三步校正,最终拼接结果的准确率可达到99.99% 以上。 项目周期如何计算? 微生物基因组测序框架图的项目周期为 25 个工作日,完成图的项目周期为 40 个工作日。工作日一般从样品提取检测合格且预付款到账后开始计算。个性化分析内容的项目周期需要视具体情况而定。 如何保证 0 Gap? 目前,采用二代和三代测序平台结合的方案,90% 以上的菌株能直接拼接得到完整的全基因组序列图谱。剩余的 10% 左右的菌株我们一般会利用一代测序平台来填补Gap。 |
|