
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于蛋白质语言模型ESM-2的SARS-CoV-2宿主因子预测框架TransFactor及其在抗病毒治疗中的应用
【字体: 大 中 小 】 时间:2025年09月11日 来源:Bioinformatics 5.4
编辑推荐:
本研究针对病毒宿主因子鉴定实验方法存在的高假阳性/阴性率问题,开发了基于ESM-2蛋白质语言模型的TransFactor框架。通过整合33项SARS-CoV-2研究的实验数据,该模型仅需蛋白序列即可预测促病毒宿主因子,其性能(AUROC 0.89)显著优于传统机器学习方法,并通过计算丙氨酸扫描识别出COMM/PX/RRM等关键功能域,为抗病毒药物靶点发现提供了新范式。
病毒与宿主的博弈从未停歇,从2016年寨卡病毒到COVID-19大流行,人类不断遭遇新发传染病的挑战。病毒如同精密的"分子海盗",劫持宿主细胞机器完成其生命周期。传统抗病毒药物多靶向病毒自身蛋白,但病毒快速突变导致的耐药性促使科学家将目光转向宿主因子——这些被病毒"绑架"的细胞蛋白犹如"叛徒",协助病毒完成入侵、复制和释放。然而现有宿主因子鉴定主要依赖CRISPR-Cas9或RNAi筛选,这些高通量方法存在细胞系限制、结果重复性差等缺陷,犹如在黑暗森林中盲目射击,假阳性率高达40%(Baggen et al., 2021)。
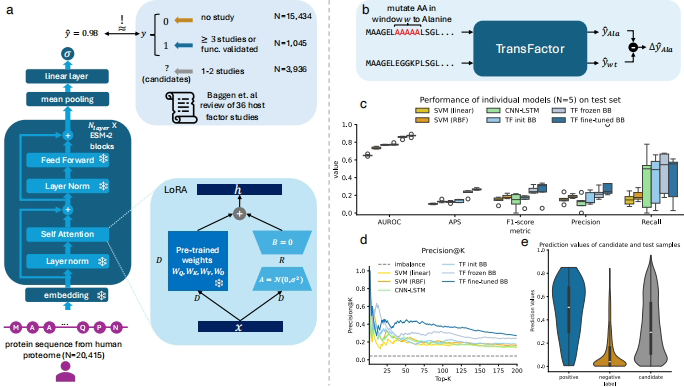
为突破这一瓶颈,Helmholtz Munich研究所的团队在《Bioinformatics》发表创新研究,开发出TransFactor预测框架。该研究巧妙利用蛋白质语言模型ESM-2的预训练知识,仅需蛋白序列即可预测SARS-CoV-2宿主因子,避免了繁琐的组学实验。通过整合33项独立研究的实验数据构建金标准数据集,采用低秩自适应(LoRA)微调策略,模型在测试集达到AUROC 0.89的优异性能。更引人注目的是,通过计算丙氨酸扫描技术,首次系统识别出COMM、PX和RRM等结构域可能是介导促病毒功能的关键"分子开关"。
关键技术方法
研究从UniProtKB获取20,415条人类蛋白序列,基于33项SARS-CoV-2研究筛选出1,038个阳性宿主因子。采用ESM-2模型提取蛋白特征,创新性结合LoRA微调策略降低计算成本。通过mmseq2聚类(序列相似度10%)确保数据无泄漏,使用Optuna优化超参数。验证阶段采用5折交叉验证,通过计算丙氨酸扫描实现模型可解释性分析,GO富集使用DAVID数据库完成。
研究结果
模型性能验证
TransFactor显著优于传统方法:在测试集AUROC达0.89,较CNN-LSTM模型提升11%。Precision@100达0.37,意味着每验证100个预测靶点可发现37个真实宿主因子,较实验筛选(平均精度0.13)效率提升近3倍。
生物学一致性验证
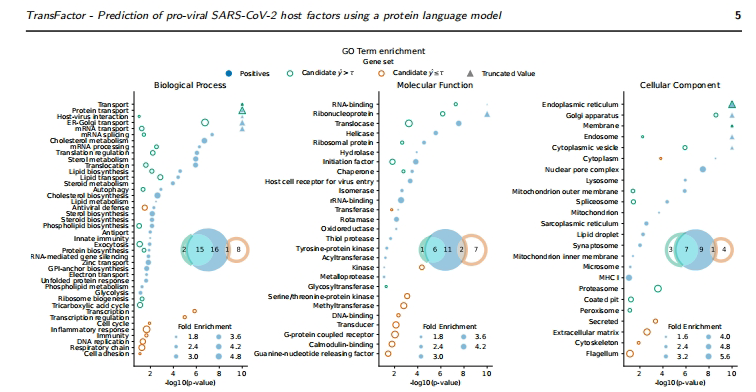
对3,936个候选蛋白的预测显示,高评分候选者与已知宿主因子具有高度一致的GO富集模式(OR=15.0, p<0.0001),如内吞作用调控、RNA结合等通路。这提示模型成功捕捉到宿主因子的功能特征,而非简单记忆序列模式。
关键功能域发现
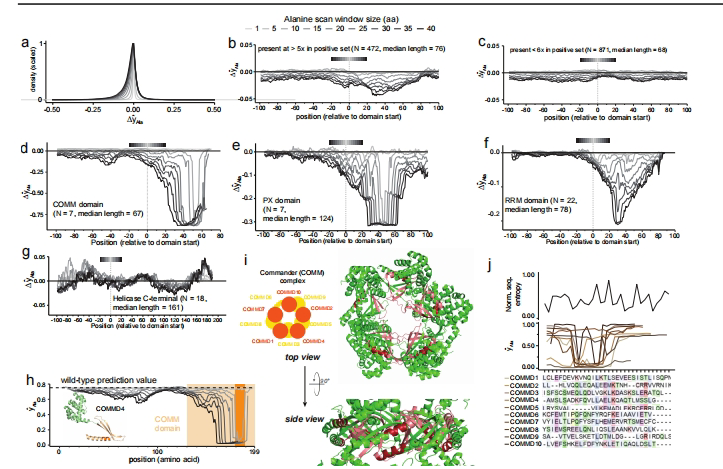
计算丙氨酸扫描揭示:COMM结构域(如COMMD4 172-199位点)的突变导致预测评分下降最显著(Δ?Ala<-0.5),这与COMMD家族蛋白在病毒内体运输中的已知功能吻合。PX和RRM结构域同样被识别为关键区域,而解旋酶结构域等则影响微弱。
研究启示
该研究开创性地将蛋白质语言模型应用于宿主因子预测,其价值体现在三方面:首先,TransFactor仅需序列信息即可预测,解决了新兴病毒研究中组学数据缺失的难题;其次,模型识别出的COMM等结构域为抗病毒药物设计提供了精确靶标;最后,该框架可扩展至其他传染病研究,如对SARS-CoV宿主因子的预测召回率达42%。
局限性在于当前模型仅处理1024个氨基酸以内的序列,且跨病毒属预测性能有待提升。未来结合结构预测模型AlphaFold或能进一步提升精度。这项研究标志着计算生物学在抗病毒领域的重大突破,为应对未来大流行提供了新的"数字武器"。
 生物通微信公众号
生物通微信公众号
知名企业招聘