
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于组合优化算法ALLEGRO的真菌界CRISPR向导RNA跨物种设计研究
【字体: 大 中 小 】 时间:2025年08月21日 来源:Nucleic Acids Research 13.1
编辑推荐:
本研究针对CRISPR-Cas9系统在非模式生物中应用受限的难题,开发了ALLEGRO(Algorithm for a Linear program Enabling Guide RNA Optimization)算法。通过整数线性规划技术,该研究成功设计了靶向2263种真菌转录组的最小化sgRNA文库(仅需9条sgRNA),并在Kluyveromyces marxianus等多物种中验证了编辑效率(90-100%)。这项工作为跨物种基因组编辑提供了通用解决方案,显著推进了比较基因组学和合成生物学研究。
基因组编辑技术CRISPR-Cas9的革命性突破为生命科学研究带来了全新工具,但其应用长期受限于物种特异性设计瓶颈。传统sgRNA设计工具通常仅针对单一物种,面对真菌等高度多样化的生物类群时,研究者不得不为每个物种单独设计向导RNA,这不仅耗时耗力,更阻碍了跨物种比较研究和规模化基因操作。特别是在工业微生物改造、病原真菌研究和进化生物学领域,亟需开发能够覆盖整个生物类群的通用型CRISPR工具。
这项发表于《Nucleic Acids Research》的研究提出了创新性解决方案。研究团队开发了ALLEGRO算法,该技术核心在于将sgRNA设计转化为组合优化问题,利用整数线性编程寻找覆盖多物种基因靶点的最小sgRNA集合。通过设置不同设计轨道(track Am和Em)和冗余阈值,系统能智能平衡文库规模与编辑效率。
关键技术方法包括:1)基于DIAMOND的orthogroup分析确定跨物种保守靶点;2)uCRISPR算法预测sgRNA活性;3)Cas9-核糖核蛋白(RNP)递送系统验证跨物种编辑效率;4)利用2263个真菌基因组(来自NCBI、FungiDB等数据库)进行大规模计算验证;5)在Kluyveromyces marxianus等5种酵母中通过原质体转化进行实验验证。
研究结果
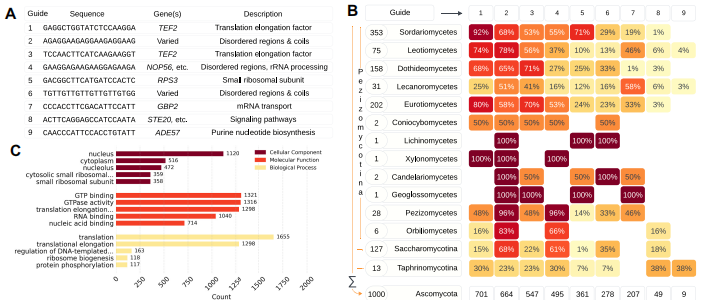
1.九条sgRNA覆盖千种子囊菌转录组
配置A1轨道时,ALLEGRO仅需9条sgRNA即可靶向1000种Ascomycota的全部转录组。如图2所示,首条sgRNA靶向翻译延伸因子TEF2,覆盖92% Sordariomycetes和80% Eurotiomycetes物种。
2.营养缺陷型基因的跨物种靶向
针对CAN1等6个营养缺陷标记基因,E1轨道设计的文库(1485条sgRNA)相较传统方法减少89%。实验验证显示,在Yarrowia lipolytica中TRP1基因编辑效率达100%,显著优于对照组(图4C)。
3.新型真菌的编辑验证
通过Cas9-RNP系统,将文库成功应用于训练集未包含的Rhodotorula araucariae,FCY1和LYS2基因编辑效率分别达85%和78%(图5B),证实了方法的普适性。
讨论与意义
该研究突破了CRISPR技术在非模式生物中应用的三大障碍:首先,ALLEGRO算法通过数学优化将sgRNA文库规模降低1-2个数量级;其次,建立的通用型Cas9-RNP递送体系克服了物种转化效率差异;最后,交叉验证证实90%测试物种可被预设计文库覆盖,为"设计-验证"模式提供了新范式。
这项工作由加州大学河滨分校的Amirsadra Mohseni和Reyhane Ghorbani Nia等学者合作完成,其创新性体现在:1)首次实现真菌界规模的CRISPR工具设计;2)开发了可调节的轨道系统(track Am/Em)满足不同研究需求;3)通过冗余阈值等启发式算法解决超大规模计算难题。这些突破不仅将加速真菌基因组学研究,其方法论更可拓展至植物、动物等真核生物,为合成生物学和精准医学提供跨物种编辑新工具。
 生物通微信公众号
生物通微信公众号
知名企业招聘