
-

生物通官微
陪你抓住生命科技
跳动的脉搏
精准、可扩展的结构变异基因分型:复杂基因组群体尺度分析新工具SVGAP
【字体: 大 中 小 】 时间:2025年07月30日 来源:Molecular Biology and Evolution 11.0
编辑推荐:
研究人员针对复杂植物基因组中结构变异(SV)检测工具适用性有限的问题,开发了SVGAP流程,实现了群体尺度高质量基因组组装的结构变异发现、分型和注释。通过模拟数据和26个玉米基因组的应用验证,该工具在SV检测准确性和完整性上优于现有方法,尤其擅长解析着丝粒区LTR反转座子驱动的基因组多样性,为泛基因组构建和未探索基因组区域的遗传解析提供了新方案。
在基因组学研究领域,结构变异(SV)作为长度超过50bp的DNA序列差异,包括缺失、插入、倒位等类型,长期以来是遗传变异研究中最难表征的部分。尤其在植物基因组中,高度的重复序列和结构复杂性使得SV检测面临巨大挑战。现有工具多针对特定比对器或动物基因组优化,难以应对大规模植物基因组数据的分析需求,这严重限制了科学家对着丝粒等复杂区域遗传多样性的认知。
针对这一技术瓶颈,华南农业大学园艺学院农业农村部华南地区园艺作物生物学与种质创制重点实验室的研究团队开发了SVGAP(Structural Variants Genotyping of Assemblies on Population scales)流程。这项发表在《Molecular Biology and Evolution》的研究,通过创新的全基因组比对分析框架,实现了群体尺度基因组组装的结构变异系统性检测。
研究采用多步骤生物信息学方法:首先评估14种全基因组比对工具在植物基因组中的性能,筛选出Last等6种最优比对器;其次基于UCSC工具构建比对链(chain)和网(net)结构识别SV;进而开发合并算法整合跨样本SV数据集;最后通过局部比对重新分型生成标准VCF文件。关键技术包括RSVSim模拟基准数据集、SynNetFilter.pl过滤旁系同源比对,以及CRM(Centromeric Retrotransposon of Maize)特异性注释模块。
研究结果部分:
比对器性能评估:测试显示Last在植物基因组比对中综合表现最优,其与SVGAP组合对水稻SV检测的F1值达99.4%,而Lastz在复杂基因组中表现最差。
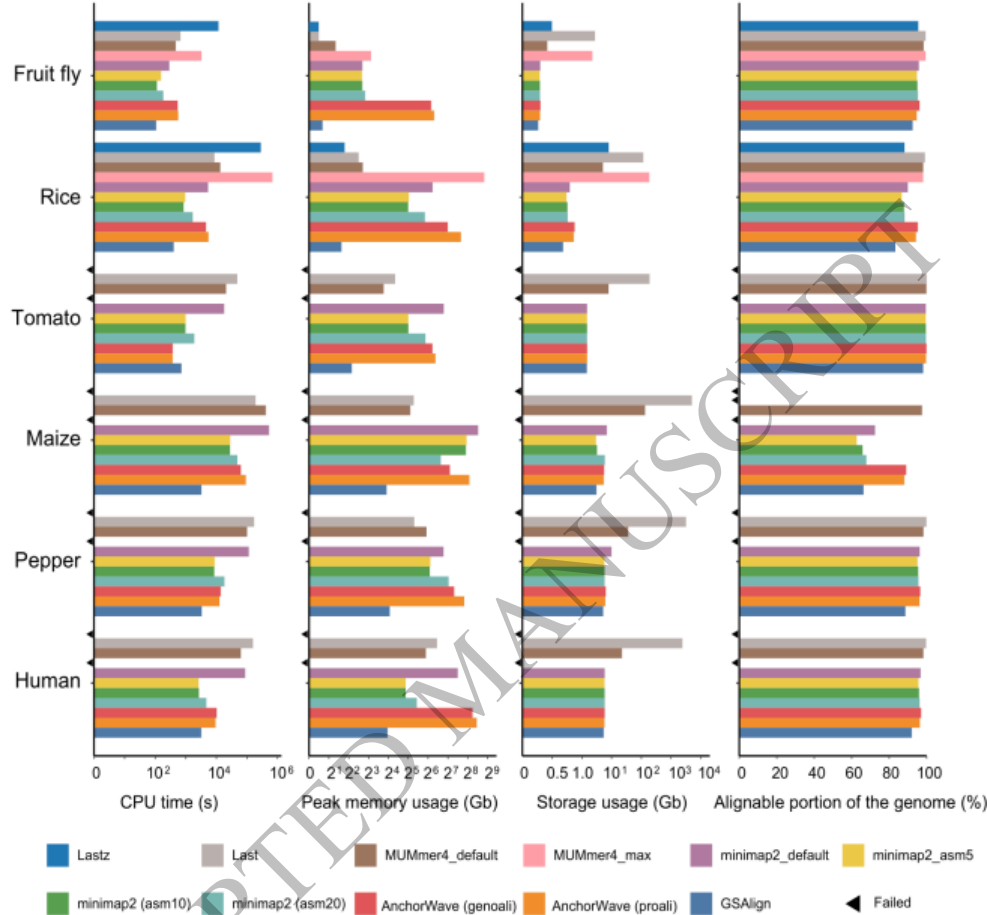
跨工具比较:SVGAP在检测灵敏度上显著优于SyRI等7种工具,尤其在长片段SV(>5kb)识别方面,AnchorWave虽检出数量最多但假阳性率较高。
群体分型验证:模拟21个水稻基因组测试显示,SVGAP合并步骤可恢复99.8%的模拟SV,分型准确率达98.2%,错误率仅0.6%。
玉米基因组应用:分析26个玉米品系发现251,807个插入和89,342个缺失,数量远超既往研究。特别在着丝粒区域(如Cen3)发现267个CRM插入事件,揭示转座子驱动局部SV多样性突破传统SNP分析局限。
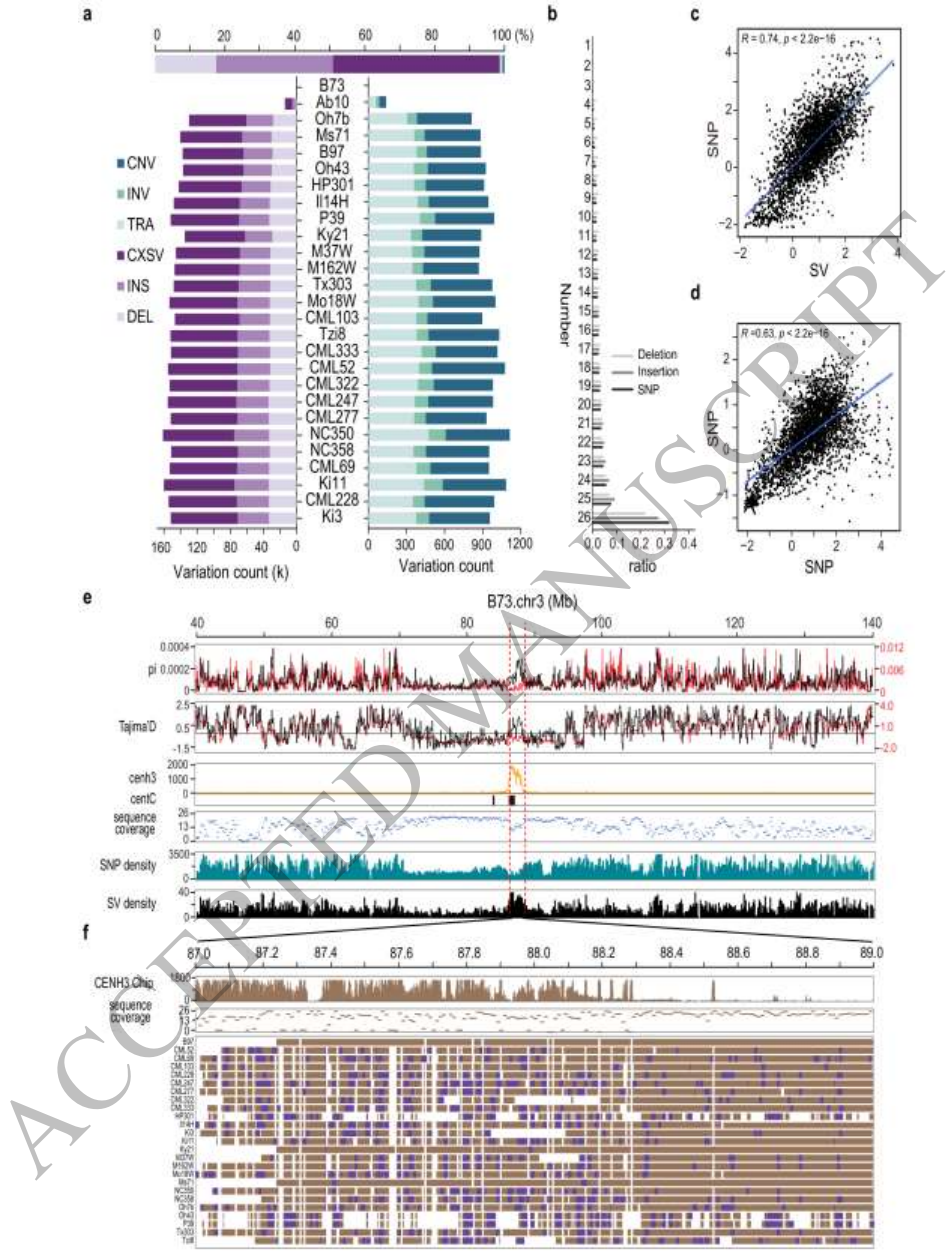
结论部分强调,SVGAP解决了植物基因组结构变异研究的三大核心问题:1)突破比对器依赖限制,支持多种全基因组比对工具输入;2)首次实现组装基因组样本的群体水平SV分型;3)通过TE注释等功能解析SV形成机制。该工具在玉米着丝粒区发现的大量CRM插入事件,为解释"着丝粒悖论"(高重组抑制与局部变异积累并存)提供了新证据。研究建立的标准化分析流程(MIT开源许可)将推动作物育种和进化研究中"暗物质"变异的挖掘,特别适用于番茄、胡椒等复杂基因组的泛基因组构建。
未来改进方向包括:开发植物特异的比对算法优化复杂SV检测,建立标准植物SV基准数据集,以及扩展对嵌套转座子等复合变异的注释能力。该研究为探索基因组"最后一公里"的遗传多样性设立了新标准。
 生物通微信公众号
生物通微信公众号
知名企业招聘