
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于PacBio HiFi和Hi-C测序的芒果天牛染色体水平基因组组装揭示其宿主适应机制
【字体: 大 中 小 】 时间:2025年07月02日 来源:Scientific Data 5.8
编辑推荐:
本研究针对全球重要蛀干害虫芒果天牛(Batocera rufomaculata)的基因组基础研究空白问题,通过整合PacBio HiFi、Illumina和Hi-C测序技术,首次完成其338.08 Mb染色体水平基因组组装(包含10条假染色体,N50达37.00 Mb),鉴定出17,887个预测基因和27.89%重复序列。该研究为揭示鞘翅目害虫的宿主适应性进化机制提供了关键基因组资源,并为开发RNAi和CRISPR/Cas9等靶向防控策略奠定基础。
芒果天牛(Batocera rufomaculata)作为长角象甲科(Lamiinae)的典型代表,是全球范围内危害超过50种落叶乔木的重要蛀干害虫,对芒果、榴莲等经济作物造成严重损失。这种害虫的幼虫在树干内部蛀食长达一年,成虫继续啃食嫩枝树皮,传统物理防治手段效率低下。尽管其生态经济影响显著,但关于其宿主适应性的基因组机制始终未获解析。
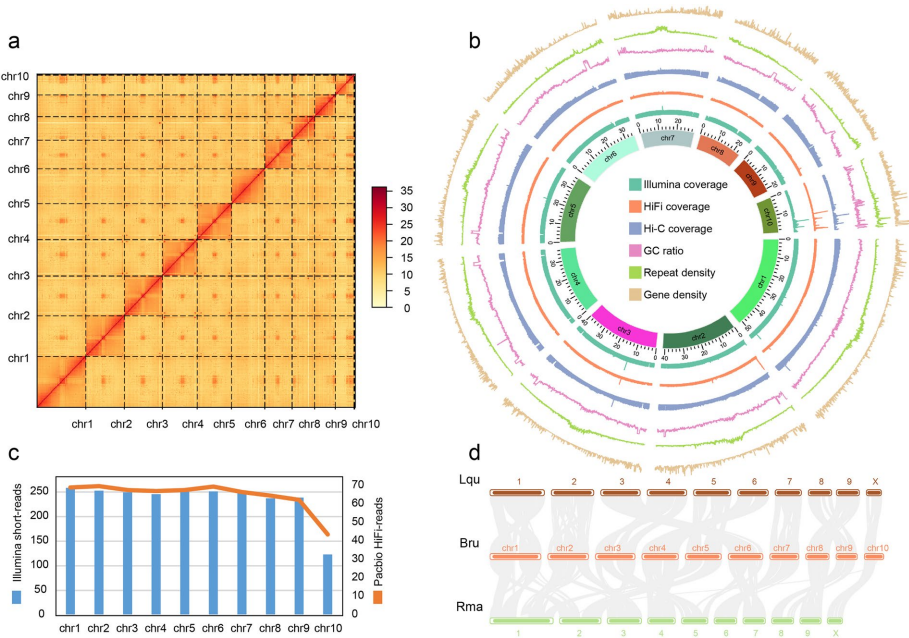
为填补这一空白,中国科学院昆明动物研究所联合云南省农业科学院的研究团队,采用多组学技术首次构建了芒果天牛染色体水平基因组。研究采集自云南保山芒果园的样本,通过PacBio Revio平台生成21 Gb HiFi数据(Q20以上质量),结合61 Gb Hi-C数据和89 Gb Illumina短读长数据,使用hifiasm和3D-DNA流程完成组装。最终获得338.08 Mb基因组(与k-mer估计的325.90 Mb接近),包含10条假染色体(99.77%序列锚定),其中chr10通过共线性分析和测序深度验证为X染色体。
重复序列注释
通过整合de novo预测和同源比对方法,发现基因组中27.89%为重复序列(94.29 Mb),其中转座元件(TE)占24.93%,以DNA转座子(7.53%)和LTR反转录转座子(3.45%)为主。
基因预测与功能注释
基于RNA-seq、同源比对和de novo预测,共鉴定17,887个蛋白编码基因,其中12,729个(71.16%)获得功能注释。BUSCO评估显示基因组完整性达99.3%,基因区完整性97.2%。特别值得注意的是,与鞘翅目近缘物种(如光肩星天牛Anoplophora glabripennis)的共线性分析证实了基因组组装质量。
技术方法亮点
该研究创建的基因组资源为解析芒果天牛的生态适应性提供了分子基础,其揭示的转座元件扩张模式可能与其广谱宿主适应性相关。研究者特别指出,X染色体的鉴定为研究鞘翅目性染色体演化提供了新线索。更重要的是,这一高质量基因组将助力开发基于RNAi的靶向基因沉默技术,例如针对关键解毒酶P450基因的干扰策略,为突破当前害虫防控瓶颈提供新思路。
研究团队在讨论部分强调,这是首个结合HiFi和Hi-C数据的鞘翅目蛀干害虫染色体水平基因组,其97.1%的BUSCO完整性和>99.5%的读段比对率设定了高质量标准。未来可通过比较基因组学进一步挖掘与木质素降解、宿主识别相关的基因家族扩张事件。该成果发表于《Scientific Data》时,审稿人高度评价其"为理解长角象甲科害虫的基因组演化提供了里程碑式资源"。
 生物通微信公众号
生物通微信公众号
知名企业招聘