
-

生物通官微
陪你抓住生命科技
跳动的脉搏
参考引导迭代优化策略提升纳米孔测序碱基识别精度以推动治疗性RNA质量控制新标准
【字体: 大 中 小 】 时间:2025年10月02日 来源:Communications Biology 5.1
编辑推荐:
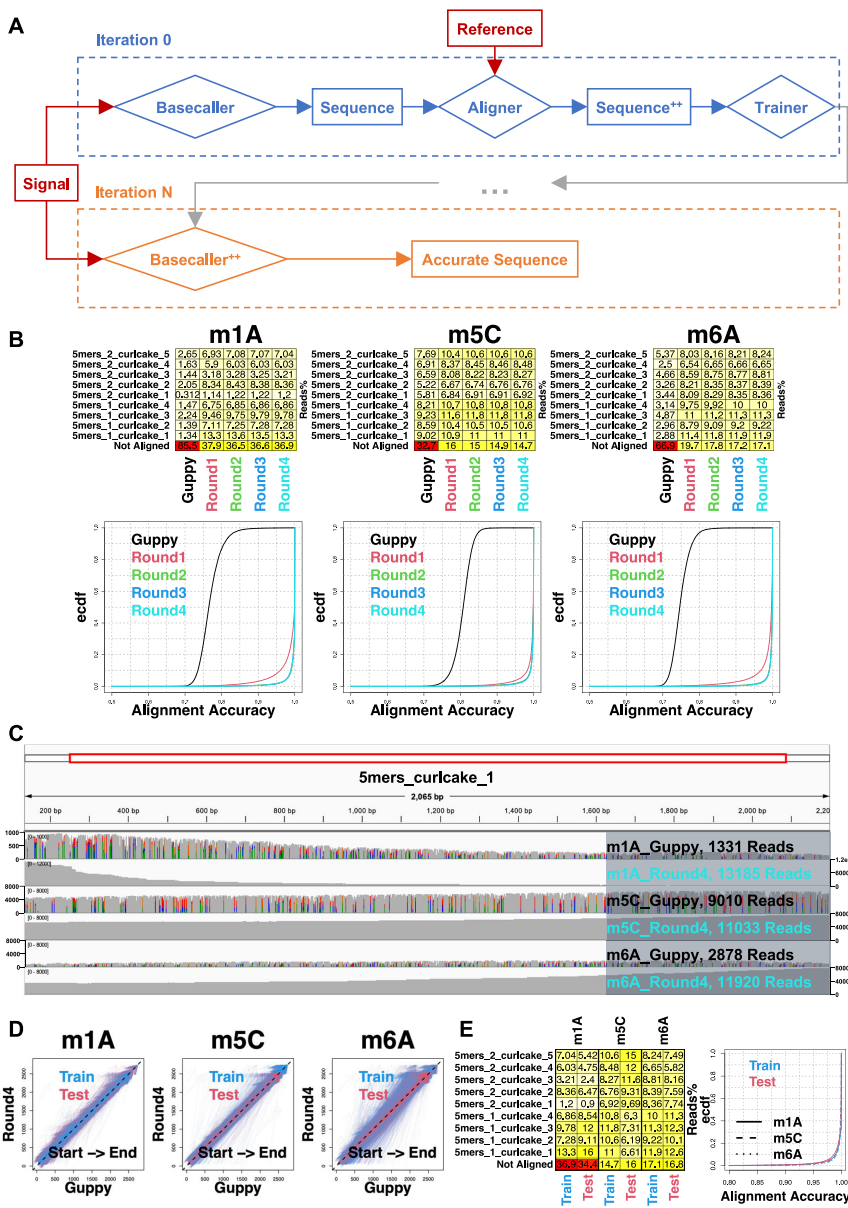
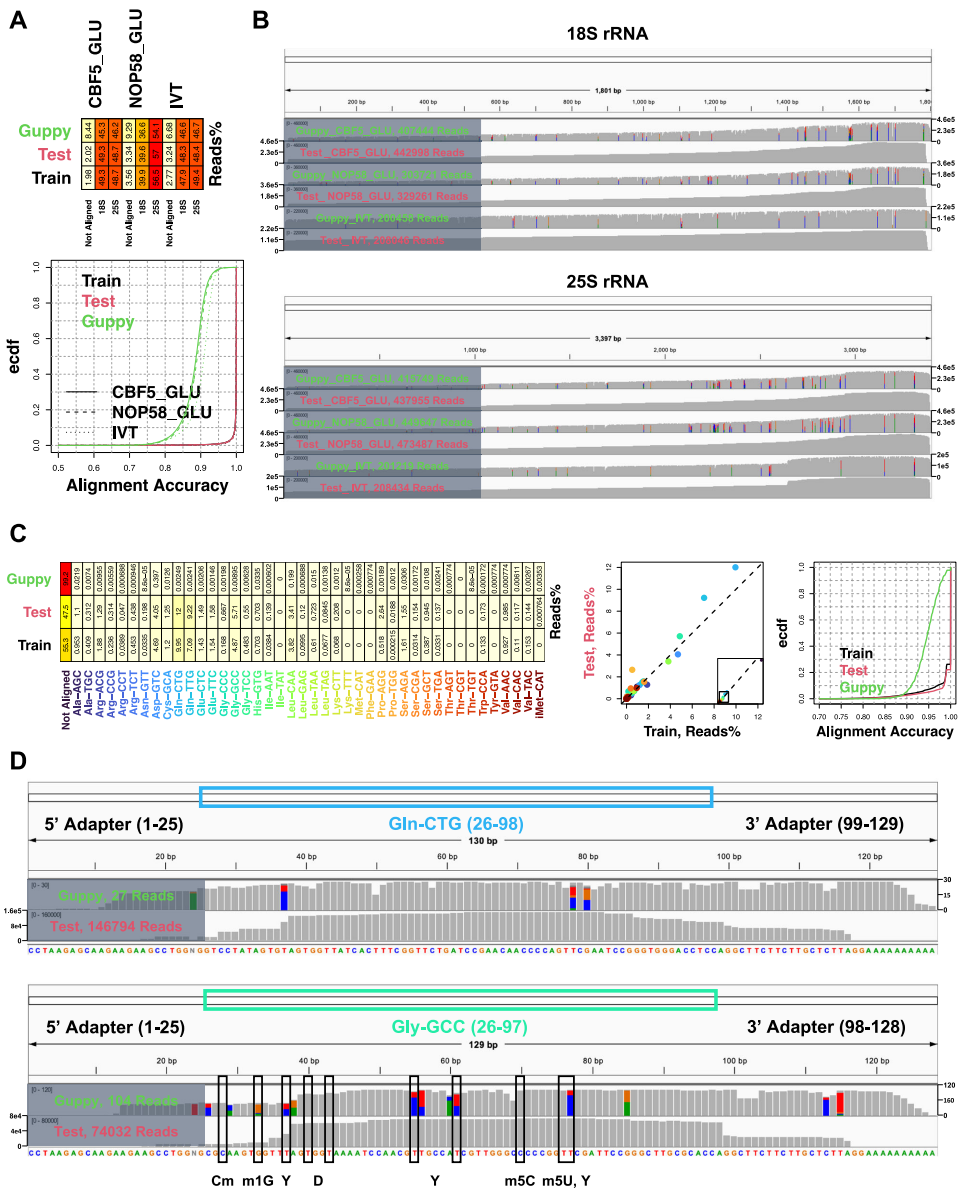
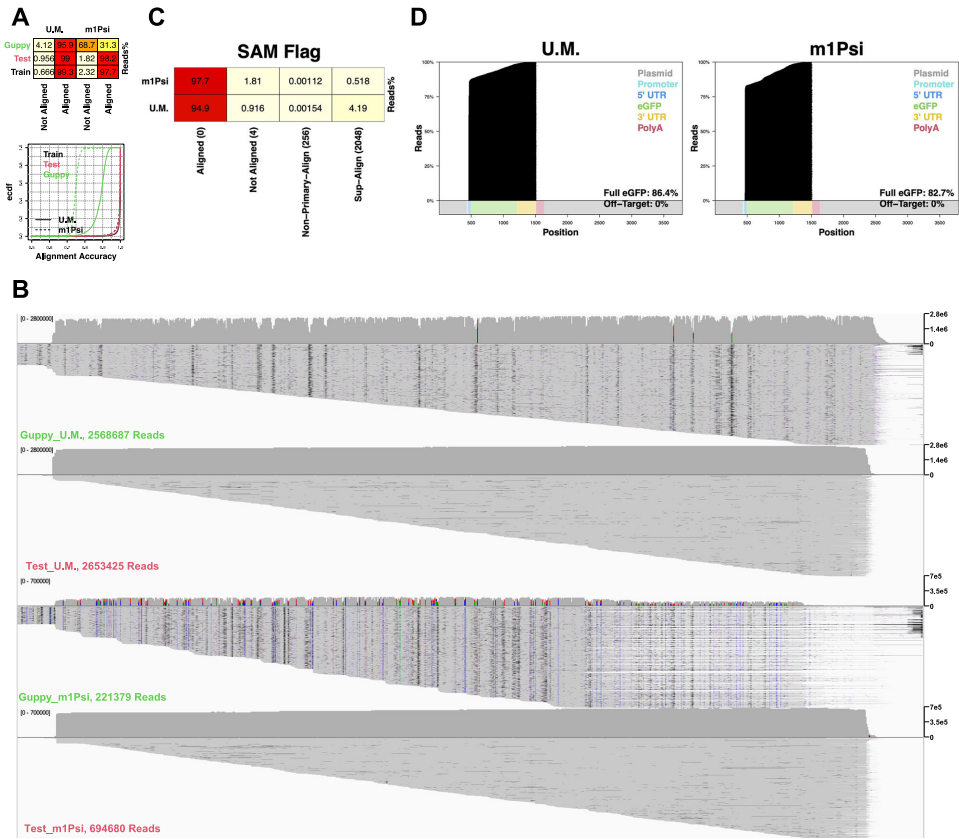
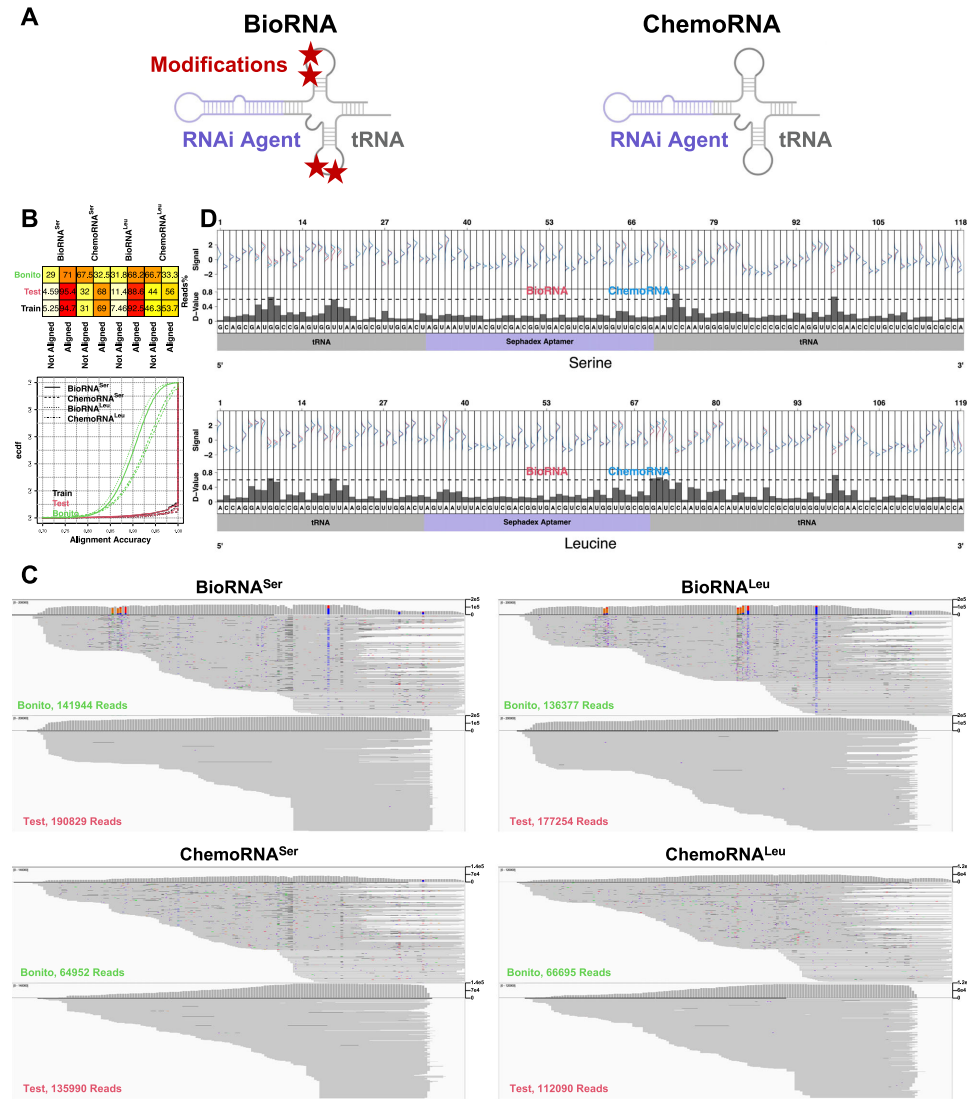
本研究针对纳米孔测序中核苷酸修饰干扰碱基识别准确性的技术瓶颈,开发了一种参考引导的迭代式碱基识别优化方法(iterative basecalling)。该研究通过训练生物分子特异性的高精度碱基识别模型,成功实现了对人工修饰mRNA疫苗和天然修饰BioRNA的精确序列解析,将治疗性RNA质量控制从序列层面提升至修饰状态层面,为RNA药物的质量评估提供了突破性技术方案。
 生物通微信公众号
生物通微信公众号
知名企业招聘