
-

生物通官微
陪你抓住生命科技
跳动的脉搏
陶生策团队发表关于噬菌体免疫沉淀测序技术的综述
【字体: 大 中 小 】 时间:2024年08月30日 来源:上海交大系统生物医学研究院
编辑推荐:
2024年8月19日,上海交大系统生物医学研究院陶生策团队在Molecular & Cellular Proteomics杂志在线发表了题为“Phage immunoprecipitation and sequencing—a versatile technique for mapping the antibody reactome”的综述文章,总结了噬菌体免疫沉淀测序(PhIP-seq)领域的现状,为开展PhIP-seq研究提供了有力支撑
2024年8月19日,上海交大系统生物医学研究院陶生策团队在Molecular & Cellular Proteomics杂志在线发表了题为“Phage immunoprecipitation and sequencing—a versatile technique for mapping the antibody reactome”的综述文章,总结了噬菌体免疫沉淀测序(PhIP-seq)领域的现状,为开展PhIP-seq研究提供了有力支撑。

抗体能够反映个体的感染、过敏以及自身免疫性疾病等病史,表征抗体反应组对疾病标志物发现和免疫学研究至关重要。PhIP-seq技术通过构建等长且包含重叠序列的噬菌体展示肽库,利用免疫沉淀捕获样品中的抗体和相应的噬菌体,再通过二代测序(NGS)技术对噬菌体展示肽进行解码,从而获得抗体反应组的抗原或表位信息(图)。2011年,H. Benjamin Larman和Stephen J. Elledge等人开发了PhIP-seq技术,使用人蛋白质组肽库对神经系统副肿瘤综合征患者脊髓液样本中的自身抗体进行鉴定,发现并验证了与该疾病有关的多种已知或未见报道的自身抗体。目前,PhIP-seq技术已被应用于表位研究、疫苗学、生物标志物发现、免疫治疗、炎症性疾病、感染性疾病、自身免疫性疾病以及过敏等领域。已建立的噬菌体展示肽库涵盖多个物种。与血清蛋白质组分析(SERPA)和芯片等技术相比,PhIP-seq技术通量更高,成本更低,同时,等长肽段的设计在一定程度上减少了实验过程中的偏倚。
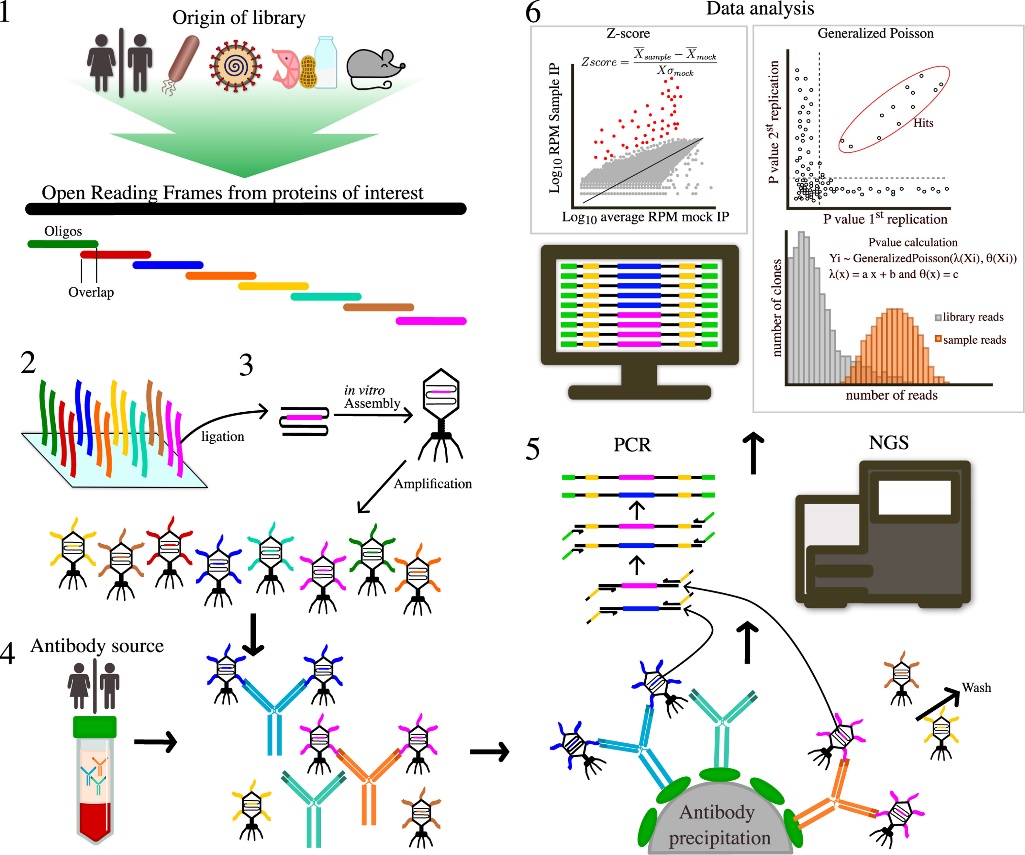
图 PhIP-seq技术路线
1. 将蛋白质的开放阅读框(ORF)分割成等长的肽段,相邻肽段间包含重叠序列从而避免表位破坏;2. 将氨基酸序列转换为DNA序列,合成寡核苷酸;3. 通过连接、包装和扩增,构建噬菌体展示文库;4. 淘选:将文库与血清/血浆/脑脊液样本共孵育,利用抗体结合磁珠(通常为Protein G/Protein A)下拉,洗去未结合的噬菌体;5. 通过PCR扩增核酸并添加Barcode序列,利用NGS解码噬菌体展示肽;6. 数据处理,获得阳性肽
本文整理并总结了PhIP-seq领域现有的噬菌体展示文库(表)、实验设计、数据处理方案与应用,指出了PhIP-seq技术的局限性与未来发展方向。首先,PhIP-seq的测序结果中存在由于非特异性吸附造成的噪音,导致数据处理存在挑战,也对测序深度提出了更高的要求。为了解决这一问题,可尝试从实验或算法层面进行优化。其次,噬菌体展示文库中的肽缺乏翻译后修饰,可能与天然蛋白质存在差异。另外,PhIP-seq技术在发现构象表位方面仍存在困难
稿件来源:陶生策课题组
撰稿:徐昊沄
图文编辑:王华瑶