
-

生物通官微
陪你抓住生命科技
跳动的脉搏
【来自客户的声音】空间成像技术评测
【字体: 大 中 小 】 时间:2024年07月11日 来源:10x Genomics
编辑推荐:
在此,我们将重点介绍近期的两项研究,加拿大和美国的研究人员将10x Genomics公司的Xenium原位分析与NanoString公司的CosMx空间分子成像仪和Vizgen公司的MERSCOPE平台进行了比较。
在选购单细胞空间成像平台时,由独立研究人员领导的分析性能比较研究可能会更加客观并对您有所帮助。在此,我们将重点介绍近期的两项研究,分别来自加拿大Lunenfeld-Tanenbaum研究所的Cook等人(1)和来自美国麻省理工学院和哈佛大学的Broad研究所的Wang等人(2),他们将10x Genomics公司的Xenium原位分析与NanoString公司的CosMx空间分子成像仪和Vizgen公司的MERSCOPE平台进行了比较。
重点归纳
这些研究由不同的独立研究团队开展,对不同的单细胞空间成像平台进行了深入探究,其重点在于:
比较转录本检测的特异性
使用正交技术(RNA-seq和scRNA-seq)验证分析性能
利用共有基因进行横向比较
评估细胞聚类、分割和转录本的空间分布
公开数据以便再次分析


我们在第一部分提炼了这些研究的主要成果,您也可以继续向下阅读,深入了解作者们所做的工作以及他们的成果!
主要成果:
#01-Xenium表现出更高的特异性
Cook等人观察到基因检测转录本与阴性对照探针的比例:Xenium为372:1,CosMx为4.7:1
Wang等人表明,对于测试的每种组织和基因组合类型,Xenium的特异性始终高于MERSCOPE和CosMx(以错误发现率来衡量)
#02-Xenium在正交基准测试中表现出更高的一致性
Cook等人表明,Xenium与snRNA-seq的相关性良好,但CosMx对低表达基因的计数有所夸大
Wang等人指出,Xenium检测到的基因表达水平比MERSCOPE高出2.6-13.6倍;Xenium检测到的表达水平比CosMx高出12.3-14.6倍
#03-Xenium检测到共有基因中的更多转录本
Cook等人观察到,Xenium检测到的每个基因的转录本数量约为CosMx的2倍;若仅限于两个平台间共有的基因,则Xenium检测到的每个细胞的转录本数量约为3倍
Wang等人发现,尽管Xenium的多重分析能力低于CosMx,但对于20种组织类型中的6种,Xenium检测到的独特基因总数多于MERSCOPE和CosMx,且检测到的on-panel基因占比最大
#04-Xenium能够更好地检测和区分细胞类型
Cook等人表明,他们能够通过Xenium分析自信地检测CD8+ T细胞和淋巴细胞,但在CosMx中却无法做到
Wang等人发现:
- Xenium和MERSCOPE都能通过非重叠的标志物基因清晰区分6个细胞谱系,但CosMx只能区分2个谱系
- 在几种组织类型中,Xenium鉴定出的细胞类型始终比CosMx和MERSCOPE更多
比较分析特异性以指导结果解释
为什么特异性很重要?
特异性在本案例中被定义为真正的生物学转录本而不是噪声在检出转录本中所占的比例,它是准确解释灵敏度(见下文)、动态范围及其他分析性能指标的基础。
作者在这些研究中如何使用特异性?
除了靶向感兴趣基因的探针,Xenium原位分析和CosMx SMI还包括阴性对照探针。在Cook等人的研究中,作者通过比较基因靶向探针检测到的转录本中位数与阴性对照探针检测到的转录本中位数的比例来评估特异性。
Wang等人指出,由于Vizgen MERSCOPE默认不包含阴性对照探针,他们必须采取不同的方法。为了确定所有三个平台的特异性,他们首先比较了在靶(on-target)转录本计数与阴性对照条形码(不对应任何基因探针的可检测条形码)的比例。
Wang等人还利用Xenium和CosMx都包含解码对照和阴性对照探针的情况来计算每个平台的错误发现率(FDR)。他们指出,尽管之前的计算在对照和条形码的相对数量方面引入了偏倚,但FDR对这一干扰因素进行了归一化。
研究人员发现了什么?
Cook等人发现,在CosMx SMI中,基因检测转录本中位数与阴性对照探针中位数的比例为4.7:1;而在Xenium分析中,这一比例为372:1(图1)。
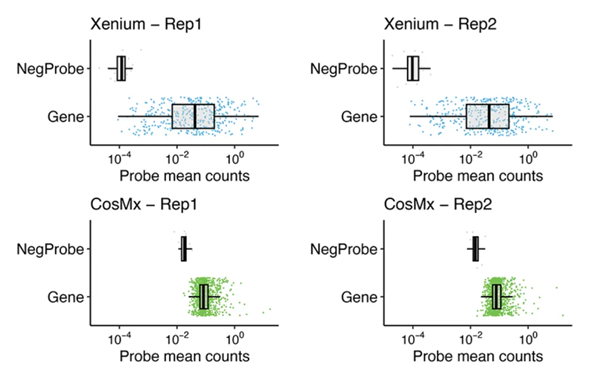
图1. 对Xenium和CosMx SMI之间的在靶转录本检出与阴性对照探针的比例进行比较。图片经过授权,改编自Cook et al., 2023。
同样,Wang等人发现,对于测试的三种组织类型,Xenium和MERSCOPE的在靶检出比例均高于CosMx。对于每种组织和基因组合类型,Xenium的FDR始终比CosMx和MERSCOPE更低(即特异性更高)(图2)。
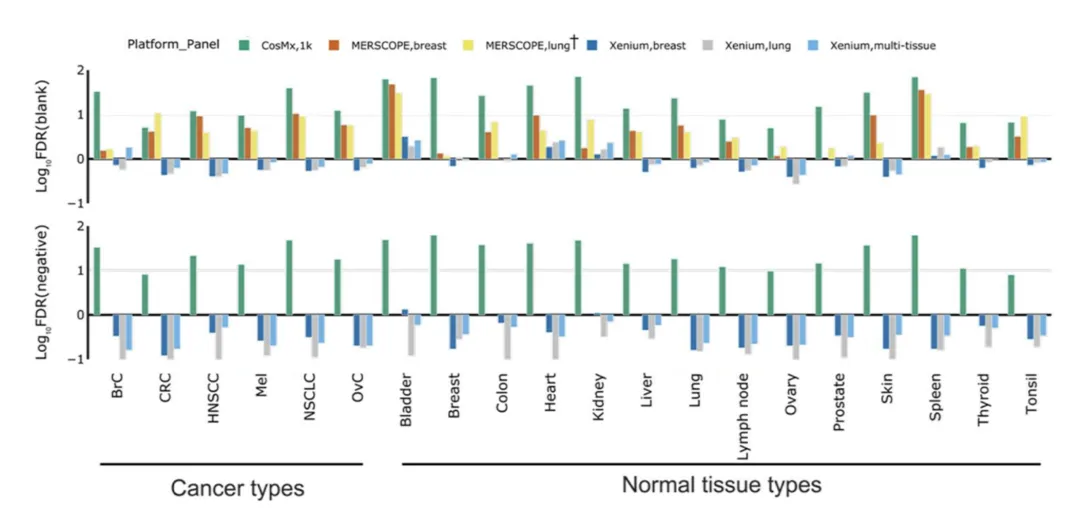
图2. 根据FDR对Xenium、CosMx SMI和MERSCOPE之间的分析特异性进行比较。图片经过授权,改编自Wang et al., 2023。
作者获得了哪些启示?
Xenium和MERSCOPE在多种组织类型中始终表现出高特异性。与MERSCOPE 或Xenium相比,CosMx的各个基因表现出特征性的上升曲线,表明它在低表达情景下有着更频繁的检出。这一点,再加上CosMx在多种组织中的特异性较低且错误发现率高,表明CosMx在检出低表达基因时容易出错。
——Wang等人
利用正交技术进行基准测试以确定真实数据
为什么正交技术很重要?
成熟的正交技术,如RNA-seq或单细胞核RNA测序(snRNA-seq),可以帮助研究人员确保相对基因表达水平、整体灵敏度和确定的分析动态范围反映真实的生物学,而不仅仅是噪声。
作者在这些研究中如何使用正交技术?
Cook等人对与Xenium和CosMx分析所用切片相邻的组织切片进行了snRNA-seq,以开展正交验证。Wang等人从每个平台上运行的组织芯片中生成了pseudobulk基因表达数据,并将其与癌症(TCGA)和正常(GTEx)组织样本的批量细胞RNA-seq数据进行比较,这些样本与空间成像分析所用的癌症和组织类型相匹配。
研究人员发现了什么?
Cook团队发现,Xenium数据在整个动态范围内与snRNA-seq数据的相关性良好,而且与CosMx相比,Xenium数据的动态范围更宽,灵敏度更高(图3,上图)。在检查CosMx数据时,作者指出,“……低表达基因明显膨胀,导致其动态范围被压缩”。
Wang等人强调了Xenium、MERSCOPE、CosMx与批量细胞RNA-seq数据之间的良好相关性(图3,下图)。他们的数据也证明低表达转录本存在同样的潜在问题。他们报告称,“……即使与正交数据相比,CosMx也存在特征性的上升,进一步表明CosMx数据中低表达基因的假阳性率较高”。
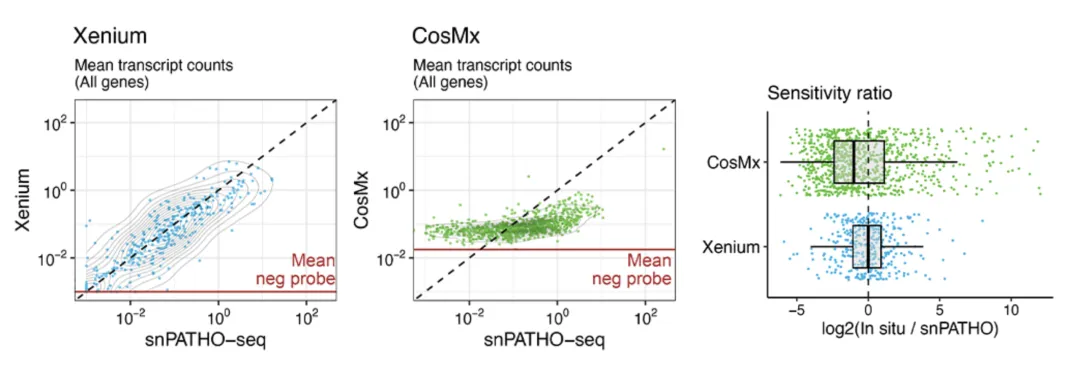
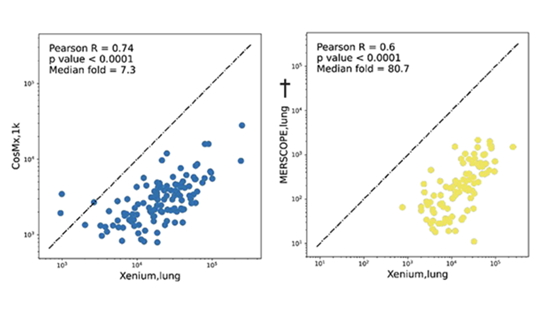
图3. 利用RNA-seq和snRNA-seq对空间成像分析性能进行基准测试。上图:Cook等人强调,Xenium数据在Xenium的整个动态范围内与snRNA-seq数据的相关性良好,而且灵敏度高于CosMx。下图:Wang等人表明,测试的所有三个平台都与RNA-seq数据的相关性良好,但指出CosMx在低表达转录本上表现出特征性的上升曲线,而Xenium检测每个基因时的灵敏度高于另两个平台。图片经过授权,改编自Cook et al., 2023和Wang et al., 2023。
Wang等人还特意比较了不同基因组合的灵敏度。具体来说,他们发现,“Xenium的乳腺基因组合(10 μm)检测的中位数表达比MERSCOPE高2.6倍,肺部基因组合(5 μm)检测的中位数表达高出13.6倍”。与CosMx相比,Xenium的表现更佳,Wang等人指出:
“对于肿瘤TMA中的相同基因,Xenium始终表现出比CosMx更高的表达水平,Xenium乳腺基因组合的计数比CosMx多种组织数据集高出14.6倍。Xenium多种组织基因组合的数据显示出的差异略小,其中相同基因的表达水平高出12.3倍,而肺部基因组合的数据是在切片后最接近的时间点获取的,表达中位数也高出14.0倍。”
作者获得了哪些启示?
我们随后将两个平台的转录本丰度与匹配的snPATHO-seq进行比较。尽管这些计数是整个群体的平均值,但我们观察到Xenium数据在整个检测范围内与snPATHO-seq数据的相关性良好。相比之下,CosMx数据在低表达基因上表现出明显的膨胀,导致其动态范围被压缩。因此,snPATHO-seq数据中相差三个数量级的基因在CosMx数据中表现出相似的检测结果。让情况更加复杂的是高表达基因的灵敏度降低,其回收水平低于在snPATHO-seq中观察到的水平。
——Cook等人
利用重叠基因比较分析性能
为什么重叠基因很重要?
基因组合上的基因总数(多重分析能力)会使某些指标产生偏倚,包括每个细胞检测到的独特基因总数以及(单个细胞和整个组织)检测到的转录本总数,尤其是在作者没有考虑噪声的情况下。在不同平台上分析相同的基因以及高于噪声的转录本有助于更直接地比较分析性能。
作者在这些研究中如何使用重叠基因?
Cook等人明确指出了重叠基因的数量(125个),以及基因组合的总规模(Xenium检测377个基因,CosMx检测960个基因)。在比较可能受到多能分析能力影响的指标(比如每个细胞检测到的独特基因数和每个基因的转录本中位数)时,他们提供了整体数据、按靶点数量归一化的数据,以及仅对共有基因进行比较的数据。
Wang等人将Xenium和MERSCOPE的多个组织基因组合与CosMx进行比较,并指出在他们重点关注的六个基因组合中,共有基因超过94个,而且Xenium和MERSCOPE基因组合之间的相似度很高。与Cook的研究相似,团队也报告了所有基因和共有基因的指标。
研究人员发现了什么?
与预期一致,在Cook的研究中,CosMx基因组合鉴定出的每个细胞的独特基因数要多于Xenium,因为它的多重分析能力更高。然而,在研究所有基因中检测到的每个基因转录本中位数时,Xenium解析的每个基因的转录本数量大约是CosMx的两倍。与这些结果一致,在仅限于基因组合之间共有的125个基因时,Xenium解析的每个细胞的转录本数量也是CosMx的三倍(图4,左图)。
Wang等人的分析结果与Cook研究的结果一致。在比较Xenium、MERSCOPE和CosMx时,若考虑所有基因组合的基因,多重分析能力更高的CosMx基因组合在14种测试组织类型(总共20种)中检测到绝对数量最多的独特基因。然而,Xenium不仅在另外6种样本类型中检测到绝对数量更多的基因,而且在所有20种样本中检测到最高比例的on-panel基因,其次是MERSCOPE或CosMx(具体取决于样本类型)(图4,右图)。
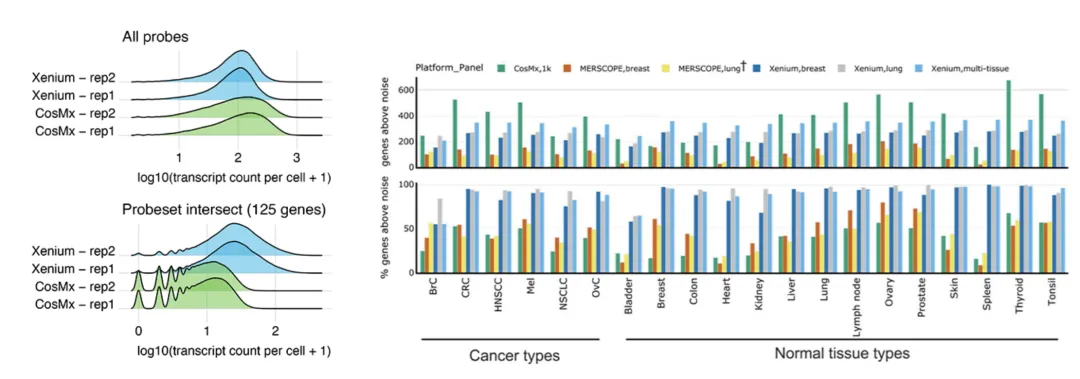
图4. 检测到的转录本绝对数量、检测到的基因比例和检测到的共有基因的性能比较。左图:Cook团队发现,多重分析能力更高的CosMx基因组合检测到的每个细胞中的转录本数量更多,但在只关注两个平台共有的转录本时,Xenium检测到的转录本数量约为CosMx的三倍。右图:Wang等人表明,CosMx和Xenium分别解析了14种和6种组织类型中绝对数量更多的基因。对于所有20种组织类型,Xenium都检测到了更高比例的on-panel基因。图片经过授权,改编自Cook et al., 2023和Wang et al, 2023。
作者获得了哪些启示?
我们发现,当分析仅限于共有基因时,Xenium检测到的每种组织类型的表达水平都较高,且在肿瘤组织或正常组织上的表现没有明显差异。
——Wang等人
比较细胞分割、细胞聚类和空间结构
为什么这很重要?
单细胞空间成像技术需要可靠的细胞分割和转录本检测,以确保您能够捕获单个细胞,并为其分配正确的转录本。
作者在这些研究中如何使用它?
Cook等人利用细胞异质性重点分析了基质,并检查了分割细胞中上皮细胞、成纤维细胞、肌细胞和巨噬细胞等的标志物。他们根据组织中的细胞类型来表征空间结构。最后,他们试图检测一种罕见的细胞类型(CD8 T细胞,通过CD3、CD8A和GZMA的共表达定义)。
Wang等人首先检测了数对非重叠的标志物基因的表达情况,并解释说良好分割的细胞中成对表达标志物基因的细胞比例应当很低。他们还根据经典标志物查看了几种组织类型中每个平台能够分辨的细胞类型。
研究人员发现了什么?
两项研究都强调,Xenium在细胞分割时所用的细胞核边界扩展*所产生的细胞始终比CosMx和MERSCOPE更大。尽管如此,Wang团队发现,Xenium能够清晰分离以非重叠标志物基因表示的细胞谱系(CD19和CD3e表示B细胞和T细胞,CD8和CD4表示T细胞亚群,CD3E和EPCAM表示上皮癌)。尽管MERSCOPE也能分辨这些细胞谱系,但CosMx只能分辨EPCAM和CD3E,而不能分辨其他基因对(作者推测这可能是由于假阳性或细胞分割错误造成的,因为这些免疫基因的计数较低)。
Wang等人发现,Xenium捕捉到的细胞类型始终比CosMx更多(图5)。Xenium从每个乳腺、肺部和乳腺癌样本中鉴定出9种不同的细胞类型。相比之下,CosMx只能分别确定6种、6种和8种细胞类型,而且无法分辨出乳腺和肺部样本中所有已知的主要细胞类型。MERSCOPE从乳腺癌样本中鉴定出6种细胞类型,研究人员指出,“……MERSCOPE和Xenium聚类之间的一对一映射比Xenium和CosMx聚类之间的映射更清晰”。
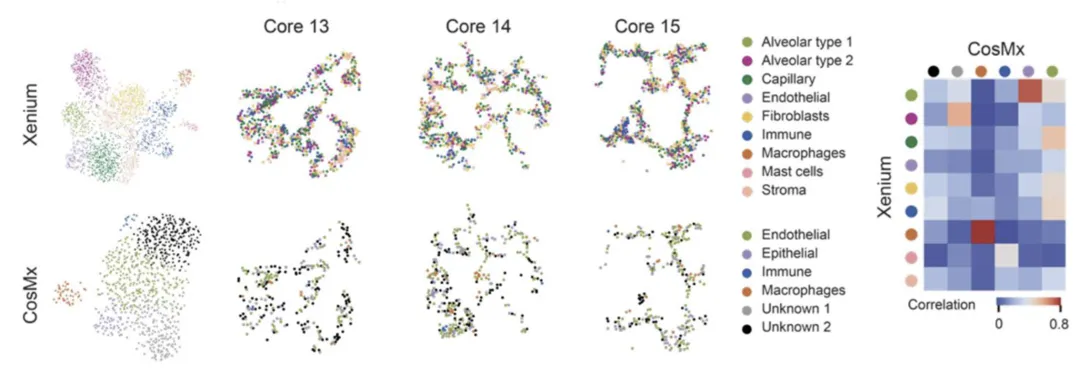
图5. 正常肺部组织中的细胞聚类和细胞类型。Xenium和CosMx回收的细胞类型的UMAP(左图)和空间分布(右图)。图片经过授权,改编自Wang et al., 2023。
以snRNA-seq作为基准真实值,Cook等人发现Xenium数据中肌细胞的巨噬细胞和成纤维细胞标志物的比例高于预期。同样地,他们在周细胞中也发现了内皮细胞、肌细胞和成纤维细胞的标志物,他们推测是邻近细胞污染所致。在CosMx数据中,他们注意到标志物基因的特异性降低,并指出这可能是由于“在靶标志物的丰度较低和噪声水平较高,而不是分割错误”。
最后,Cook等人试图利用Xenium和CosMx检测CD8 T细胞。Xenium鉴定出一个相应群体,但这个群体未出现在CosMx数据中(图6,左图)。此外,淋巴细胞(RNA含量低的小细胞)的标志物在Xenium的组织中呈点状表达,而在CosMx SMI中,这些标志物分布在整个组织中,不足以用于细胞表征(图6,右图)。
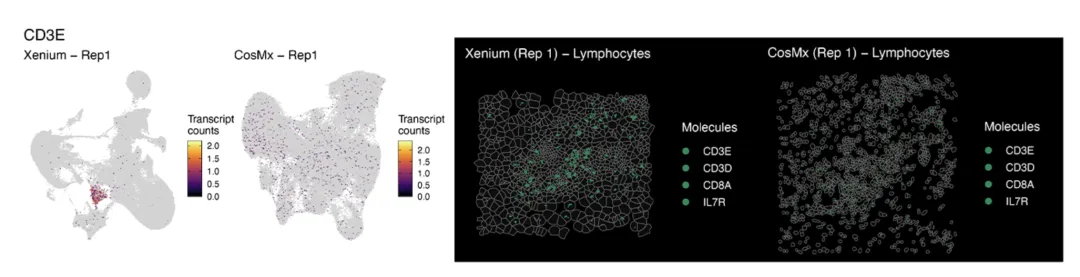
图6. 尝试利用Xenium和CosMx分辨罕见细胞类型。左图:CD3E(CD8 T细胞标志物)在整个细胞簇中的表达。右图:淋巴细胞标志物在相邻切片上的分割细胞中的表达。图片经过授权,改编自Cook et al., 2023。
作者获得了哪些启示?
整体和组织聚类结果表明,CosMx也能识别主要细胞类型,但无法确定细胞亚型。此外,由于细胞簇富含的基因与已知的标志物无法对应(可能是由于灵敏度和特异性低导致表达水平低),细胞类型注释尤为困难。
——Wang等人
*注:在开展这些研究时,Xenium细胞分割的默认参数是细胞核向外扩展15 µm。这一距离目前已经缩短至5 µm,而且Xenium现在还提供利用Xenium多模态细胞分割试剂盒进行形态学驱动的细胞分割选项。
强调这些结果的一致性
我们不仅要关注这两项独立研究的结果,还要强调结果的一致性。尽管在正交基准测试时使用了不同工具、不同组织类型和不同方法,但这两项研究发现:
Xenium表现出的特异性比CosMx更高(通过在靶基因相对阴性对照探针的百分比或FDR来衡量)。
Xenium在整个动态范围内与snRNA-seq和RNA-seq的相关性良好,而CosMx对低表达转录本的计数有夸大现象(两篇论文都明确强调了这一点)。
对于两个基因组合中共有的基因,Xenium检测到的转录本始终比CosMx更多。
Xenium能够检测到CosMx无法检测的细胞类型,包括淋巴细胞和罕见/难以检测的细胞类型。
此外,两个研究团队都公开了他们的数据,以便重新分析和纳入未来的比较研究。
比较研究的局限性
一项规划良好的研究会强调其实验设计中的局限性。例如:
Cook等人承认样本量有限,只包括一名患者的单个组织类型(不过作者后续正在开展一项更大规模的研究)。考虑到组织类型和复杂性对分析性能的潜在影响,这是一个重要的考虑因素。此外,这项研究(以及Wang等人的研究)重点关注FFPE组织,未在新鲜冷冻样本(空间分析常用的另一种样本保存方法)上评估分析性能。
Wang等人承认,组织切片和成像之间的时间不相同。尽管他们尝试对每种技术的时间点进行配对,但MERSCOPE组织的切片厚度比制造商建议的要薄,导致Xenium和MERSCOPE在配对时间点上的比较可能不相同(不过他们按照制造商建议的厚度运行了另一组样本)。我们在本篇文章中的大部分比较数据都集中在变化最小的样本上。他们还指出,为了让MERSCOPE基因组合与所有组织兼容,需要删除几个基因,这可能会影响其与Xenium的性能比较。
捕捉一幅更完整的图像:了解其他比较研究
本文深入探讨了两项近期发表的独立基准测试研究,它们采用了不同的方法,但得出了相似的结论。最近还开展了另外两项研究。
第一项独立的研究(3)由纽约基因组中心的Hartman和Satija领导。他们利用基于Baysor的细胞分割策略,在小鼠大脑样本上比较了6种不同的单细胞空间成像技术(包括Xenium和MERSCOPE)。作者指出了他们的研究有几个局限性,特别是他们只关注了单一组织类型(新鲜冷冻小鼠大脑)中的单一解剖特征(皮层)。这项工作使用了单细胞空间成像平台和scRNA-seq分析的公开数据集,而不是对同一小鼠或神经解剖位置进行对比。
第二项研究由NanoString赞助(4),由美国西北大学的Dulai团队开展。他们的研究比较了Xenium和CosMx在FFPE人类回肠和结肠活检样本中的性能。尽管网络研讨会没有谈到这项研究的局限性或注意事项,但还有几点值得考虑:
没有提供数据来衡量分析特异性和/或噪声(考虑到Cook等人和Wang等人在CosMx数据中观察到特异性总体偏低和‘上升曲线’,这是一个相关的考虑因素)。
尽管使用了公开的scRNA-seq数据集来注释细胞,但没有使用正交技术来提供‘真实数据’,以比较两个平台和单个样本之间的分析性能。
作者指出,CosMx在细胞类型比例方面的可变性比Xenium小,但没有提供每个平台检出细胞类型的准确性数据。
没有显示UMAP中细胞聚类或细胞类型注释的可视化结果,也没有显示空间分布情况,因此很难确定是检出的细胞类型聚集在一起还是噪声假象。
作者称,无论RNA质量如何,CosMx的表现都是一致的;不过文中只显示了16个样本中的5个样本,而且没有承认CosMx数据与质量较高的样本存在负相关
作者声称,CosMx检测到的每个细胞中的基因数是Xenium的6倍;这一说法并非由网络研讨会上的讲者提出,而是根据每个基因组合上的所有基因(而不是重叠基因)得出的,并且没有提及高于噪声的基因百分比。
随着多重分析、分析方法、细胞分割模态和软件等方面的进步,单细胞空间成像技术也在迅猛发展。这些研究代表了作者在开展研究时能接触到的技术,而了解文章发表后的技术进步可确保您选择最符合您需求的平台。
希望深入了解单细胞成像平台之间的比较?欢迎观看由David Cook和Luciano Martelotto博士主讲的网络研讨会!
如果您对我们的产品和技术感兴趣
欢迎扫码留下联系方式

参考资源:
1. https://www.biorxiv.org/content/10.1101/2023.12.13.571385v1
2. https://www.biorxiv.org/content/10.1101/2023.12.07.570603v2
3. https://www.biorxiv.org/content/10.1101/2024.01.11.575135v1
4. http://surl.li/ulsgv