
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Cas12核酸酶检测RNA新方法:TracrRNA重编程 让靶向DNA的Cas12核酸酶直接检测与pam无关的RNA
【字体: 大 中 小 】 时间:2024年07月23日 来源:news-medical
编辑推荐:
CRISPR-Cas系统,细菌中的防御系统,已经成为分子诊断技术的丰富来源。
细菌有特殊的防御机制来保护自己免受病毒的侵害,CRISPR-Cas系统就是其中之一。研究领域的各路高手纷纷大力于开发源于不同细菌中的各种CRISPR-Cas系统,已经成为分子诊断技术的丰富资源。位于德国维尔茨堡的亥姆霍兹RNA感染研究所(HIRI)的研究人员又进一步开发了一种新方法来扩展这个CRISPR-Cas工具箱。他们的新方法,——简称为PUMA( (Programmable tracrRNAs Unlock protospacer-adjacent Motif-independent detection of ribonucleic Acids by Cas12 nucleases),可以用Cas12核酸酶检测RNA,并且被检测靶标无需PAM,有望在一众基于CRISPR的检测新方法中脱颖而出——可是 Cas12核酸酶天然靶向DNA,PUMA是如何可实现高精确度检测RNA的?研究小组在《Nature Communications》杂志上发表文章介绍了他们的研究结果。
背景
除了应用于基因组编辑,CRISPR在诊断领域的应用同样获得高度关注,CRISPR检测方法因为相比PCR检测方法更快速、便宜且便携,越来越受重视。检测领域用得较多的有CRISPR相关(Cas)核酸酶Cas12a。基于Cas12核酸酶的dsDNA检测技术已经成为从疾病诊断到食品质量控制和环境污染物检测等众多应用的主要焦点,在已经发现的十几个亚型的Cas12同源物中不少被开发用于核酸检测。
尽管靶向dsDNA的Cas12核酸酶在核酸检测中越来越受重视,但它们通常有两个明显的限制:严格要求侧翼原间隔邻近基序(PAM)和对dsDNA靶标的限制。这些限制阻碍了在检测领域的广泛应用。突破这些限制就意味着更广泛的应用前景和”钱景“。
研究亮点
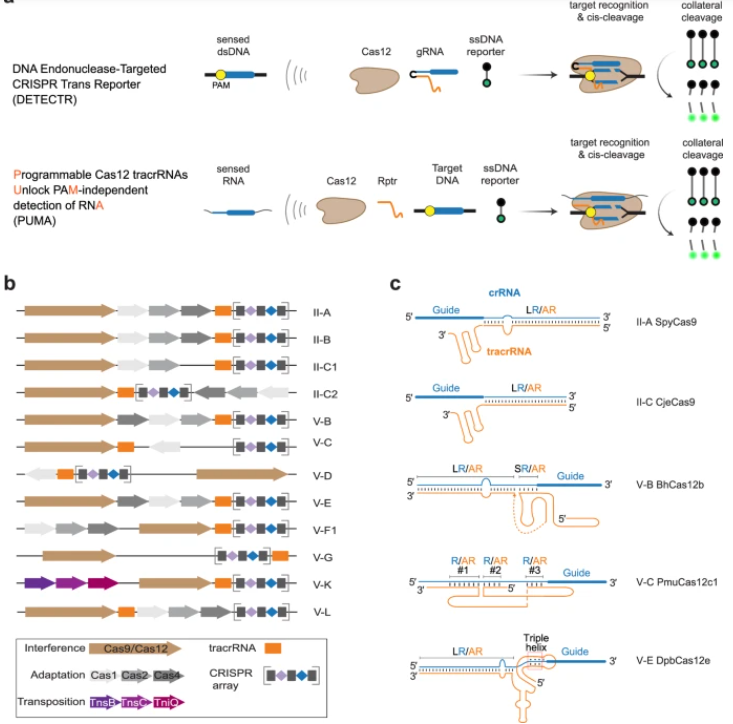
在CRISPR-Cas系统中,CRISPR核糖核酸(crRNA)可以识别外来基因组的区域——例如病毒DNA,作为“向导RNA”指引CRISPR相关核酸酶(Cas)像剪刀一样将特定外来基因切割成无害的片段。许多CRISPR-Cas免疫系统——如在II型CRISPR-Cas系统(例如Cas9)和大多数V型CRISPR-Cas系统(例如Cas12)——使用反式激活CRISPR RNA (TracrRNAs)产生向导(g)RNA:tracrRNA杂交到crRNAs中的保守重复区域,形成的RNA双链、通常由核糖核酸内切酶RNase III进行加工并与同源的Cas核酸酶结合形成复合物,结合的RNA双链作为gRNA,指导核酸酶结合和切割目标dsDNA。
Beisel实验室与JMU于2021年合作开发的Cas9诊断平台LEOPARD时就利用重编程tracrRNA,与感兴趣的靶标RNA杂交并将其转化为gRNA,指导Cas9靶向序列特异性DNA,将检测RNA的存在与Cas9介导的双链DNA切割关联起来。在新研究中,Chase Beisel领导的团队将LEOPARD的独特功能扩展到了Cas12—— Cas12相比Cas9更不容易脱靶,Cas12还可以通过独特的反式切割活性来增加输出信号,可以实现比Cas9更灵敏的RNA检测;但这种反式切割活性使得Cas12不像Cas9那样可以进行多路检测。研究人员将这种方法命名为PUMA。
重新编程来自不同Cas12核酸酶的TracrRNA,将感兴趣的RNA的存在、与dsDNA切割和随后的侧链单链DNA切割联系起来——重点是:所有这些RNA都不需要原间隔邻近基序(PAM,protospacer-adjacent motif )。这是通过将PAM设计到被切割的dsDNA 上而实现的。
作者团队对不同V型CRISPR-Cas系统相关的tracrRNAs的重编程,解决了基于Cas12的检测技术的两个挑战:直接RNA检测和PAM要求。这个方法通过结合并将待检测RNA转化为靶向dsDNA的gRNA,让靶向dsDNA的Cas12核酸酶能够实现直接检测RNA——被检测的底物是RNA,相应的dsDNA靶标可以由用户提供,因此用户可以完全控制dsDNA靶标的序列、长度和化学性质。PAM被编码到dsDNA靶标中,绕过了对待测RNA对PAM的要求。几乎任何RNA序列都可以用PUMA检测,只要RNA序列遵循阐明的设计规则,并且满足包含抗重复和向导的最小长度。他们还发现缩短的dsDNA靶标可以提高反切活性,提高检测速度。
然后,作者使用该平台检测了来自五种不同细菌病原体的16个S rRNA序列。这些发现用TracrRNA重编程扩展了靶向dsDNA的Cas12核酸酶检测,提高了基于CRISPR的RNA检测的灵活性和多功能性。
克服障碍
Helmholtz RNA感染研究所(HIRI) RNA合成生物学系主任Chase Beisel说:“CRISPR,通常被称为‘基因剪刀’,是许多分子技术的基础,”“使用PUMA,我们可以重新编程tracrRNA。这使我们能够决定哪个RNA生物标记物成为向导RNA。“ 该研究的第一作者Chunlei Jiao解释说:“这种向导RNA反过来将Cas12引导到我们提供的DNA分子上,并激活基因剪刀。”Chunlei Jiao是实验室的前研究生和博士后研究员,也参与了LEOPARD的开发,现在新加坡国立大学担任教授。Beisel补充说:“DNA切割告诉我们样品中存在哪种生物标志物,例如针对不同病原体的生物标志物。”因此,这种新方法能够使用原本只能识别DNA的CRISPR核酸酶检测RNA生物标志物。“这对于只能在RNA水平上发现的分子生物标志物尤其重要。例如,这包括RNA病毒,”PUMA不需要特定的识别序列:PAM包含在提供的DNA靶分子中,显著提高该方法的速度。
“PUMA有潜力成为一种灵活而精确的RNA检测工具,”该团队通过鉴定与急性败血症相关的五种细菌病原体,证明了该方法的潜力。他们的检测依赖于一个单一的通用的、重编程的tracrRNA,它提供了一种区分不同类型细菌的简化方法。这在医学上开辟了广泛的潜在应用。Jiao说:“这项新技术代表了一种新的CRISPR诊断形式,可以在护理点进行可靠的分子检测,无论是鉴定病毒或细菌病原体,还是检测癌症生物标志物。”
研究小组已经在计划下一步:“我们的目标是实现与LEOPARD类似的多路读出,并扩大该技术的应用范围,”Beisel说,他还预计该技术将在研究界得到广泛应用:“我们希望我们的研究将促进对tracrRNA重编程的进一步探索。”
 生物通微信公众号
生物通微信公众号
知名企业招聘