
-

生物通官微
陪你抓住生命科技
跳动的脉搏
苏州医工所高欣团队两篇论文入选MICCAI 2024
【字体: 大 中 小 】 时间:2024年07月04日 来源:中国科学院苏州生物医学工程技术研究所
编辑推荐:
图 1. DeSAM的架构示意图 图 2. DeSAM在前列腺和腹部多器官的公开数据集上的分割结果 图 3. MBA-Net架构的展示图 以上成果分别以“ DeSAM: Decoupled Segment Anything Model for Generalizable Medical Image Segmentation”和“ MBA-Net: SAM-driven Bidirectional Aggregation Network for Ovarian Tumor Segmentation”发表于 MICCAI 2024上
MICCAI (International Conference on Medical Image Computing and Computer Assisted Intervention) 是医学图像分析领域国际公认最具影响力的学术会议,代表了医学影像人工智能方法创新的前沿水平。近日,MICCAI 2024 论文接收结果公布,苏州医工所高欣研究员团队两篇论文被录用,其中一篇为提前录用(前11%)。
近年来,深度学习在医学图像分割领域取得了显著成果,但常面临域偏移问题,即在源域训练的模型难以泛化到未见过的目标域,比如用一家医疗中心的数据构建的模型在本中心数据上性能良好,但在另一家医疗中心的数据上表现欠佳。这一问题严重限制了深度学习模型在临床实践中的应用。基于大规模数据集预训练的视觉基础模型,如Segment Anything Model (SAM),虽然展现出强大的泛化能力,但这种能力主要体现在交互式分割任务中。当应用于医学图像的全自动分割场景时,由于缺乏特定领域的专业知识和精确的提示信息,其性能往往显著下降。
针对这一局限性,苏州医工所高欣团队提出了一种名为DeSAM (Decoupled Segment Anything Model)的新型网络架构,用于医学图像分割的域泛化(图1)。DeSAM通过解耦SAM的掩码解码器,引入了提示相关分数模块和提示解耦掩码模块,能够利用基础模型的知识,同时最大程度避免了由错误提示引起的性能下降。在公开的跨站点前列腺和跨模态腹部图像分割数据集上的实验结果表明,DeSAM达到了已报道的最佳性能(图2)。
此外,团队针对卵巢肿瘤分割中模型泛化性较差的挑战,还提出了一种新型的基础模型驱动的双向聚合网络MBA-Net。该网络可从基础模型中继承强大的分割能力,并通过双向特征聚合机制与针对卵巢肿瘤领域设计的分支相结合(图3)。在多模态卵巢肿瘤超声数据集上,该网络的平均Dice指标达到90.75%,在畸胎瘤和高级别浆液性癌分割上,相比现有最先进方法,指标分别提升了2.75%和4.53%。在多中心卵巢肿瘤MRI数据集上,MBA-Net的平均Dice指标达到89.04%,相比其他方法有明显优势。
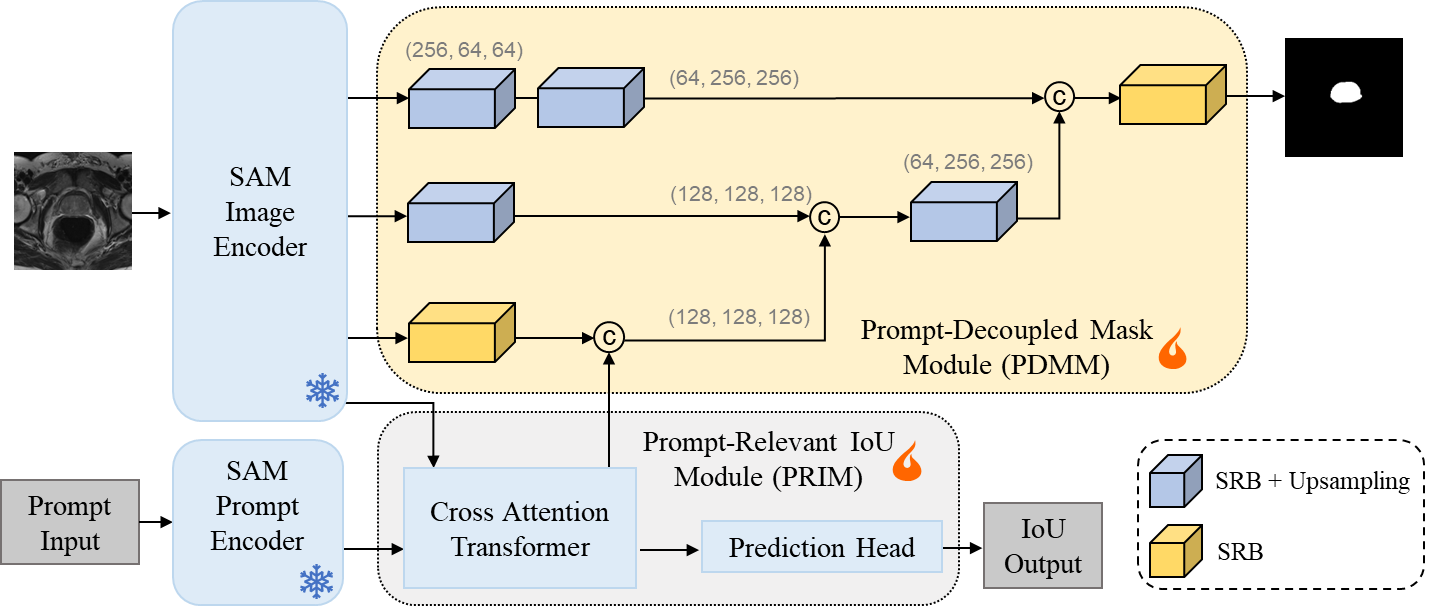
图1. DeSAM的架构示意图

图2. DeSAM在前列腺和腹部多器官的公开数据集上的分割结果

图3. MBA-Net架构的展示图
以上成果分别以“DeSAM: Decoupled Segment Anything Model for Generalizable Medical Image Segmentation”和“MBA-Net: SAM-driven Bidirectional Aggregation Network for Ovarian Tumor Segmentation”发表于MICCAI 2024上。论文的第一作者为苏州医工所博士研究生高亦凡,通讯作者为高欣研究员。该研究得到国家自然科学基金的支持。