
-

生物通官微
陪你抓住生命科技
跳动的脉搏
跨物种单细胞组学数据分析的新工具CACIMAR
【字体: 大 中 小 】 时间:2024年07月03日 来源:中国科学院广州生物医药与健康研究院
编辑推荐:
相关成果以“CACIMAR: cross-species analysis of cell identities, markers, regulations, and interactions using single-cell RNA sequencing data”为题目发表在学术期刊Briefings in Bioinformatics上
近日,中国科学院广州生物医药与健康研究院王杰课题组开发了新的计算工具CACIMAR(Cross-species Analysis of Cell Identities, Markers, Regulations),用于分析跨物种单细胞转录组测序数据(scRNA-seq),揭示物种间细胞类型、标志基因、细胞内调控以及细胞间相互作用的进化保守性。相关成果以“CACIMAR: cross-species analysis of cell identities, markers, regulations, and interactions using single-cell RNA sequencing data”为题目发表在学术期刊Briefings in Bioinformatics上。
物种不仅在基因层面,也在细胞类型、基因表达、基因调控、细胞间相互作用等方面显示出进化保守性。单细胞转录组测序技术已广泛用于不同物种的单细胞表达研究,为在细胞类型水平研究物种的进化保守性提供了新机会。已有的算法能通过分析跨物种单细胞转录组数据识别进化保守的细胞类型,如Seurat和SAMap等。但是,这些方法主要通过跨物种的单细胞聚类确定保守细胞类型,其准确性在很大程度上取决于跨物种批次效应校正的能力。这些软件对进化距离较远物种进行聚类分析,仍存在明显不足。此外,目前仍没有方法能确定细胞间相互作用的进化保守性。
针对以上问题,我院王杰课题组开发了新的R软件包 CACIMAR ,用于分析跨物种的单细胞转录组数据。CACIMAR的保守性分析主要包含三个步骤(图1)。第一步,CACIMAR 基于每个物种内的单细胞聚类结果确定每个物种的细胞类型、标志基因、细胞内调控和细胞间相互作用。该策略不用进行跨物种的单细胞聚类,有效避免了跨物种的批次效应。第二步,基于标志基因的统计值(statistical power)和物种同源性(homolog),CACIMAR计算细胞类型的保守性分值。此外,CACIMAR还建立了特征加权和的模型(weighted sum?model),分别计算细胞内调控网络的保守性分值和细胞间相互作用的保守性分值。最后一步,基于计算的不同保守性分值分别确定跨物种保守或特异的细胞类型、细胞内调控和细胞间相互作用。该研究将CACIMAR应用于小鼠、斑马鱼和小鸡的视网膜损伤再生的公共单细胞转录组数据中,有效地确定了小鼠、斑马鱼和小鸡视网膜中保守的细胞类型、标志基因、细胞内调控和细胞间相互作用。
总体上,CACIMAR提供了一种新的算法,能克服现有跨物种单细胞转录组数据分析的挑战,特别是在识别跨物种保守和特异的细胞类型、基因调控以及细胞间相互作用方面。通过该算法,研究人员能深入地分析不同物种的细胞和分子水平的进化保守性,为理解物种进化的机制提供了新的视角。
广州健康院的王杰研究员和徐雪丽助理研究员为本论文的共同通讯作者,蒋俊尧和李金连为论文的共同第一作者。该研究得到了国家自然科学基金、国家重点研发计划、广东省自然科学基金等项目的资助。
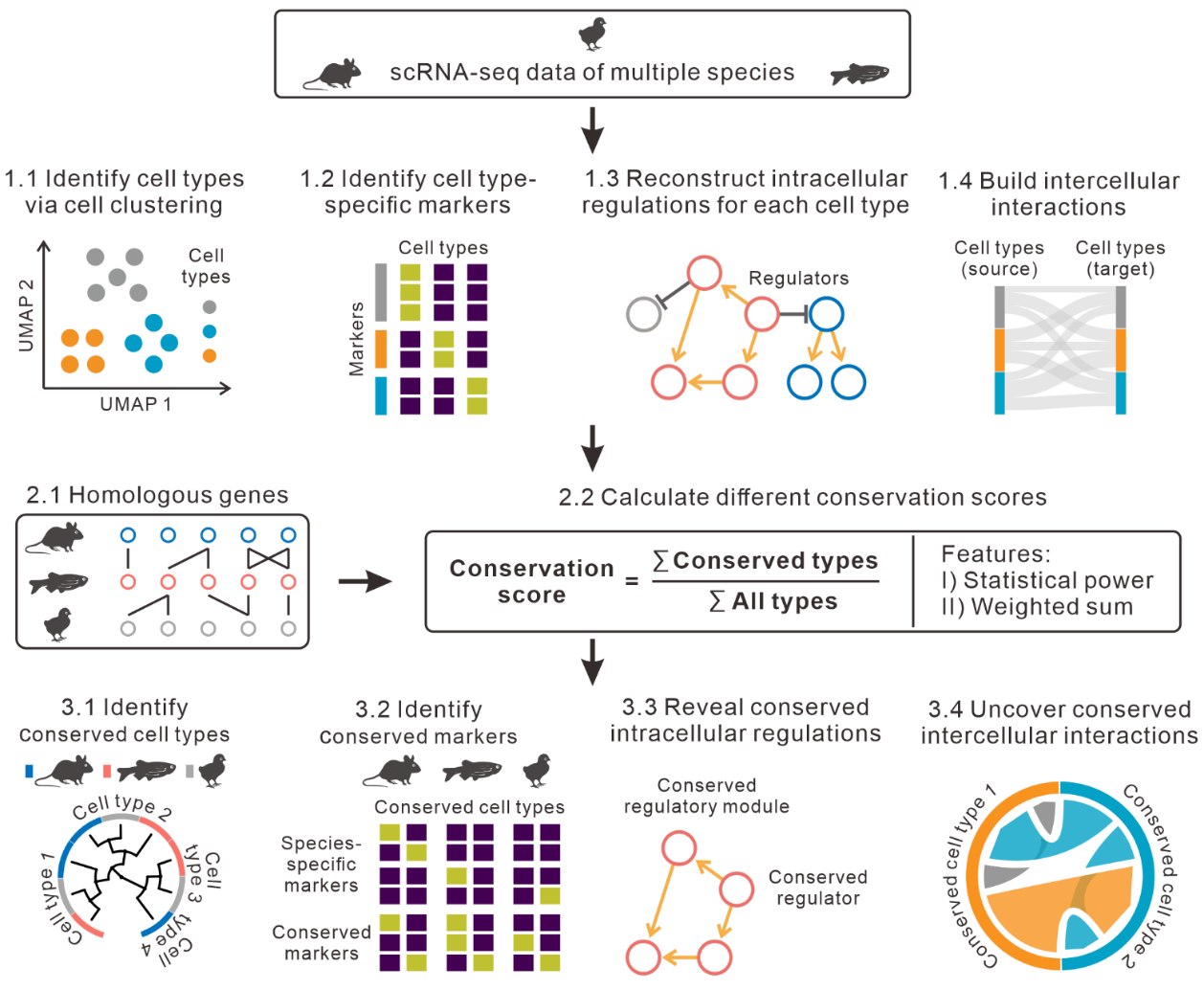
图1?CACIMAR进行跨物种单细胞转录组数据分析示意图