
-

生物通官微
陪你抓住生命科技
跳动的脉搏
PacBio HiFi测序揭开来自贝加尔湖新多样性菌群的神秘面纱
【字体: 大 中 小 】 时间:2024年06月07日 来源:基因有限公司
编辑推荐:
作者首次应用PacBio CCS 宏基因组学方法来深入分析了生活在贝加尔湖1600米深水体中的 Ca. Patescibacteria。
背景介绍
候选门级辐射类群(candidate phyla radiation,CPR)是一类在自然界广泛分布的庞大细菌类群,成员众多,基于宏基因组测序结果已划分出75个门水平分支。CPR细菌是研究微生物进化与各类生境微生物协同作用的理想材料与系统,其在系统进化树中位于细菌域的最外侧,与古菌域外侧的DPANN超门毗邻,进化地位特殊。该类群具有独特的生物学特性,如细胞个体小,基因组精简,通常需要与宿主共生、纯培养困难,目前仅在人类口腔中成功分离到一株CPR细菌(TM7菌株UB2523)。
贝加尔湖是世界上最深的淡水湖,在其最深处含有大量的Candidatus Patescibacteria (formerly CPR),然而,先前获得的CPR宏基因组组装结果非常不理想。在今天要介绍的《A novel and diverse group of CandidatusPatescibacteria from bathypelagic Lake Baikal revealed through long read metagenomic》一文中,作者首次应用PacBio CCS 宏基因组学方法来深入分析了生活在贝加尔湖1600米深水体中的 Ca. Patescibacteria。

PacBio的研究意义
在这项工作中,作者在前期利用短读长宏基因组测序已经进行过深入研究的环境中,发现了Ca. Patescibacteria的一个新的微生物类群,它们的丰富性和巨大的基因组多样性在以前的研究中很可能被忽略了,因为来自 Illumina的宏基因组组装结果已被证明有偏好性。而PacBio HiFi测序凭借无偏好性的特点,应该更多的用于发现原核生物的多样性并产生更可靠和更丰富的MAG(metagenome-assembled genomes)信息。

部分数据展示
贝加尔湖PacBio宏基因组研究
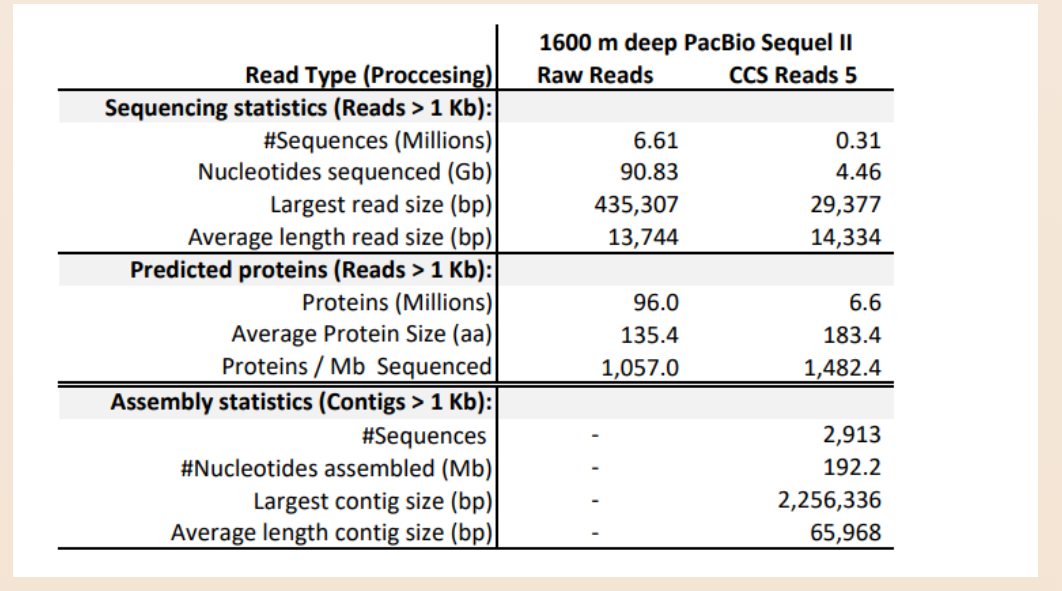
作者使用 PacBio CCS 对夏季收集的深水样本(1600米)进行长读长宏基因组(LAGs)测序,以补充以前在夏季和冬季在1250和1350米深的贝加尔湖收集的Illumina数据集。总共产生90Gb的原始数据,平均reads大小为14.3 Kb(表1) 。与之前的Illumina数据相比,有些微生物群体的比例发生较大的变化。放线菌的比例较高(51.7%)(几乎是在海拔1350米处检测到的比例的两倍) ,其次是Ca. Patescibacteria (15.2%) ,其丰度是1250-1350米处 Illumina 16S rRNA基因丰度的三倍。除此之外,在PacBio 1600m 宏基因组中,Crenarcheota (泉古菌门 1.1%)和 Nitrospirota (硝化螺旋菌门 0.21%)数量较少,比例较低(约10-20倍)。以上观察到的这些微生物群落差异可能是由于样品深度(1350/1600米)和16S rRNA测序读长(短读长约100bp 和PacBio测序的1000bp)的差异引起的。
表1. 贝加尔湖1600米水域长读长测序和宏基因组组装的数据概要统计
Ca. Patescibacteria 16S rRNA基因研究
为了更详细地对Ca. Patescibacteria分类,作者对以前组装的所有16S rRNA序列进行提取和分类,包括来自公开可用的MAG和PacBio CCS 组装的基因组(LAGs)序列,同时也将不需要任何组装的PacBio CCS 的16S rRNA reads涵盖其中。由此产生的系统发育树(图1)显示出一条很深的分支,包含了大约一半的Ca. Patescibacteria 16S rRNA CCS reads(278个中的149个),这条分支没有任何已知的进化枝 ,这表明来自1600米深水域的Ca. Patescibacteria内存在一个丰富的新类群,作者将其命名为Baikalibacteria。
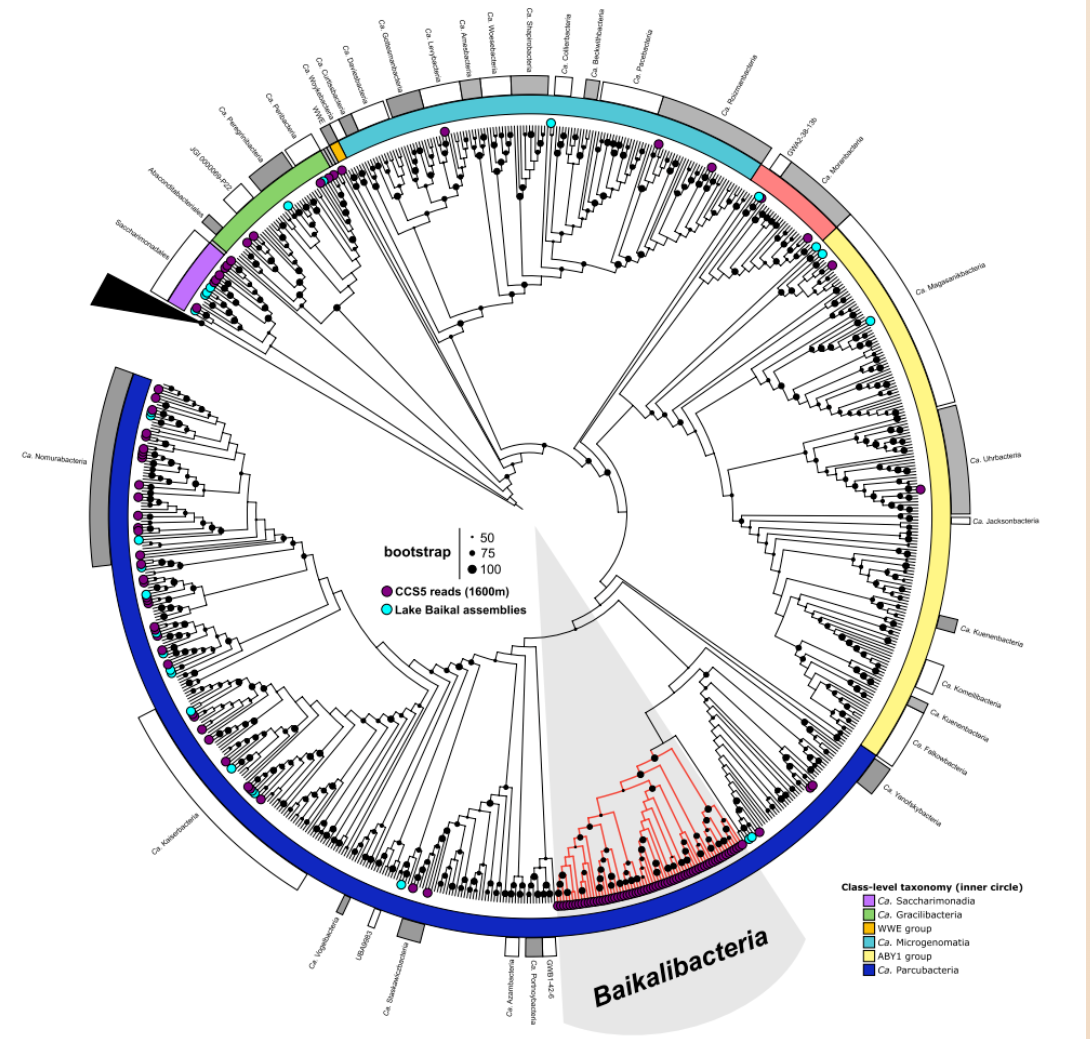
图1. Ca. Patescibacteria系统发育树。来自贝加尔湖1250米、1350米(Illumina)和1600米(PacBio CCS)的MAGs呈蓝色。1600米样本的PacBio CCS 16S RNA 序列呈紫色。
Baikalibacteria 泛基因组分箱
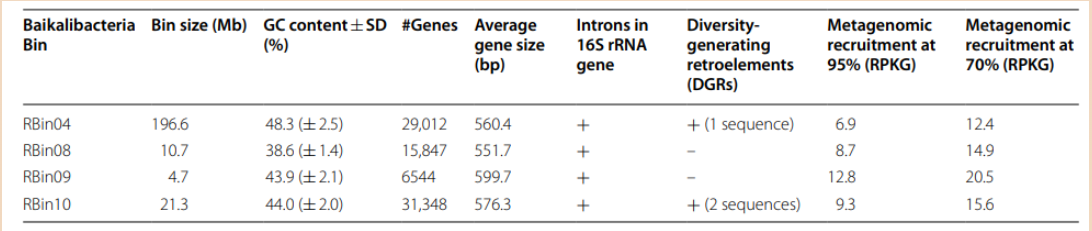
为了提高对Baikalibacteria这个新微生物群体的认知,作者将含有Baikalibacteria 16S rRNA的reads作为seeds,将所有被归类为Ca. Patescibacteria的reads分箱在一起,从而能够得到具有一致性的基因组参数(GC含量和四碱基频率)的独立的分箱(bins)数据。最终含有Baikal 16S rRNA 基因的contigs分成四个read bins(即 RBin04,RBin08,RBin09和 RBin10),它们都含有其所有16S rRNA 序列的近70% 。因此,作者集中在这四个bins上来推断他们的泛基因组的一些生态特性。
表2. Baikalibacteria bins的特性
Baikalibacteria泛基因组分箱代谢预测
对Baikalibacteria泛基因组的代谢预测与先前发表的用Ca. Patescibacteria 的MAGs进行推断得到的结论相似,它们缺乏构建核苷酸、氨基酸、脂质和维生素的大部分基本生物合成反应(图2) ,使得它们高度依赖于外部资源。它们还共同利用葡萄糖通过糖酵解 (EMP)途径获得能量,尽管它们存在于含氧的水中,但它们都缺乏三羧酸循环(TCA)氧化途径和电子传递链复合物I - IV ,因此,它们可能依赖于丙酮酸与乳酸、乙酸和 3-羟基-2-丁酮的混合酸发酵反应来再生NAD+。

图2. 四个Baikalibacteria分箱的代谢预测(KEGG)。刻度(颜色和大小)表示检测到的完整通路所需的基因的比例
结论
通过PacBio长读长测序对16S rRNA基因的全覆盖,加上使用长读长分箱来揭示“隐藏”的原核生物群的高度多样性,是生态基因组微生物学向前发展的关键策略。
基因有限公司作为PacBio公司中国区合作伙伴,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。



