
-

生物通官微
陪你抓住生命科技
跳动的脉搏
吕绍武教授研究团队在探索DNA重组机制的前沿研究中取得重要进展
【字体: 大 中 小 】 时间:2024年06月22日 来源:吉林大学生命科学学院
编辑推荐:
研究成果于2024年1月30号在学术期刊《International Journal of Biological Macromolecules》正式发表(Comparative analysis of structural dynamics and allosteric mechanisms of RecA/Rad51 family proteins: Integrated atomistic MD simulation and network-based analysis)
吕绍武教授、杨朔副教授科研团队深入解析了RecA/Rad51重组酶家族的结构动态和功能调控机制,为理解这些关键蛋白在同源重组中的作用提供了新的视角。研究成果于2024年1月30号在学术期刊《International Journal of Biological Macromolecules》正式发表(Comparative analysis of structural dynamics and allosteric mechanisms of RecA/Rad51 family proteins: Integrated atomistic MD simulation and network-based analysis)。论文第一完成单位为吉林大学生命科学学院,吉林大学生命科学学院潘跃博士为该论文的第一作者,吉林大学吕绍武教授和杨朔副教授为该论文的共同通讯作者。

同源重组在双链断裂修复、停滞复制叉修复和减数分裂中起关键作用。RecA/Rad51家族重组酶催化同源重组过程中发生的DNA链入侵反应。此家族蛋白之间的序列差异较大,但它们通常能够折叠成高度相似的三维结构,并发挥着保守的同源重组活性。然而此家族蛋白动力学机制的分子细节,特别是在残基水平上的机制仍然不清楚,其生物学意义背后的分子水平动态机制尚未得到充分表征。吕绍武教授和杨朔副教授课题组选取了涵盖主要生物界(原核生物、真核生物、古细菌和病毒)的5种具有代表性的RecA/Rad51重组酶家族成员,采用全原子分子动力学模拟、微扰响应扫描和蛋白质结构网络分析等多种技术,系统性地探索了各重组酶突触前丝保守的生物学意义,低序列一致性背后的分子结构和动态行为(图1)。
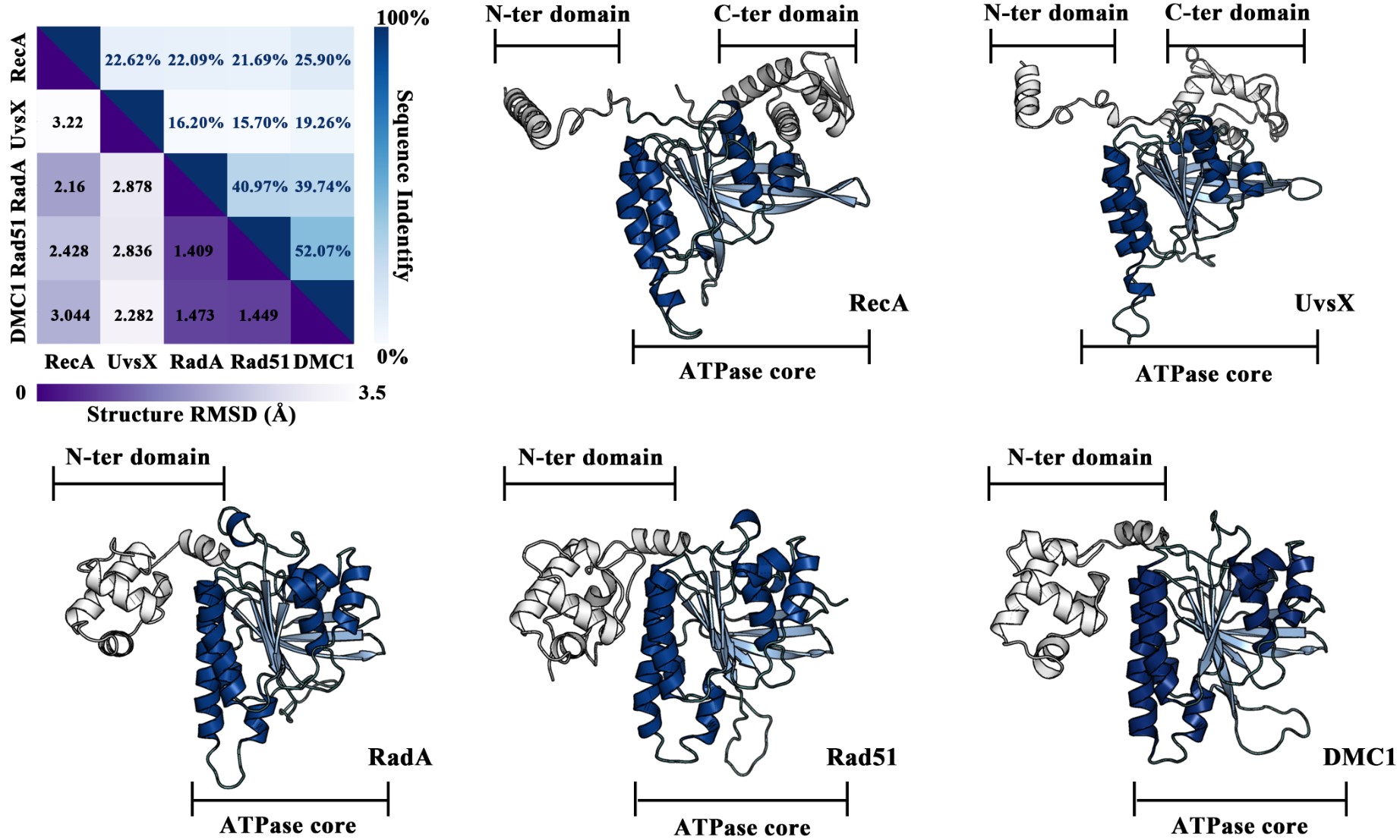
图1:五种重组酶的序列与结构对比
研究结果揭示了各重组酶蛋白结构区域间保守的协同运动、敏感性和效应性残基分布以及枢纽残基分布等(图2)。动态差异性主要表现在ATP-DNA结合域的通讯路径上,即RecA与UvsX之间的通讯路径密集,而RadA、Rad51和DMC1的通讯路径稀疏(图3)。这可能是因为ATP的结合显著增加了RecA和UvsX对ssDNA的亲和力,而对RadA、Rad51、DMC1的影响较小。课题组后续对UvsX蛋白进行了更深入研究,团队构建了UvsX结合双链DNA的二聚体模型,并在298 K和310 K温度下进行了全原子分子动力学模拟,并以RecA蛋白作为参考。结果表明:在310 K时,RecA和UvsX的单体之间动态互相关性增强,蛋白质更容易达到稳定构象,ATP结合能力也更强。RecA在310 K时与dsDNA的结合自由能剧烈波动,而UvsX在同温度下与dsDNA结合良好。本文的研究结果为RecA/Rad51家族蛋白的功能保守性和独特性提供了分子水平的理解,并为进一步探索RecA和UvsX在dsDNA结合状态下的结构特征和结合特性提供了理论基础。这些发现不仅为探究DNA重组酶的作用机制提供了必要基础,还为蛋白生物工程应用和药物开发提供了新思路。
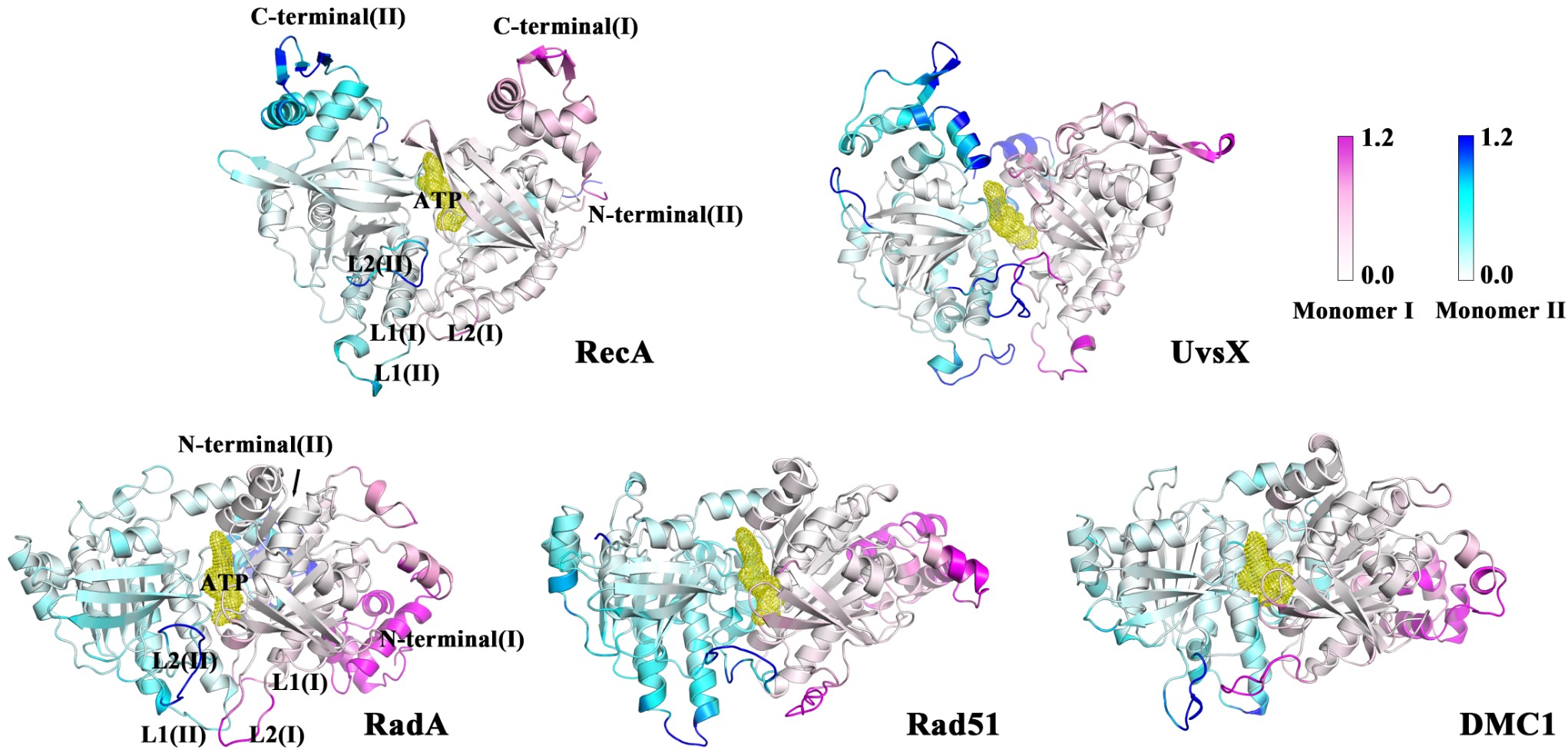
图2:蛋白骨架柔韧性可视化图。
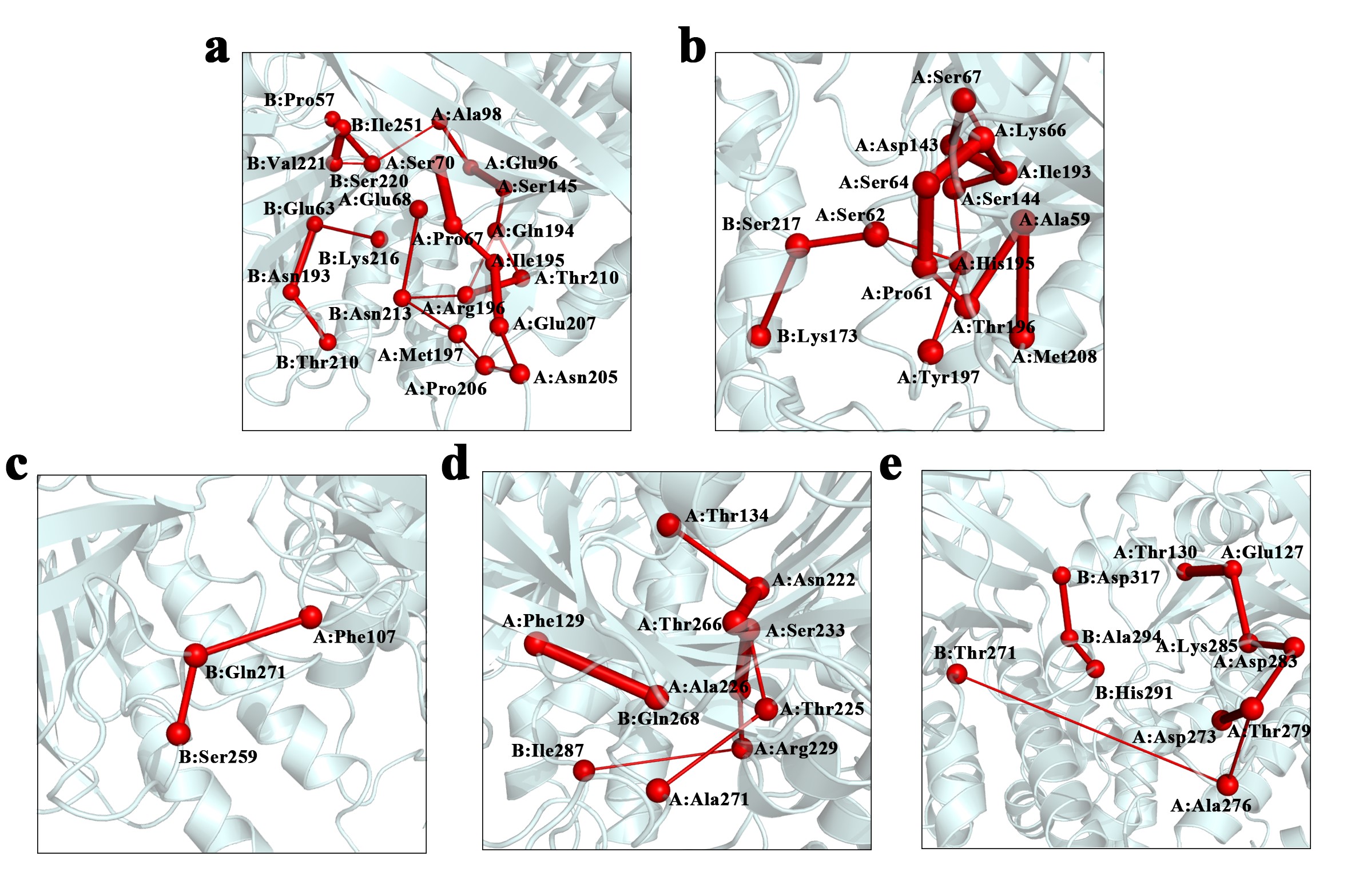
图3:ATP结合位点与DNA结合位点之间的通讯路径。
a. RecA, b.UvsX, c.RadA, d. Rad51, e. DMC1。
该研究工作得到了国家重点研发计划项目(2022YFF0904000)和吉林大学学科交叉创新项目(JLUXKJC2021ZZ01)的资助。
全文链接:https://doi.org/10.1016/j.ijbiomac.2024.129843