
-

生物通官微
陪你抓住生命科技
跳动的脉搏
热点论文!Nature Methods指出了高通量测序中的一个重要错误源头
最新研究提高了高通量测序中分子定量的准确性
【字体: 大 中 小 】 时间:2024年04月30日 来源:生物通
编辑推荐:
牛津大学纳菲尔德骨科、风湿病学和肌肉骨骼科学系(NDORMS)的一个研究小组开发了一种新方法,可以显著提高RNA测序的准确性。他们指出短读和长读RNA测序中不准确定量的主要来源,并引入了“majority vote”纠错的概念,从而大大提高了RNA分子计数的准确性。

牛津大学纳菲尔德骨科、风湿病学和肌肉骨骼科学系(NDORMS)的一个研究小组开发了一种新方法,可以显著提高RNA测序的准确性。他们指出短读和长读RNA测序中不准确定量的主要来源,并引入了“majority vote”纠错的概念,从而大大提高了RNA分子计数的准确性。
研究重点:
过往科学家没有重视的PCR循环是很多精准测序当中的一个重要错误源头;
新研究发明了一种新型的同源三聚体的分子生物标记法来达到精准测序的目的。研究人员报告了再批量测序和单细胞测序下的99%和98%的测序精度。这称之为几乎逼近绝对精准定量的测序水平。这是该领域目前最为精准的纠错测序技术。
短读和长读RNA测序目前主要存在的问题
遗传物质的准确测序在现代生物学中是至关重要的,特别是对于理解和解决与遗传异常有关的疾病方面。然而,目前的方法遇到了很大的限制。
论文一作Jianfeng Sun博士解释道:“短读长测序技术在常规RNA测序中的精度是很高的,然而其在单细胞RNA测序中的精度根据不同的测序条件设置忽高忽低。比如,将PCR体外扩增次数增高后再去测序的精度其实并不高。长读长RNA测序目前在单细胞和常规RNA测序中的精度均比短读长要低一些。因为测序平台在不断推陈出新,所以目前在一般情况下开展的测序精度不会出现过低的情况。
但是,无论短读长还是长读长测序中只要出现一定量的错误,那么这可能就会潜在地影响数据分析的质量,从而可能潜在地影响生物研究结论。
所以最主要的问题还是精度问题。
另外,短读长测序的价格要比长读长低很多。但是长读长的价格普遍还是居高不下。如何在测序精度和价格之间寻找平衡是其中一个重要的议题。”
创新新方法
这项具有里程碑意义的研究由牛津大学计算生物学副教授Adam Cribbs和Jianfeng Sun领导完成,他们开发了一种创新的方法,用于纠正高通量测序中广泛出现的PCR扩增错误。
研究发表在《自然方法》(Nature Methods)杂志上,指出PCR人工产物是定量不准确的主要原因,这解决长期以来在生成准确的RNA分子绝对计数方面所面临的挑战,这对基因组学研究的各种应用至关重要。
在这篇文章中,研究人员重点研究了特异性分子标记(Unique Molecular Identifiers, UMIs,生物通注),这是一种随机的寡核苷酸序列,用于消除PCR扩增过程中引入的偏差。虽然UMIs已被广泛应用于测序方法,但该研究表明,PCR错误可能会破坏分子定量的准确性,特别是在不同的测序平台上。
Sun说:“PCR扩增对于大多数RNA测序技术来说都是必不可少的,但它可能会引入误差,损害数据的完整性。我们通过使用同源三聚体核苷酸块合成UMI条形码来解决这个问题,增强了纠错能力,实现了近乎绝对的RNA分子定量,显著提高了分子计数的准确性。”
“测序一般需要使用聚合酶链式反应(PCR)对分子序列进行扩增。PCR扩增后的分子进入测序池子后会影响正确的分子计数。所以待测分子需要用一些分子序列条形码对其进行身份标记,在PCR扩增后进行剔除。但是PCR这个过程会引入错误,称为PCR错误。如果条形码也错了,PCR扩增的分子的正确识别可能就会遇到困难,所以可能会破坏分子定量的准确性。在不同的测序平台上,PCR错误的影响是很不同的。例如,在短读测序平台,错误率并不高。但是基于电信号识别碱基从而测序的牛津纳米孔测序会相对高。”
“majority vote”方法
同源三聚体是由三个相同碱基组成的核苷酸序列,如AAA、CCC、GGG。通过评估同源三聚体核苷酸相似性,研究人员可以通过“majority vote”方法检测和纠正错误(图1)。
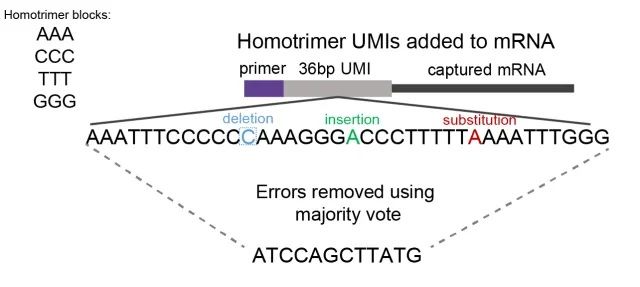
图1:显示同源三聚体UMI majority vote错误纠正的示意图。我们用同源三聚体核苷酸块(由AAA、CCC、GGG、TTT组成的组合)构建了UMIs。通过评估三聚体核苷酸的相似性,通过“majority vote”系统识别和纠正删除、插入或替代的错误,选择最常见的核苷酸。
“‘majority vote’纠错这一概念具体是指使用多数投票法对同源三聚体中的错误测序的碱基进行纠错。比如,同源三聚体AAA在测序后变成了AGA,那么就可以使用多数投票法将其投票为A。不同的同源三聚体均可以按照这样的方式进行一一纠错,最后形成一条连续的序列,” Sun补充说。
该研究表明,在分析差异表达基因和转录本(DEGs和DETs)时,同源三聚体UMIs在减少假阳性折叠富集方面明显优于传统单体UMIs。这种增强对于DEGs或DETs的准确识别和定量至关重要,特别是在批量测序方法中。此外,在单细胞测序中,通常需要广泛的PCR扩增,同源三聚体UMIs已被证明可以有效减轻PCR人工产物的影响,从而大大提高测序数据的可靠性。
“通过构建同源核苷块的UMIs,我们的目标是提高短读和长读测序的纠错能力,这是我们对提高测序技术应用的承诺,”Cribbs说。
意义深远
这项研究具有深远的意义。通过纠正UMIs中的PCR误差,极大地提高了各种测序应用中的分子定量准确性。它是大量RNA、单细胞RNA和DNA测序研究人员的重要工具,可以实现准确的基因表达和分子谱分析。增强的UMI纠错不仅减少了假阳性的发生率,而且还提供了多种诊断应用,特别是在需要对样本进行纵向分析的情况下。
Sun解释说:“UMI纠错是PCR纠错的其中一种方式。如果UMI纠错情况得到改善,那么PCR错误的分子计数就会变好。这样PCR扩展的分子被错误归入原始待测分子的可能性就低,所以假阳性就低。从而,分子表达量测准了之后就会帮助后续的疾病诊断(判断表达量是否异常等),并且可能会增加更多的诊断应用的可能性(例如,疾病诊断中的假阴性问题,使用测序错误较少精度高的表达数据做鉴定会帮助排除出现假阴性的鉴定结果,可靠度高应用存在的可能性就越高)。在纵向研究中,不同的样本或是实验重复之间存在的差异有可能很大。普通的纠错方法在底/高错误率的情况下鲁棒性可能差异比较大。然而我们在不同的样本或是实验重复中得到的鲁棒性是比较强的,也证明了该方法在应用阶段的稳定性。”
这篇论文目前是《Nature Methods》有数据追踪以来与同期发表文章相比最受欢迎的文章,排名第1,而且在所有期刊当中发表的同期可追踪的202,746篇文章中网络热度位居1962名(详情请看https://www.nature.com/articles/s41592-024-02168-y/metrics)。
同时该文章也吸引了各媒体的报道,牛津大学也详细报道了这项研究:
https://www.ox.ac.uk/news/2024-02-08-new-research-improves-accuracy-molecular-quantification-high-throughput-sequencin
原文标题:
Correcting PCR amplification errors in unique molecular identifiers to generate accurate numbers of sequencing molecules
作者简介:
Jianfeng Sun
POSTDOCTORAL RESEARCH ASSOCIATE IN SINGLE-CELL SEQUENCING ANALYSIS
I obtained my Ph.D. (Nov. 2017 - Feb. 2021) in deep learning-based structural biology from the Technical University of Munich, Germany. Since Jul. 2021, I have been a postdoctoral researcher in Prof. Cribbs' lab in NDORMS at the University of Oxford. My main responsibility is the improvement of long-read sequencing accuracy with computational approaches as well as the analysis of single-cell sequencing data. Besides, my other research interests are systems biology and structural biology. I am fascinated by deciphering intricate biological networks by capitalizing on computational modeling algorithms, which show promise to illuminate the mechanisms of biomolecules.
 缂傚倸鍊搁崐鐑芥倿閿旂晫浠氭繝娈垮枟鑿ч柛鏂块叄閸┾偓妞ゆ帊绀侀崰鏇犵磼閻樺啿鐏撮柟顔哄劦閺佹捇鏁撻敓锟� | 闂傚倷娴囨竟鍫焊濞嗘劖鍙忛柟顖嗗苯娈ㄦ繝鐢靛У閼归箖宕f繝鍥х婵烇綆鍓欓悘顏嗘偖濠靛鈷戦柛娑橈工缁楁帗淇婇锝庢疁闁诡喖鎼~婊堟偤缁€鏄筺闂傚倷鐒︾€笛呯矙閹烘鍤屽Δ锝呭枤閺佸﹪鏌″鍐ㄥ闁崇粯妫冮弻銈嗘叏閹邦兘鍋撳Δ鍛剹閹兼番鍔嶉悡鐘碘偓瑙勬礀濞层倖绂掗埡鍛厓闁靛濡囬惌鎺楁煙閾忣偅灏伴柟宄版嚇閹粌螣閸忓吋顔忛梻鍌欒兌閸嬨劑宕曢懠顒€鍨濋悘鐐靛亾閸欏繘鏌i幇顔煎妺闁绘帒顭烽弻锝夊棘閸喗鍊梺鍝勬閻╊垶寮诲澶娢ㄩ柨鏂垮⒔椤斿洨绱撴担鐟版暰闁瑰嚖鎷�>>
缂傚倸鍊搁崐鐑芥倿閿旂晫浠氭繝娈垮枟鑿ч柛鏂块叄閸┾偓妞ゆ帊绀侀崰鏇犵磼閻樺啿鐏撮柟顔哄劦閺佹捇鏁撻敓锟� | 闂傚倷娴囨竟鍫焊濞嗘劖鍙忛柟顖嗗苯娈ㄦ繝鐢靛У閼归箖宕f繝鍥х婵烇綆鍓欓悘顏嗘偖濠靛鈷戦柛娑橈工缁楁帗淇婇锝庢疁闁诡喖鎼~婊堟偤缁€鏄筺闂傚倷鐒︾€笛呯矙閹烘鍤屽Δ锝呭枤閺佸﹪鏌″鍐ㄥ闁崇粯妫冮弻銈嗘叏閹邦兘鍋撳Δ鍛剹閹兼番鍔嶉悡鐘碘偓瑙勬礀濞层倖绂掗埡鍛厓闁靛濡囬惌鎺楁煙閾忣偅灏伴柟宄版嚇閹粌螣閸忓吋顔忛梻鍌欒兌閸嬨劑宕曢懠顒€鍨濋悘鐐靛亾閸欏繘鏌i幇顔煎妺闁绘帒顭烽弻锝夊棘閸喗鍊梺鍝勬閻╊垶寮诲澶娢ㄩ柨鏂垮⒔椤斿洨绱撴担鐟版暰闁瑰嚖鎷�>>  闂傚倷绶氬ḿ褍螞濡ゅ懏鍤愭い鏍仜妗呴梺鎼炲劗閺呮瑩鎮炴繝鍥╁彄闁搞儯鍔庨妴鎺楁煟閹邦剨鍔熺紒杈ㄥ笧閹峰鎼归銈冨仒闂備胶枪椤戝棛绮欓幒鏃傜煓濠㈣泛顑嗛崕鐔兼煃閵夈儱绾ф俊顖氾躬閺岀喖宕楅崗鐓庡攭濡炪們鍎查幑鍥€佸鈧獮鏍ㄦ媴閻熼鍝楅梻浣虹《閸撴繈宕濊箛娑樼;闁圭偓鍓氶崥瀣煕閳╁喚娈樻俊顖氬閺岋絾鎯旈妸锔介敪闂佸憡鑹鹃澶愮嵁閹达附鏅插璺猴功椤斿懘姊洪棃娑辨▓闁搞劌纾划鍫熷緞閹邦厾鍘介梺褰掑亰閸犳宕甸埀顒勬⒑閹肩偛鈧倝宕抽敐鍛殾闁跨喓濮寸粈鍐煙缂佹ê绗╂い鏂款樀濮婅櫣绮欑捄銊ь唶缂備礁顦遍崗妯虹暦瑜版帒纾兼繛鎴濈-閻姊虹化鏇炲⒉妞ゃ劌鎳愬濠囧锤濡や胶鍘介梺闈涚箞閸婃牕鈽夎閺岋綁鍩勯崘顏呭櫚闂佽鍟崶褔鍞堕梺缁樻椤ユ挻绂掗幇鐗堢厽閹肩补鍓濈拹锟犳煕鐎n倿缂氱紒杈╁仧閳ь剨缍嗛埀顑惧灩缂嶅﹪寮婚妸褉鍋撻敐搴樺亾椤栥倖瀚�>>
闂傚倷绶氬ḿ褍螞濡ゅ懏鍤愭い鏍仜妗呴梺鎼炲劗閺呮瑩鎮炴繝鍥╁彄闁搞儯鍔庨妴鎺楁煟閹邦剨鍔熺紒杈ㄥ笧閹峰鎼归銈冨仒闂備胶枪椤戝棛绮欓幒鏃傜煓濠㈣泛顑嗛崕鐔兼煃閵夈儱绾ф俊顖氾躬閺岀喖宕楅崗鐓庡攭濡炪們鍎查幑鍥€佸鈧獮鏍ㄦ媴閻熼鍝楅梻浣虹《閸撴繈宕濊箛娑樼;闁圭偓鍓氶崥瀣煕閳╁喚娈樻俊顖氬閺岋絾鎯旈妸锔介敪闂佸憡鑹鹃澶愮嵁閹达附鏅插璺猴功椤斿懘姊洪棃娑辨▓闁搞劌纾划鍫熷緞閹邦厾鍘介梺褰掑亰閸犳宕甸埀顒勬⒑閹肩偛鈧倝宕抽敐鍛殾闁跨喓濮寸粈鍐煙缂佹ê绗╂い鏂款樀濮婅櫣绮欑捄銊ь唶缂備礁顦遍崗妯虹暦瑜版帒纾兼繛鎴濈-閻姊虹化鏇炲⒉妞ゃ劌鎳愬濠囧锤濡や胶鍘介梺闈涚箞閸婃牕鈽夎閺岋綁鍩勯崘顏呭櫚闂佽鍟崶褔鍞堕梺缁樻椤ユ挻绂掗幇鐗堢厽閹肩补鍓濈拹锟犳煕鐎n倿缂氱紒杈╁仧閳ь剨缍嗛埀顑惧灩缂嶅﹪寮婚妸褉鍋撻敐搴樺亾椤栥倖瀚�>>  闂傚倷娴囬~澶婎渻閸ф鍌ㄥΔ锝呭枦缂嶆牗绻濋棃娑卞剰缁绢厸鍋撻梻浣告惈濞层劍鎱ㄩ悜鑺ュ剹閹兼番鍔嶉悡鐘碘偓瑙勬礀濞层倖绂掗敂鍓х<濞达絽鎽滈幗鐘绘煙椤栨稒顥堥柡浣规崌閺佹捇鏁撻敓锟�-濠电姷鏁搁崕鎴犲緤閽樺鏆︽い鎺戝閻鏌涢埄鍐$細妞も晜鐓¢弻娑㈠焺閸愭儳姣€闂佸湱鍎ら幐缁樼閵堝棛绠鹃柛鈩兩戠亸顏堟煃瑜滈崜娆戠矓閹绢噯缍栭柕蹇嬪€栭弲鏌ユ煕濞戝崬鏋﹂柛姘煎亰濮婅櫣鎮伴垾鍏呭闂備浇宕甸崰鎰熆濡偐绀婂〒姘e亾闁哄瞼鍠撻埀顒佺⊕閿氶柣蹇ョ稻缁绘繃绻濋崘銊т淮濡炪倖娲╃紞渚€宕洪埀顒併亜閹烘垵鈧嘲鈻介鍫熺厸闁搞儮鏅涢灞剧箾閼测晛鈻堥柟顔筋殔閳藉宕樺顔藉枠闂備胶绮悧鐐测枍閿濆洤鍨濇慨妯挎硾缁€鍫澝归敐鍛础闁哄棛濞€濮婃椽鎮烽幍顔婚偗闂佺ǹ顑嗛幑鍥蓟閿熺姴閱囨い鎺嶇椤忥拷>>
闂傚倷娴囬~澶婎渻閸ф鍌ㄥΔ锝呭枦缂嶆牗绻濋棃娑卞剰缁绢厸鍋撻梻浣告惈濞层劍鎱ㄩ悜鑺ュ剹閹兼番鍔嶉悡鐘碘偓瑙勬礀濞层倖绂掗敂鍓х<濞达絽鎽滈幗鐘绘煙椤栨稒顥堥柡浣规崌閺佹捇鏁撻敓锟�-濠电姷鏁搁崕鎴犲緤閽樺鏆︽い鎺戝閻鏌涢埄鍐$細妞も晜鐓¢弻娑㈠焺閸愭儳姣€闂佸湱鍎ら幐缁樼閵堝棛绠鹃柛鈩兩戠亸顏堟煃瑜滈崜娆戠矓閹绢噯缍栭柕蹇嬪€栭弲鏌ユ煕濞戝崬鏋﹂柛姘煎亰濮婅櫣鎮伴垾鍏呭闂備浇宕甸崰鎰熆濡偐绀婂〒姘e亾闁哄瞼鍠撻埀顒佺⊕閿氶柣蹇ョ稻缁绘繃绻濋崘銊т淮濡炪倖娲╃紞渚€宕洪埀顒併亜閹烘垵鈧嘲鈻介鍫熺厸闁搞儮鏅涢灞剧箾閼测晛鈻堥柟顔筋殔閳藉宕樺顔藉枠闂備胶绮悧鐐测枍閿濆洤鍨濇慨妯挎硾缁€鍫澝归敐鍛础闁哄棛濞€濮婃椽鎮烽幍顔婚偗闂佺ǹ顑嗛幑鍥蓟閿熺姴閱囨い鎺嶇椤忥拷>>  婵犵數鍋為崹鍫曞箰婵犳碍鍋嬫繝濠傜墕閻ょ偓銇勯幇鍫曟濠殿垰鍚嬫穱濠囧Χ閸屾矮澹曢梻浣筋嚃閸犳危閺勭灃rmo Fisher闂備浇宕垫慨宥夊礋椤撶喎鍨辩紓鍌欑贰閸n噣宕归柆宓ュ洦瀵肩€涙ê鈧兘鏌熷▓鍨灓闁硅櫕鍔欓弻锝堢疀閹惧墎顔夊┑顔硷工椤兘鏁愰悙鍝勫唨妞ゆ挾鍋熼鍛存⒑闁偛鑻晶瀛橆殽閻愯尙绠伴柍钘夘槸楗即鍩€椤掑倸鍨濇い顏勵吙eld Application Scientist闂傚倷绶氬ḿ褍螞閺冣偓閹柨銆掓稊绨乪ting Develop缂傚倸鍊烽悞锔剧矙閹次诲洭鎼归銈庢锤闁诲孩绋掕摫濠殿垰顕埀顒€绠嶉崕杈╂崲閹烘梻鐭嗗鑸靛姈閸嬶綁鏌涢妷褍鍘存俊缁㈠櫍閺屾稒鎯旈敍鍕唺闂佸綊顥撴繛鈧柡灞诲妿閳ь剨缍嗛崜娆忣嚕閵娾晜鈷戞繛鑼额唺缁ㄧ粯銇勯幋婵囧枠鐎殿噮鍋婇獮妯肩磼濡桨缃曢梻浣稿暱閹碱偊骞婃径鎰;闁圭偓鍓氬ḿ鈺傘亜閹板墎鎮兼繝褍瀚板楦裤亹閹烘挻閿紓浣割槸椤曨厾鍒掗埡鍛唶闁哄洨鍋炲畵宥夋⒑缂佹ê鐏﹀畝锝呮健瀹曠儤寰勯幇顓犲幗闂佸啿鎼敃銈呪枍濞嗘垹纾奸柟鎻掝儐鐎氾拷>>
婵犵數鍋為崹鍫曞箰婵犳碍鍋嬫繝濠傜墕閻ょ偓銇勯幇鍫曟濠殿垰鍚嬫穱濠囧Χ閸屾矮澹曢梻浣筋嚃閸犳危閺勭灃rmo Fisher闂備浇宕垫慨宥夊礋椤撶喎鍨辩紓鍌欑贰閸n噣宕归柆宓ュ洦瀵肩€涙ê鈧兘鏌熷▓鍨灓闁硅櫕鍔欓弻锝堢疀閹惧墎顔夊┑顔硷工椤兘鏁愰悙鍝勫唨妞ゆ挾鍋熼鍛存⒑闁偛鑻晶瀛橆殽閻愯尙绠伴柍钘夘槸楗即鍩€椤掑倸鍨濇い顏勵吙eld Application Scientist闂傚倷绶氬ḿ褍螞閺冣偓閹柨銆掓稊绨乪ting Develop缂傚倸鍊烽悞锔剧矙閹次诲洭鎼归銈庢锤闁诲孩绋掕摫濠殿垰顕埀顒€绠嶉崕杈╂崲閹烘梻鐭嗗鑸靛姈閸嬶綁鏌涢妷褍鍘存俊缁㈠櫍閺屾稒鎯旈敍鍕唺闂佸綊顥撴繛鈧柡灞诲妿閳ь剨缍嗛崜娆忣嚕閵娾晜鈷戞繛鑼额唺缁ㄧ粯銇勯幋婵囧枠鐎殿噮鍋婇獮妯肩磼濡桨缃曢梻浣稿暱閹碱偊骞婃径鎰;闁圭偓鍓氬ḿ鈺傘亜閹板墎鎮兼繝褍瀚板楦裤亹閹烘挻閿紓浣割槸椤曨厾鍒掗埡鍛唶闁哄洨鍋炲畵宥夋⒑缂佹ê鐏﹀畝锝呮健瀹曠儤寰勯幇顓犲幗闂佸啿鎼敃銈呪枍濞嗘垹纾奸柟鎻掝儐鐎氾拷>>  闂傚倷绀侀幉锟犲礄瑜版帒绀堟繝闈涱儛閺佸鏌嶉崫鍕偓瑙勭閵堝鐓曢柕澶嬪灥鐎氼剚绂嶉柆宥嗏拺闁圭ǹ娴烽妴鎺楁煕閻樿櫕绀堢紒顔芥⒒閳ь剚绋掕摫濞存嚎鍊濋弻銊╂偆閸屾稑顏�14婵犵數濮伴崹褰掓倶閸儱鐤炬繛鎴欏灪閸嬵亪鏌涢弴銊ュ⒒婵炲牏鏅埀顒€鍘滈崑鎾绘煃瑜滈崜鐔煎箖娴兼潙唯闁宠桨鑳堕悡鎴︽⒑閹稿海绠撴繛璇х畵閹箖鏌嗗鍡欏幘闂佸憡鐟ラˇ杈╃不濮樿埖鐓曢柟鐐綑椤ュ绱掓潏銊ユ诞鐎规洖宕~婵嬵敆閳ь剚鎱ㄩ崘顔解拺闁圭ǹ楠搁崰鏇㈡煙閸戙倖瀚�>>
闂傚倷绀侀幉锟犲礄瑜版帒绀堟繝闈涱儛閺佸鏌嶉崫鍕偓瑙勭閵堝鐓曢柕澶嬪灥鐎氼剚绂嶉柆宥嗏拺闁圭ǹ娴烽妴鎺楁煕閻樿櫕绀堢紒顔芥⒒閳ь剚绋掕摫濞存嚎鍊濋弻銊╂偆閸屾稑顏�14婵犵數濮伴崹褰掓倶閸儱鐤炬繛鎴欏灪閸嬵亪鏌涢弴銊ュ⒒婵炲牏鏅埀顒€鍘滈崑鎾绘煃瑜滈崜鐔煎箖娴兼潙唯闁宠桨鑳堕悡鎴︽⒑閹稿海绠撴繛璇х畵閹箖鏌嗗鍡欏幘闂佸憡鐟ラˇ杈╃不濮樿埖鐓曢柟鐐綑椤ュ绱掓潏銊ユ诞鐎规洖宕~婵嬵敆閳ь剚鎱ㄩ崘顔解拺闁圭ǹ楠搁崰鏇㈡煙閸戙倖瀚�>>  生物通微信公众号
生物通微信公众号
知名企业招聘
今日动态 | 人才市场 | 新技术专栏 | 中国科学人 | 云展台 | BioHot | 云讲堂直播 | 会展中心 | 特价专栏 | 技术快讯 | 免费试用
版权所有 生物通
Copyright© eBiotrade.com, All Rights Reserved
联系信箱:
粤ICP备09063491号
