
-

生物通官微
陪你抓住生命科技
跳动的脉搏
万种原生生物基因组计划取得阶段性进展
【字体: 大 中 小 】 时间:2024年03月09日 来源:中国科学院水生生物研究所
编辑推荐:
数据通过万种原生生物基因组数据库( P10K database , https://ngdc.cncb.ac.cn/p10k/ )释放共享,相关论文以 “The P10K database: a data portal for the protist 10000 genomes project” 为题发表于 Nucleic Acids Research
原生生物(Protist)是一大类单细胞真核生物的集合,包括单细胞真核藻类和原生动物等,组成了原生生物界。原生生物具有高度多样性,广泛分布于各类水环境中,在生态平衡、物质和能量循环、环境健康、动植物疾病发生等方面发挥重要作用。它们是水生态系统的重要组分,是重要的初级生产力和氧气的制造者、碳循环的关键参与者,是水产动物的优良饵料、人类的营养品、生物能源,是水环境的“哨兵”、水华和赤潮的重要元凶,是人、畜、禽、鱼疾病的重要病原、互利共生的“好伙伴”。
NCBI分类系统已记录的原生生物种类超过6万种,未知数量难以估量。2019年12月,由中国科学院水生生物研究所(水生所)牵头发起了万种原生生物基因组计划(Protist 10,000 Genomes Project,P10K),旨在建立一个大规模的原生生物遗传资源数据库,变革原生生物遗传资源数据极度缺乏的局面。

“万种原生生物基因组计划(P10K)”的标志。设计理念:标志由不同原生生物组成。其中钟虫(纤毛虫)代表字母“P”,裸藻(鞭毛虫)和团藻(绿藻)的组合代表数字“10”,阿米巴代表字母“K”。双螺旋既代表DNA又代表水波,即原生生物生存的水环境。“P”中喷发出诸多原生生物,代表1676年列文虎克发现钟虫之后,高度多样的各种原生生物被发现。
近日,由中国科学院水生生物研究所及北京基因组研究所(国家生物信息中心)联合发布了P10K第一批数据。数据通过万种原生生物基因组数据库(P10K database,https://ngdc.cncb.ac.cn/p10k/)释放共享,相关论文以“The P10K database: a data portal for the protist 10000 genomes project”为题发表于Nucleic Acids Research。P10K第一批数据共收录了2959个原生生物数据集,包括1601个基因组和1358个转录组数据集,覆盖了原生生物75%的纲和45%的目。其中,从公共数据库整合了1858个数据集,P10K团队新测序了1101个数据集,以原生动物纤毛虫(Ciliate)为主。新测序数据将原生生物数据集规模整体提升了37%。新测序的样品由P10K团队从国内多种生境中采集、分离。对于不能实验室培养的绝大部分原生动物,采用了单细胞测序方法,占新测序数据的98%。为解决大规模单细胞组学数据的分析问题,P10K团队研发了一套针对原生生物单细胞测序数据的组装、去污染、物种鉴定、基因注释和评估的标准化分析流程。质量评估显示,该流程注释的基因组与公共数据库发布的基因组具有相似比例的中高等质量数据。
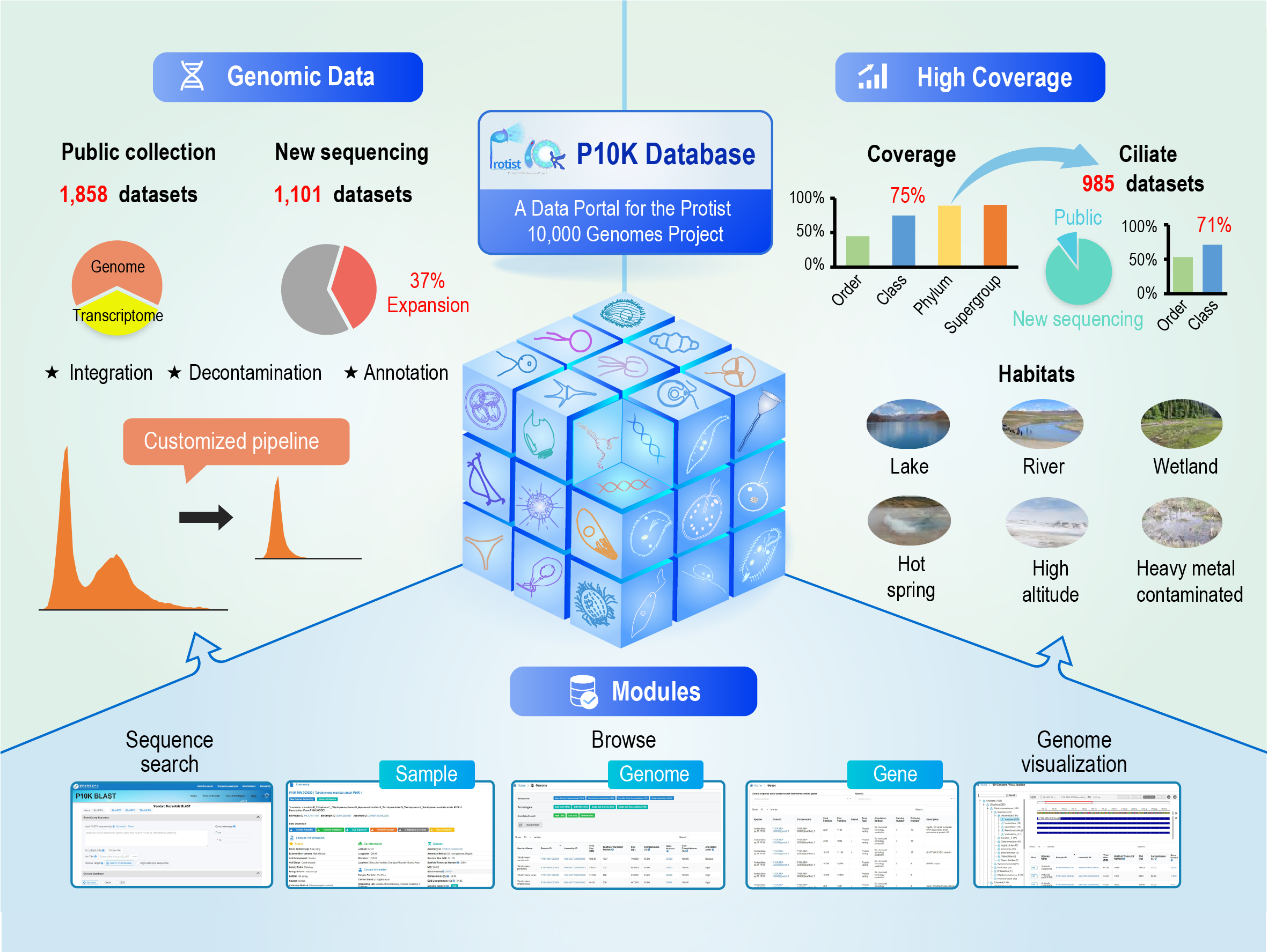
P10K数据库的特点和功能
水生所缪炜研究员、北京基因组所章张研究员与马利娜副研究员为该文共同通讯作者,水生所博士研究生高欣欣、陈凯助理研究员、熊杰研究员、北京基因组所邹东高级工程师为该文共同第一作者。该工作得到了科技部国家重点研发计划、中国科学院先导专项、国际合作计划、青年创新促进会、国家自然科学基金和IUBS开放生物多样性和健康大数据计划的资助,以及国家水生生物种质资源库和中国科学院超级计算武汉分中心的支持。
作为“万种原生生物基因组计划”的重要组成部分,P10K数据库的建立和数据共享将有助于推动对真核生物和多细胞生物起源、真核生物多样性、原生生物的极端环境适应以及微生物互作等重要基础科学问题的研究。与此同时,这项计划将促进对与生态环境保护、污染物降解和转化、营养健康以及疾病防治相关的原生生物遗传资源的挖掘和潜在应用。同时,鉴于原生生物是浮游生物的关键组成部分,P10K数据库还将为基于环境DNA的浮游生物鉴定提供支持,助力水生态健康评价。
尤为重要的是,P10K数据库还建立了国家水生生物种质资源库/国家寄生虫资源库(活体种质资源)和国家基因组科学数据中心(遗传资源)之间的紧密联系,对于促进国家科技资源共享服务平台的信息互联互通和数据共享具有重要意义。
P10K计划链接:
https://www.cell.com/the-innovation/fulltext/S2666-6758(20)30061-8
P10K 数据库论文原文:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad992/7335744