
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Advanced Science | 北京大学何航与邓兴旺团队揭示植物基因组进化中的染色质拓扑结构域变化规律
【字体: 大 中 小 】 时间:2024年12月31日 来源:北京大学现代农学院
编辑推荐:
2024年12月28日,北京大学现代农学院何航与邓兴旺团队与中科院武汉植物园李大卫研究员在Advanced Science 合作发表论文Chromatin topological domains associate with the rapid formation of tandem duplicates in plants ,揭示了植物染色体Rabl 构型的产生规律,并发现一类由串联重复基因簇形成的TAD-like 结构参与了植物基因组进化
在细胞间期,染色质丝在细胞核内的组织结构遵循一个基本原则——区室化。在此基础上,condensin II帮助各条染色体盘踞形成染色体疆域(chromosome territory);而cohesin则在DNA上滑动,挤出染色质环,并与CTCF蛋白共同介导形成染色质拓扑结构域(TADs)。目前,植物领域对染色质高级结构的研究存在着两个主要问题:1. 不同植物的染色体疆域结构由什么决定?有的植物,比如小麦,有完整的condensin II,但其染色体却没有形成疆域,反而呈现染色体臂延着丝粒对折的Rabl构型1。2. 植物中类TAD结构如何形成,且是否具有生物学功能?植物中缺少CTCF蛋白,但在很多植物的Hi-C图谱上也能观察到类似TAD的三角形结构,例如地钱中的TCP1-rich TAD2,然而这些结构的功能尚不明确。

2024年12月28日,北京大学现代农学院何航与邓兴旺团队与中科院武汉植物园李大卫研究员在Advanced Science合作发表论文Chromatin topological domains associate with the rapid formation of tandem duplicates in plants,揭示了植物染色体Rabl构型的产生规律,并发现一类由串联重复基因簇形成的TAD-like结构参与了植物基因组进化。
研究收集了10+不同植物基因组和Hi-C测序数据,发现Rabl构型的出现与否与植物基因组大小密切相关(图1)。而在更精细的尺度上,串联重复基因簇(TDGC)总是偏向性形成一个拓扑结构域。这类结构域的形成不依赖于组蛋白H3K27me3修饰,且在物种间非常保守,但其结构上又没有典型的CTCF介导的顶端互作,我们因此称之为TAD-like domain(图2)。
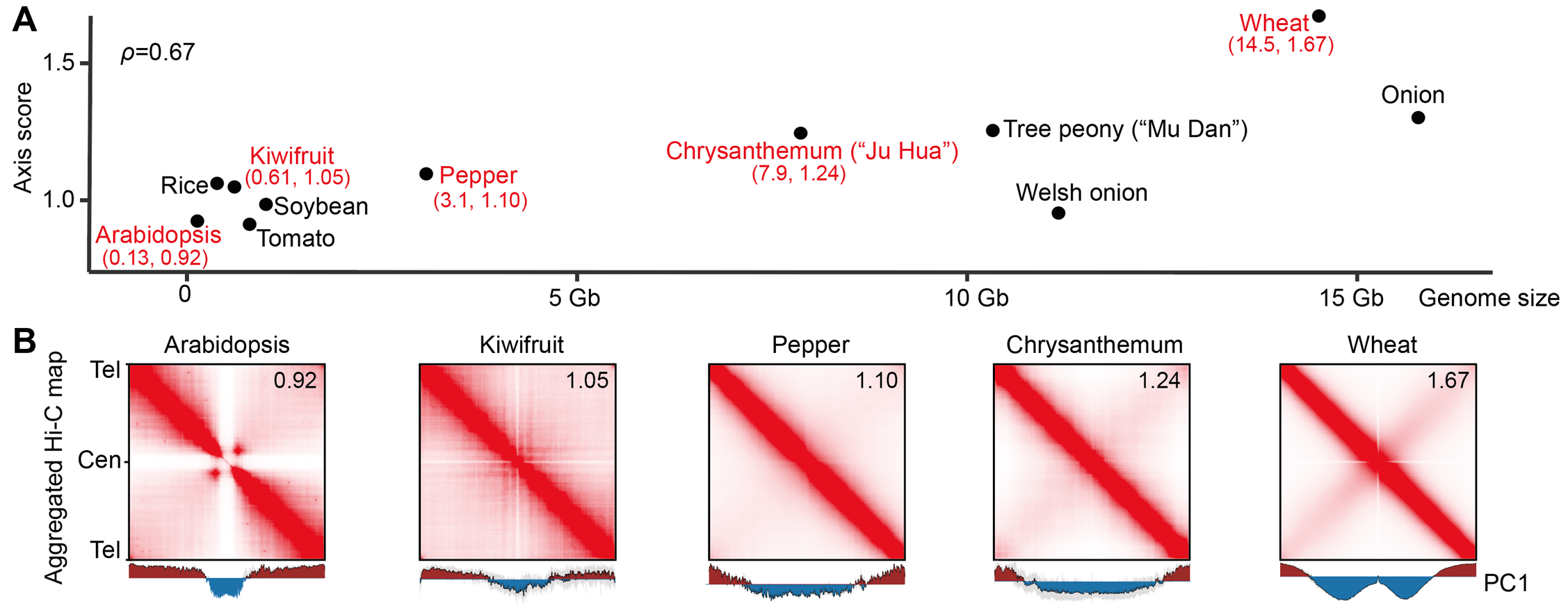
图1. 植物染色质结构与基因组大小相关。
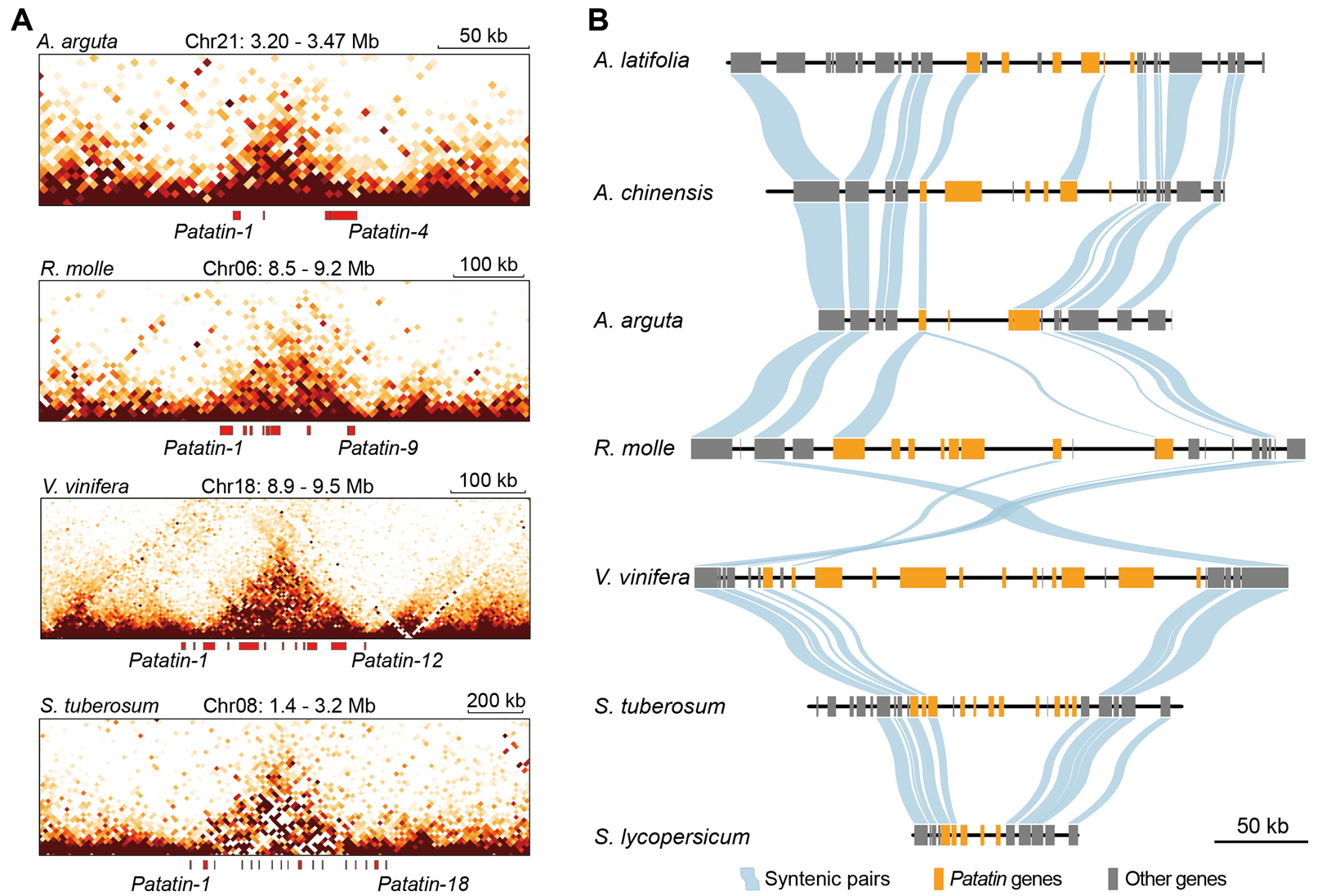
图2. TDGC形成的TAD-like domain在物种间非常保守。
关于TAD-like domain的功能存在两种假设:1. 染色质结构域可以帮助其内的基因共享转录机器和表观修饰,以实现基因协同表达3。2. 在本研究中,我们发现越大的基因簇越倾向于形成TAD-like domain,反之推测,这些结构域参与促进了基因的串联复制。在中华猕猴桃基因组中的计算结果显示,TAD-like domain对基因共表达的作用非常有限。值得注意的是,染色质结构域内部发生基因串联重复的频率要显著高于结构域外部。该现象在软枣猕猴桃中同样存在,而在猕猴桃近亲——黄花杜鹃中并没有发现(图3)。因此,TAD-like domain或可以加速串联重复基因的产生,但该功能未能在物种演化历程上被完全地保持下来。
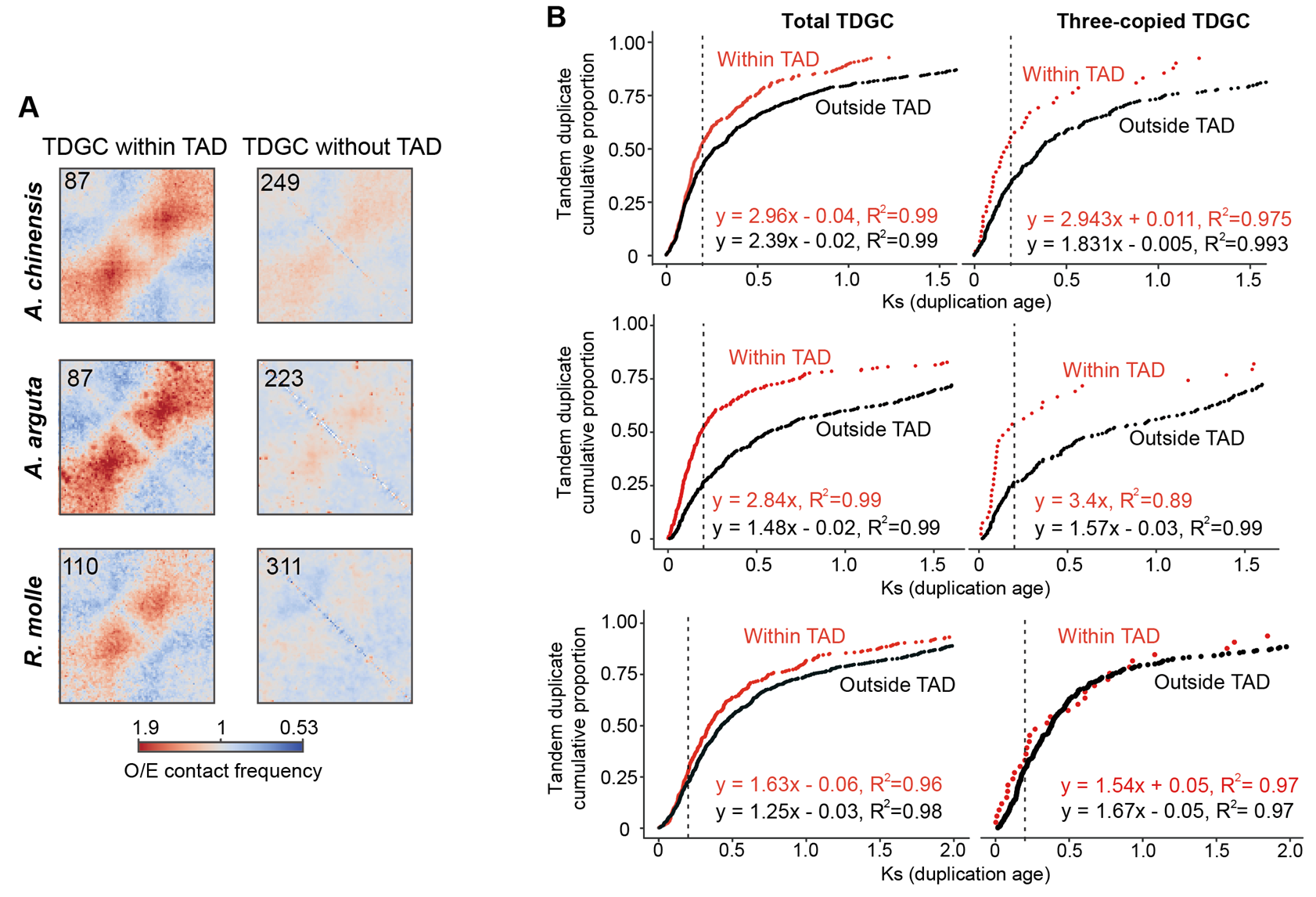
图3. 在猕猴桃中,TAD-like domain内部基因串联复制频率更高。
北京大学现代农学院何航研究员、邓兴旺教授和中科院武汉植物园李大卫研究员为该论文的共同通讯作者。北京大学博士生马妮、北京大学现代农业研究院科研助理李晓鹏和副研究员次东为共同第一作者。该研究受国家自然科学基金和山东省重点研发计划资助。北京大学现代农业研究院超算中心为本研究数据存储和计算提供支持。
参考文献: