
-

生物通官微
陪你抓住生命科技
跳动的脉搏
APS | AlphaFold3预测GPCR-配体复合物的局限性评测:实验结构仍是关键
【字体: 大 中 小 】 时间:2024年12月12日 来源:中国科学院上海药物研究所
编辑推荐:
2024年12月6日,中国科学院上海药物研究所徐华强团队在Acta Pharmacologica Sinica发表文章“AlphaFold3 versus experimental structures: Assessment of the accuracy in ligand-bound G protein-coupled receptors”
2024年12月6日,中国科学院上海药物研究所徐华强团队在Acta Pharmacologica Sinica发表文章“AlphaFold3 versus experimental structures: Assessment of the accuracy in ligand-bound G protein-coupled receptors”。该文章收集了AlphaFold3(AF3)发布后的GPCR-配体复合物实验结构,并从整体结构的准确性、口袋预测能力、复合物预测能力和可变区预测效果方面全面评估了AF3在GPCR-配体复合物方面的表现,并为进一步改进蛋白质结构预测工具提出了建议。

AF3于2024年5月发布,基于扩散模型提供了复合物从头建模的解决方案。在过去的半年中,已有多个相关工作复现了AF3的预测性能(如Protenix、chai-1等),并开放源代码供学术界使用。然而,AF3的预测表现尚缺乏在真实应用场景中的评估,特别是其新加入的配体预测模块。为此,考虑到GPCR作为重要的药物靶标,其与配体的相互作用是药物开发的重要方向,且具有高度复杂性,结构生物学也在此领域做出了大量工作,本文使用AF3服务器对其配体预测能力进行了评测,包括整体结构准确度、配体定位、口袋形状和受体结构变化。结果显示,尽管AF3有所进步,并可提供一定参考,但鉴于其在口袋和整体预测方面存在的问题,实验结构解析仍然是必要的。本文还对以AF3为代表的一系列结构预测工具的未来发展方向提出了潜在的有价值的建议。
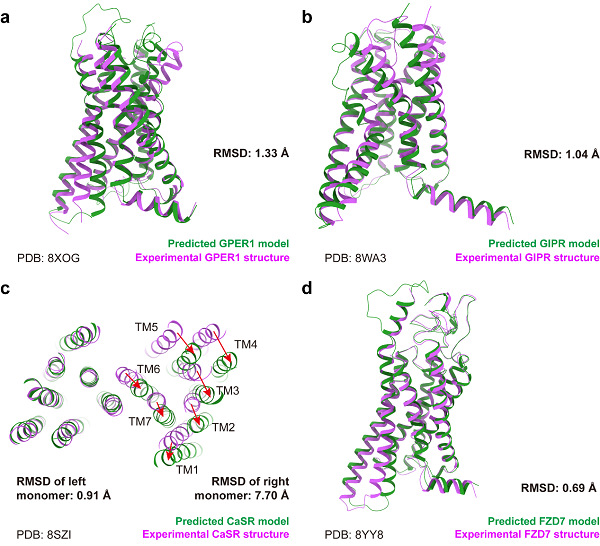
图1 AF3预测结构和实验结构的整体对比
本文为确保对比结果不受训练数据影响,选取了35个在AF3文章投稿后发布的GPCR结构。整体来看,AF3相比前代AF2在结构预测能力上有所提升,所有GPCR受体部分的Cα均方根偏差(RMSD)从1.04±0.24埃降低至0.93±0.24埃。加入可预测的配体后,受体的RMSD为1.11±0.46埃,表明整体预测能力未因配体的加入而受到影响。图1展示了四类GPCR的预测结果。在A、B、F类单体预测中,AF3仅在一些天生可变的区域,如关键激活螺旋TM6,存在和实验结果不一致的预测,因此对激活机制的准确探究可能仍需进一步提升。然而,对于二聚的C类GPCR,AF3能够准确预测单体的部分结构,但在组合两个单体时出现错误,导致了7.70埃的RMSD,所以在大蛋白复合体的预测方面,AF3还有待提升。
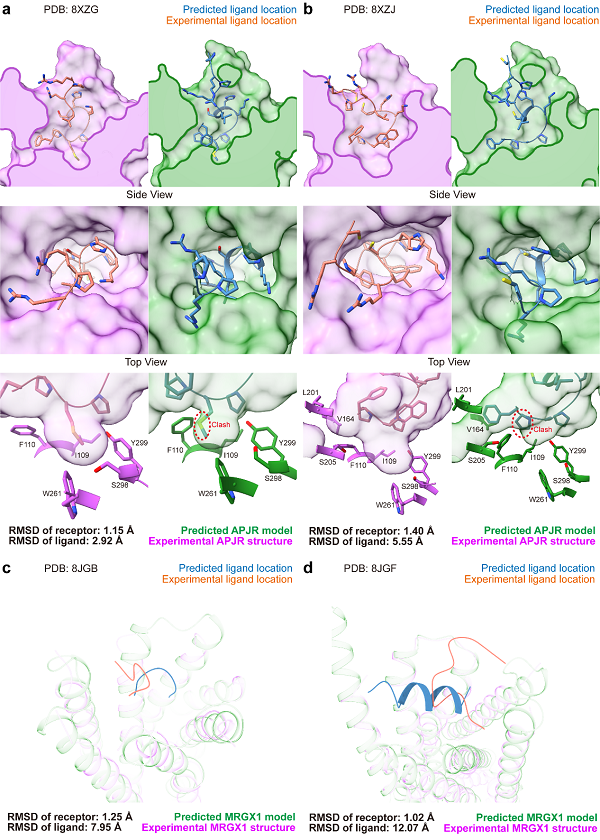
图2 AF3预测结构和实验结构在多肽类配体上的比较
尽管AF3声称能够预测通用小分子配体,但在本文撰写时该功能尚未开放。因此,本文对比评估了其在离子、多肽和蛋白类配体上的预测性能。
在多肽配体部分,配体的最小重原子RMSD也达到了2.24埃,未达到实验精度(2埃)。如图2A所示,即便是APJR中的最佳预测结果,也存在配体与口袋残基侧链构象的不一致,口袋在预测结构中有所收缩,并且预测结构中存在一定程度的受体-配体碰撞。在同一受体的非天然配体中,AF3的表现与实验结构的偏差更大。如图2B所示,配体的重原子RMSD高达5.55埃,主链的摆放方向明显不同,口袋中的疏水相互作用和侧链构象也未能得到相似的重建,同时仍存在受体-配体碰撞现象。
对于MRGX1中较大且平坦的更具挑战性的口袋,配体的重原子RMSD均超过了7埃。在图2C中,Conorfamide-Tx2复合物的建模结构与实验结构中的配体方向呈垂直关系,RMSD达到7.95埃。此外,在BAM8-22复合物的建模中,配体被预测为较为稳定的α螺旋结构,而实验解析的结构却为loop。这可能与AF3训练集中较多的α螺旋结构有关,这一现象也对AI模型的泛化能力提出了挑战。
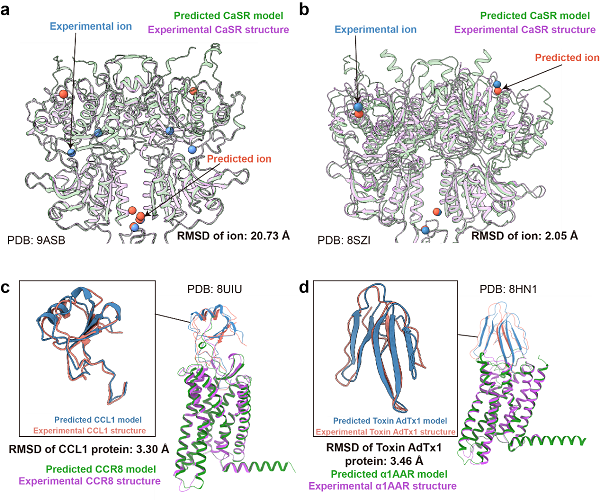
图3 AF3预测结构和实验结构在离子和蛋白类配体上的比较
图3展示了对离子和蛋白类配体的预测结果。在CASR(图3a、b)中,不同数量的钙离子对应不同的预测表现。当配体中包含6个钙离子时,实验结构中的钙离子位于胞外区中部,而预测模型中的钙离子则分布在上端和下端,位置差异较大(20.73埃)。相比之下,当配体中包含4个钙离子时,实验结构和预测模型中的钙离子位置均位于上端和下端,表现出较高的一致性。这表明离子数量与预测准确度之间存在相关性。
蛋白类配体的预测整体较为准确,配体的重原子RMSD均小于3.5埃,且配体在摆放位置和整体构象上与实验结构高度相似,体现了AF3在预测蛋白-蛋白相互作用方面能力的提升。
考虑到抗体分子在药物开发中的重要性,本文特别将抗体作为单独模块进行评估,并纳入了重要药物靶标TSHR。如图4a-d所示,失活态的抗原-抗体复合物预测表现不佳。尽管存在抑制剂形式的抗体,AF3仍将TSHR胞外区预测为激活态构象,且抗体的朝向由指向细胞膜转变为与细胞膜平行,说明AF3预测结构可能受到激活态构象的影响。在CCR8的自主研发抑制剂抗体复合物预测中,AF3预测了与实验结构不同的表位和抗体朝向,导致RMSD超过30埃,未能成功识别抗体和受体的结合。
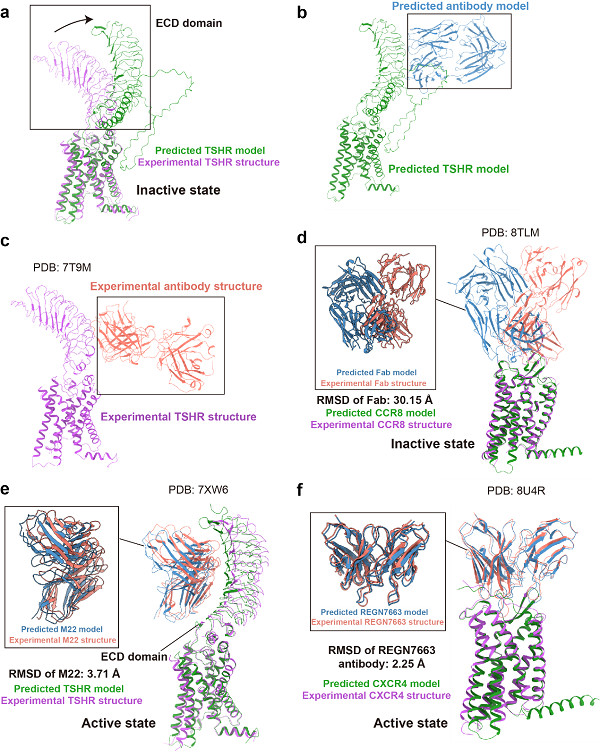
图4 AF3预测结构和实验结构在GPCR-抗体复合物上的比较
不过,在激活型抗体的预测中,AF3表现良好。例如,TSHR的M22激动型抗体的表位预测准确,整体结构相似,尽管主链存在轻微错位,导致配体的RMSD为3.71埃,但胞外区(ECD)的放置基本准确(图4e)。商业抗体REGN7663与CXCR4的复合物结构整体RMSD仅为2.25埃,显示出非常高的预测准确性。本文推测,AF3在预测较新的抗体复合物时,仍需谨慎验证其预测结果。
考虑到GPCR与G蛋白相互作用的重要性,本文基于AF3提供的蛋白复合体建模功能对其进行了评测。整体而言,加入G蛋白后,受体本身的预测结果得到了提升。对于数据库中有大量结构的A类GPCR(图5a),CXCR4与G蛋白三聚体的复合物呈现出正确的建模结构,包括TM6的开启构象、G蛋白三聚体及大部分界面残基。然而,部分界面残基(如F354、K349等)表现出不同的侧链构象,这些细微的构象差异可能会影响基于结构的药物设计。
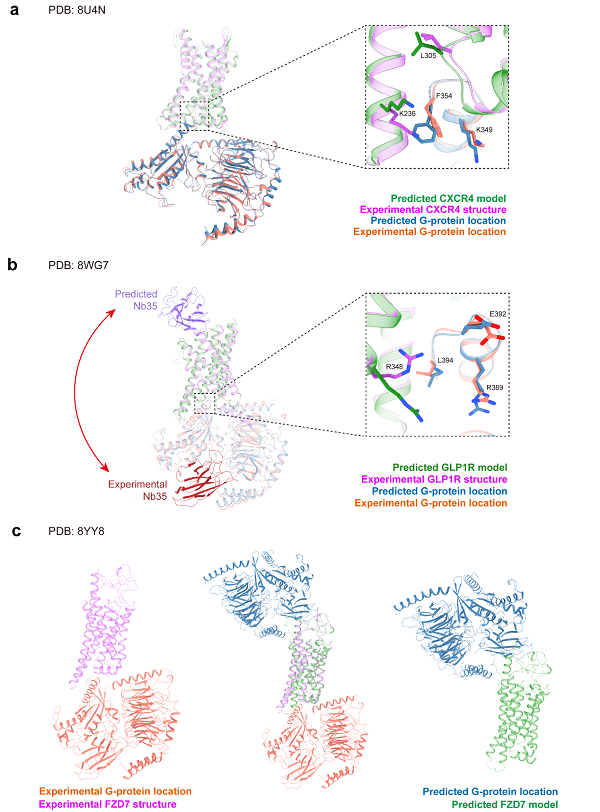
图5 AF3预测结构和实验结构在GPCR-G蛋白复合体上的比较
在B类GPCR的建模中,尽管GPCR-G蛋白复合物仍然得到了正确建模,但抗体被错误地建模在胞外区,实际结合位置应该位于G蛋白三聚体附近,这反映了AF3在建模抗体结构时的不确定性。此外,G蛋白与受体的界面残基侧链朝向也存在差异。
对于F类GPCR-G蛋白三聚体的建模,尽管GPCR本身的TM6构象正确,但AF3错误地将G蛋白放置在胞外区,这与生物学原理不符。这一错误提示尽管加入G蛋白确实有助于GPCR的建模,蛋白-蛋白相互作用界面的预测仍然不完全可靠。
最后,本文还评估了AF3在关键可变区域的预测表现,如关键激活螺旋TM6和配体识别激活的loop。我们注意到,这些高可变性区域的预测存在一定的不确定性。图6a和6c中的案例表现较好,而图6b、6d和6e中的预测模型与实验结构之间则存在较大的差异。
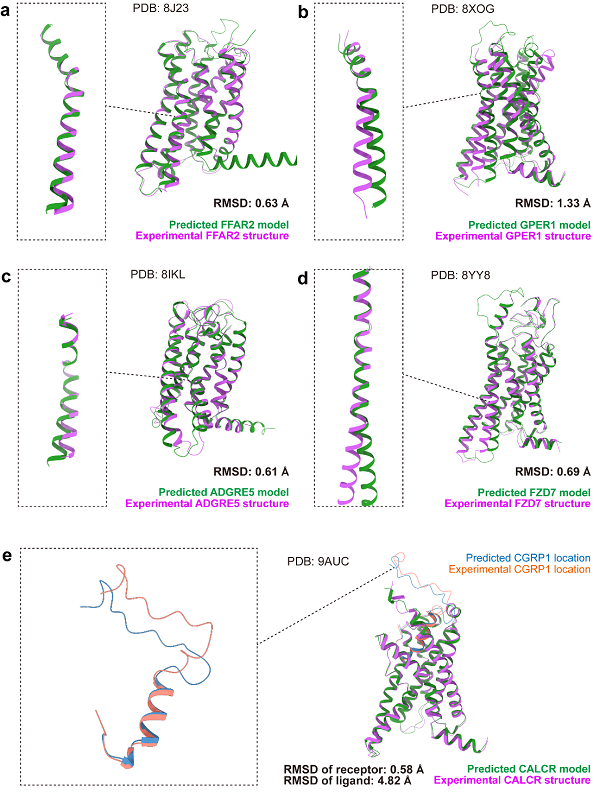
图6 AF3预测结构和实验结构在高可变性区域上的比较
此外,本文还对不同类型GPCR上AF3的结构预测表现进行了分类评估。A类和F类GPCR的整体预测较为准确,但可变区域的预测不确定性较大;B类GPCR几乎都被预测为激活态构象,这可能与训练数据集的偏差有关;C类GPCR的预测表现参差不齐,缺乏一致性。值得注意的是,AF3在C类GPCR二聚体的建模中经常出现错误的复合体组装问题,这再次强调了在药物设计中实验结构的必要性。
通过对AF3的评测,本文展示了AF3相较于前代AF2的进步,并证明了AF3在缺乏实验先验知识时依然具有一定的模型价值。然而,侧链构象的预测和四级结构的组装依然是其主要挑战。此外,AF3在不同GPCR家族的泛化能力及动态区域的预测能力也需要进一步提高。尽管AF3取得了重要进展,但目前仍不足以支持可靠的药物开发和应用,仍需依赖实验验证和算法的进一步优化,而非完全替代实验结构生物学。
图7展示了一些可能的算法改进方向。例如,引入物理化学原则的编码信息可能有助于减少配体与受体之间的碰撞;对多肽类配体进行原子级建模可能提升其预测能力;此外,类似于ESM3在结构预测中引入功能信息的做法,在AF3中引入生物学原则的信息可能帮助避免G蛋白位置预测的重大失误,基因测序信息以及突变与表型之间的关联信息也可能提供有价值的参考。在这些改进下,AF3的预测有望为序列-结构-功能关系提供更全面的理解,并为药物设计提供更强有力的支持。?
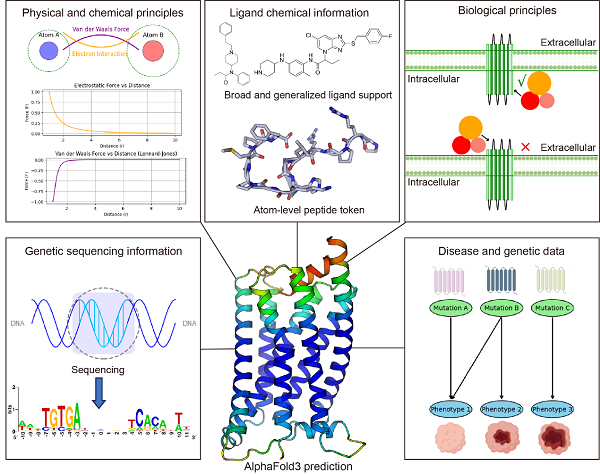
图7 可能的提升AF3预测能力的建议
综上所述,AF3的GPCR结构预测能力有所提升,但依然存在一定的能力限制,仍需要进一步实验验证。通过整合实验与计算方法,研究者对重要靶标GPCR的理解会更深刻,也更有利于药物研发。
上海药物所博士研究生何欣恒、助理研究员李俊睿、博士研究生沈诗怡为本文共同第一作者;上海药物所徐华强研究员为本文通讯作者。本研究得到了国家自然科学基金,中国科学院战略性先导科技专项,上海市科技重大专项和临港实验室的基金资助。