
-

生物通官微
陪你抓住生命科技
跳动的脉搏
生命科学学院高宁课题组揭示真核DNA复制中冈崎片段引物去除过程的新分子机制
【字体: 大 中 小 】 时间:2024年12月01日 来源:北京大学新闻网
编辑推荐:
2024年11月22日,北京大学生命科学学院、北大-清华生命科学联合中心、核糖核酸北京研究中心高宁课题组在The EMBO Journal发表了题为“Structural insight into Okazaki fragment maturation mediated by PCNA-bound FEN1 and RNaseH2”的研究论文,对含有PCNA的内源...
2024年11月22日,北京大学生命科学学院、北大-清华生命科学联合中心、核糖核酸北京研究中心高宁课题组在The EMBO Journal发表了题为“Structural insight into Okazaki fragment maturation mediated by PCNA-bound FEN1 and RNaseH2”的研究论文,对含有PCNA的内源相关复合物进行结构研究,获得了两组PCNA复合物(PCNA-FEN1、PCNA-FEN1-RNaseH2)的结构,为理解冈崎片段成熟的后期步骤提供了一系列的结构快照。

论文截图
真核生物DNA复制在复杂的染色质环境下进行,与多种分子生物学过程互相协调,例如DNA复制与修复的偶联以及DNA复制与新生核小体组装的偶联等1,2。PCNA(Proliferating cell nuclear antigen)作为DNA复制体不可缺少的辅助蛋白,在DNA复制中结合各种DNA聚合酶以赋予其持续合成的能力,也可以将多种因子募集到DNA复制、修复等DNA代谢事件发生的位点,并促进相应的生物学反应,是DNA代谢的中心枢纽之一3,4。与PCNA相互作用的蛋白超过两百多种,包括DNA复制相关蛋白(Pol δ、Pol ε、Pol η、Pol κ、FEN1、LIG1等)、解旋酶(WRN、BLM、RECQ5等)、DNA修复蛋白(MSH3、MSH6、XPG、FANCM等)、表观遗传因子(CAF-1、DNMT1、p300、HDAC1等)及参与细胞周期调控、凋亡等过程的一些蛋白分子,它们都是通过自身保守的PIP box(PCNA-interacting protein box)或PIP-like基序结合PCNA4。
真核生物PCNA是环绕双链DNA的同源三聚体,每个单体都包含一个PIP box结合位点,因此一个PCNA环可以至多结合3个含有PIP box的蛋白因子。领域内认为PCNA的协调功能是通过“工具带,toolbelt”的形式实现,即PCNA同时结合处于上下游过程的不同的酶,以提高DNA代谢相关事件的整体效率4。一个具有代表性的例子是DNA复制过程中的滞后链冈崎片段的加工成熟,参与这一过程的所有具有催化活性的酶都与PCNA有相互作用,包括Pol δ、RNaseH2、FEN1、EXO1、PIF1、DNA2以及LIG14—6。参与引物去除的主要因子是Pol δ、RNaseH2和FEN1,RNaseH2作为RNA核酸酶去除由Pol α与引物酶复合物所合成的RNA-DNA引物中的RNA;DNA聚合酶Pol δ在完成滞后链上的新生DNA链的延伸之后,会进行链置换反应,继续向前合成将临近冈崎片段中的引物顶起,形成5'-flap DNA结构;FEN1作为结构特异性的DNA核酸内切酶结合并切割5'-flap DNA;通过这一系列步骤,保真度较差的DNA引物及RNA引物得以去除。领域前期通过体外重组的方式,已经获得了PCNA-FEN1-Pol δ、PCNA-FEN1-LIG1复合物,对它们的结构和功能进行了表征,部分地验证了PCNA作为工具带的分子角色7,8。然而很多重要的机制性的问题还未得到解答,例如PCNA如何特异性地选择并协调参与同一DNA代谢事件的不同蛋白因子;同时结合在PCNA上的蛋白因子之间是否进行相互的变构调节等。
课题组以PCNA作为诱饵蛋白,首先建立了一套从染色质中快速纯化PCNA内源复合物的方法,得到了一系列含有PCNA且参与DNA复制、核小体组装、DNA修复等过程的内源复合物。
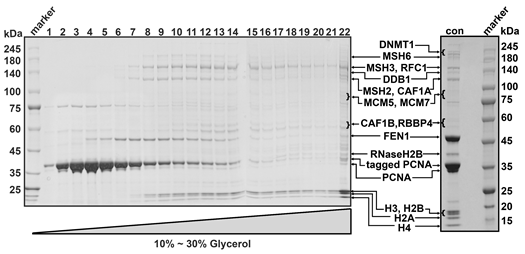
图1 PCNA内源相关复合物的获得
课题组结合冷冻电镜单颗粒三维重构技术解析了两套复合物PCNA-FEN1与PCNA-FEN1-RNaseH2不同状态的三维结构。其中PCNA-FEN1复合物中FEN1处于催化后状态,5'-flap DNA被切割但尚未离开FEN1的酶活中心,表明FEN1的5'-flap DNA产物释放在细胞内是限速步骤。
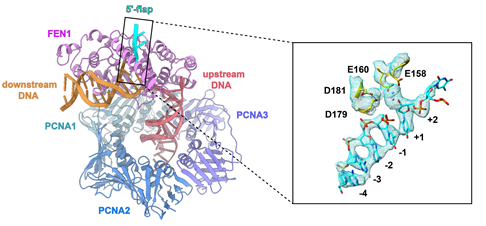
图2 PCNA-FEN1复合物中5'-flap DNA已被酶切
该研究在PCNA-FEN1-RNaseH2复合物中,发现FEN1与RNaseH2分别结合PCNA的不同亚基,证实了PCNA工具带的功能。对不同状态的PCNA-FEN1-RNaseH2复合物进行结构分析,发现RNaseH2始终结合在FEN1催化位点上游固定的双链DNA区域,并且此区域不包含任何未去除的RNA残基,这表明同时结合PCNA的FEN1与RNaseH2存在一种未被认识到的功能上的耦合。因为5'-flap DNA是聚合酶Pol δ继续向前合成进行链置换的产物,其核苷酸序列与其上游双链DNA序列一致,所以在细胞中5'-flap DNA很可能侵入上游的双链DNA,产生3'-flap DNA,一旦3'-flap长度大于1nt,FEN1将无法切割5'-flap DNA。然而,当RNaseH2结合在5'-flap DNA上游双链DNA时,由于空间位阻的存在,5'-flap DNA无法进行链入侵,大幅降低了5'-flap DNA转化形成3'-flap DNA的概率,因此RNaseH2可能通过维持DNA底物的特定构象以促进FEN1 5'-flap DNA的酶切效率。
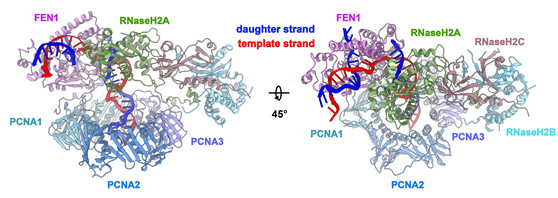
图3 人源PCNA-FEN1-RNase H2复合物结构
此外,结构分析表明在PCNA-FEN1-RNaseH2复合物中RNaseH2蛋白通过RNaseH2A亚基结合PCNA,而不是之前报道的RNaseH2B PIP box结合PCNA,序列分析鉴定到RNaseH2A亚基中一个新的PIP box,通过体外生化实验验证了RNaseH2A及RNaseH2B亚基的PIP box都可以结合PCNA。结合免疫荧光电镜的成像实验,表明RNaseH2A PIP box与RNaseH2B PIP box在不同的生理学过程中发挥功能。
本研究表明PCNA在细胞内可以作为工具带同时结合多个蛋白因子。之前的模型认为同时结合PCNA的蛋白是按照顺序发挥功能,一个酶发挥完功能后与DNA解离,释放出DNA底物以供另一个酶结合;与之前的模型不同,在PCNA-FEN1-RNaseH2复合物结构中,FEN1与RNaseH2稳定结合同一个DNA底物,RNaseH2可能通过对底物DNA的构象调控来促进FEN1的酶切效率。这表明细胞内同时结合PCNA的不同蛋白因子间可能会对它们酶促反应动力学进行互相调控。RNaseH2除了作为RNA核酸酶参与冈崎片段引物RNA的去除外,本研究发现了RNaseH2的另一个可能的新功能,即以不依赖于核酸酶活性的形式作为双链DNA结合蛋白来调节FEN1的5'-flap DNA酶切功能。

图4 RNaseH2在冈崎片段成熟过程中发挥作用的模型
高宁为本论文的通讯作者。生命科学学院2020级博士研究生田宇辉为本论文第一作者。生命科学学院研究员李宁宁在实验方法探究及结构计算方面提供了帮助。生命科学学院教授李晴为该论文提供了支持与帮助。本研究得到了国家自然科学基金和昌平实验室的支持。北京大学冷冻电镜平台、昌平实验室冷冻电镜平台、北京大学高性能计算平台、生命科学学院仪器中心及国家蛋白质基础设施(北大分平台)对本项目提供了重要的技术支持。
参考文献:
1. Bellush, J. M. & Whitehouse, I. DNA replication through a chromatin environment. Philos Trans R Soc Lond B Biol Sci 372 (2017).
2. Li, N. et al. Parental histone transfer caught at the replication fork. Nature 627, 890—897 (2024).
3. Slade, D. Maneuvers on PCNA Rings during DNA Replication and Repair. Genes 9, 416 (2018).
4. Boehm, E. M., Gildenberg, M. S. & Washington, M. T. The Many Roles of PCNA in Eukaryotic DNA Replication. Enzymes 39, 231—254 (2016).
5. Sun, H. et al. Okazaki fragment maturation: DNA flap dynamics for cell proliferation and survival. Trends in Cell Biology(2022).
6. Levikova, M. & Cejka, P. The Saccharomyces cerevisiae Dna2 can function as a sole nuclease in the processing of Okazaki fragments in DNA replication. Nucleic Acids Res 43, 7888—7897 (2015).
7. Blair, K. et al. Mechanism of human Lig1 regulation by PCNA in Okazaki fragment sealing. Nat Commun 13, 7833 (2022).
8. Lancey, C. et al. Structure of the processive human Pol δ holoenzyme. Nature Communications 11 (2020).