
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Cell Genomics︱崔庆华/王国庆团队提出m6A位点预测新型人工智能算法
【字体: 大 中 小 】 时间:2024年11月23日 来源:北京大学基础医学院
编辑推荐:
众多生物学家和医学家利用SRAMP工具成功发现了所研究RNA的m6A位点,其中不乏(30余篇)发表在Science、Nature Cardiovascular Disease、Sci Immunol、Neuron等CNS期刊的论文

N-6-methyladenosine (N6-腺苷酸甲基化,m6A)是目前已知丰度最高的一种RNA化学修饰,具有重要的分子功能,在众多生理与病理生理过程中均发挥了重要作用。因此,研究m6A在健康与疾病中的作用是重要科学问题,但是前提是首先要精准的确定RNA上的m6A修饰位点。为此,2016年,崔庆华课题组利用机器学习建立了完全基于RNA序列信息的m6A位点预测算法SRAMP(www.cuilab.cn/sramp, Nucleic Acids Res. 2016 Jun 2;44(10):e91.)。SRAMP也是国际上第一个哺乳动物m6A位点预测的生物信息学工具,被国际同行评价为该领域性能最好的算法(Top performing),也是该领域被应用最多最广泛的工具(citation = 660)。众多生物学家和医学家利用SRAMP工具成功发现了所研究RNA的m6A位点,其中不乏(30余篇)发表在Science、Nature Cardiovascular Disease、Sci Immunol、Neuron等CNS期刊的论文。

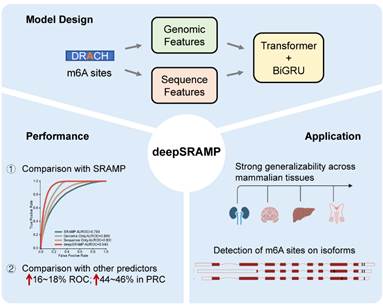
近年随着大量来自实验的m6A位点数据的产生,研发基于深度学习复杂模型的m6A位点预测算法则显得尤为重要且具备了数据基础。为此,近日,崔庆华课题组联合吉林大学基础医学院王国庆课题组在《细胞·基因组学》(Cell Genomics)发表题为“A combined deep learning framework for mammalian m6A site prediction”的研究型文章,提出了集成Transformer神经网络和循环神经网络的组合深度学习算法deepSRAMP。

结果表明,deepSRAMP算法相较于基于机器学习的SRAMP算法性能获得显著提升(AUC和AUPRC分别提升15.0%和30.9%)。相较于近年其他集成序列和基因组位置特征或基于深度学习的算法如WHISTLE和DeepPromise,deepSRAMP也大幅领先(AUC: 0.966 vs. 0.805 and 0.783; AUPRC: 0.814 vs. 0.375, and 0.350)。更重要的是,deepSRAMP算法在不同测序技术、不同组织或细胞系、不同物种来源的m6A修饰位点的预测中均展现出较高的精准性。此外,deepSRAMP还集成多实例学习方法,能够实现基因转录本isoform水平的m6A修饰位点预测,进而为解析m6A修饰的生物学功能提供了理论基础。该研究体现了计算科学、生物医学等多学科交叉融合的优势,为精准预测m6A位点提供了一种高性能的人工智能工具。
原文链接:https://www.cell.com/cell-genomics/fulltext/S2666-979X(24)00326-4
北京大学基础医学院医学生物信息学系博士生樊锐(已于2024年7月毕业)和博士后崔春梅(已于2024年11月出站)为该论文的共同第一作者,崔春梅博士、吉林大学基础医学院王国庆教授、武汉体育学院运动医学院、北京大学医学部崔庆华教授为共同通讯作者。本研究受到国家自然科学基金项目(批准号:62025102、32301239、81921001, U23A20269)的支持。
下载安捷伦电子书《通过细胞代谢揭示新的药物靶点》探索如何通过代谢分析促进您的药物发现研究
10x Genomics新品Visium HD 开启单细胞分辨率的全转录组空间分析!
 生物通微信公众号
生物通微信公众号
知名企业招聘
今日动态 | 人才市场 | 新技术专栏 | 中国科学人 | 云展台 | BioHot | 云讲堂直播 | 会展中心 | 特价专栏 | 技术快讯 | 免费试用
版权所有 生物通
Copyright© eBiotrade.com, All Rights Reserved
联系信箱:
粤ICP备09063491号
