
-

生物通官微
陪你抓住生命科技
跳动的脉搏
赵美萍研究团队在Nature Biomedical Engineering发文报道利用精准的DNA分子剪切工...
【字体: 大 中 小 】 时间:2023年08月29日 来源:北京大学新闻网
编辑推荐:
2023年7月27日,北京大学化学与分子工程学院赵美萍教授课题组在《自然—生物医学工程》(Nature Biomedical Engineering)在线发表了题为“Detection of low-frequency mutations in clinical samples by increasing mutation abundance via the ex...
基因突变的快速准确检测对临床治疗方案选择、疗效评价、疾病伴随诊断及实现精准医疗等都具有至关重要的意义。然而,由于血液或微量组织样本中大量共存的DNA野生链带来的背景干扰,目前广泛使用的方法在检测变异等位基因频率(VAF)低至接近0.01%甚至更低水平的基因突变时,其检出能力明显不足。近年来,尽管新的方法不断涌现,但在可控性、普适性、定量能力、检测时间和成本等方面仍存在较多的局限性。
2023年7月27日,北京大学化学与分子工程学院赵美萍教授课题组在《自然-生物医学工程》(Nature Biomedical Engineering)在线发表了题为“Detection of low-frequency mutations in clinical samples by increasing mutation abundance via the excision of wild-type sequences”的研究论文。该研究发展了一种具有单碱基区分能力的DNA分子剪切工具,能够高度特异性地清除基因组DNA(gDNA)和循环游离DNA(cfDNA)中的高背景野生链,从而使突变位点的VAF大幅度提升。这一新颖、高效且通用的方法为基因突变的高灵敏检测提供了全新的途径。
赵美萍课题组一直致力于研究各种核酸酶与核酸分子之间的相互作用机理。研究团队在早期的研究中发现,虽然DNA分子的骨架被磷硫酰化修饰后完全抵抗脱氧核糖核酸内切酶I(DNase I)的水解作用,但当一条磷硫酰化修饰的DNA单链(sspDNA)与另一条完全互补的正常DNA单链杂交后,DNase I仍然可以快速水解该双链中的正常DNA单链。通过将sspDNA与DNase I进行预组装,他们得到的蛋白-sspDNA复合物在对正常DNA进行水解时表现了序列选择性。对与sspDNA序列完全互补的底物DNA序列可以快速水解,而对存在错配或完全无关的DNA序列,水解速率显著下降(Chemical Science 2016, 7, 2051)。
研究团队通过进一步探索sspDNA与DNase I之间的相互作用特点,揭示出sspDNA因骨架上硫原子的荷电能力和疏水性与氧原子的差异,使其与DNase I的亲和力明显高于正常的DNA链。基于这一特性,他们巧妙地构建了一种精准的DNA分子剪切工具(sgDNase)。通过竞争性结合和水解机制,sgDNase以sspDNA作为惰性模板链,在DNA样本中实时引导DNase I对野生型DNA链(WT)进行特异性水解,而突变链(MT)由于存在碱基序列的差异而得以保留(图1a)。实验结果表明,sgDNase对sspDNA覆盖范围内所有类型的突变均具备单碱基分辨能力(图1b—d)。
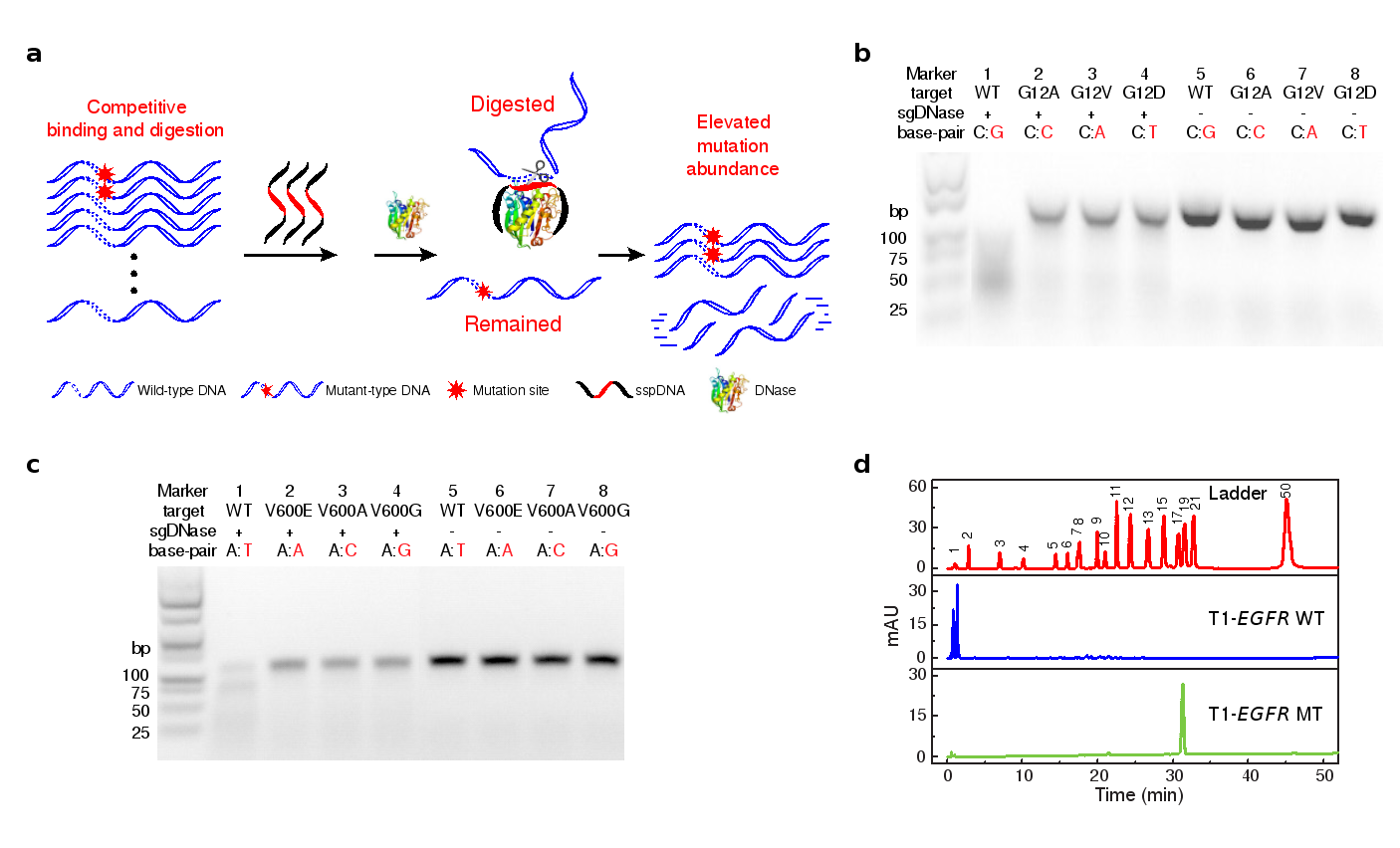
图1. (a) sgDNase对DNA分子进行序列特异性剪切的原理示意图;(b—d) sgDNase的单碱基区分能力展示:(b)KRAS G12野生链和不同突变链(KRAS G12 WT/G12A/G12V/G12D)经sgDNase水解所得产物的凝胶电泳图;(c) BRAF V600 野生链和不同突变链(BRAF V600 WT/V600E/V600A/V600G)经sgDNase水解所得产物的凝胶电泳图;(d)EGFR L858R 野生链(T1-EGFR WT)和突变链(T1-EGFR MT)经sgDNase水解产物的HPLC色谱图
在sgDNase这一DNA分子剪切工具的作用下,低丰度基因突变的样本转变为高丰度基因突变的样本,而后者可以轻松地通过各种常规突变检测方法,如Sanger测序法、二代测序法(NGS)、数字液滴PCR法(ddPCR)或荧光探针法等检出。在图2a中,经过sgDNase处理后,样本中EGFR L858R突变的VAF从0.01%提升至22%,通过Sanger测序法即可迅速检出。将sgDNase体系拓展至18种常见的热点突变,这些基因突变即使初始VAF低至0.01%,在经过sgDNase处理后,也可通过Sanger测序法(图2b)或NGS(图2c)快速检出。该方法可用于多位点基因突变的同时富集,与NGS联用,目前可实现至少300个不同位点的同时高灵敏检出。
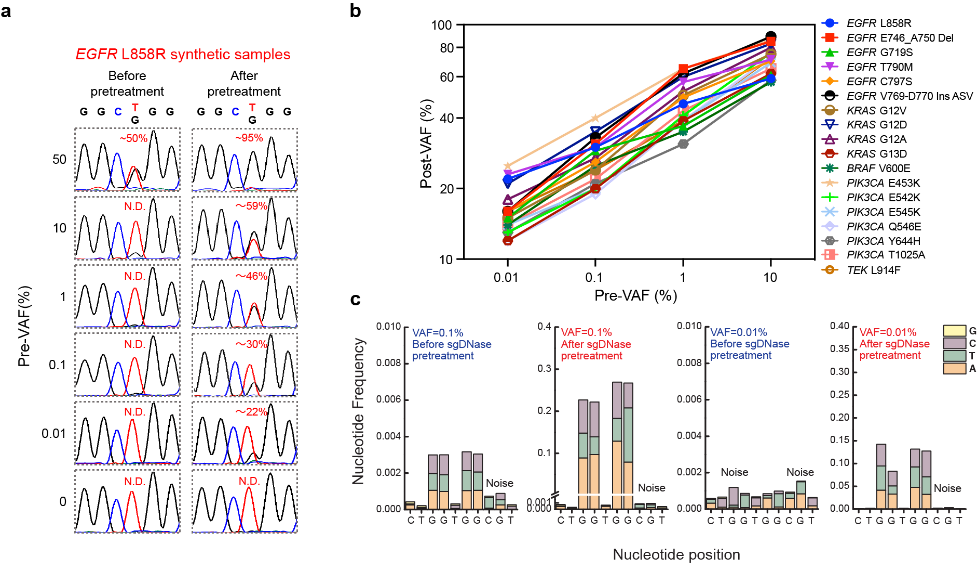
图2. (a)sgDNase处理前后,样本中EGFR L858R突变的VAF变化对比(Sanger测序结果);(b)18种常见的热点突变在经过sgDNase处理后VAF的变化对比;(c) sgDNase处理前后,样本中KRAS G12G13突变的VAF变化对比(NGS测序结果)
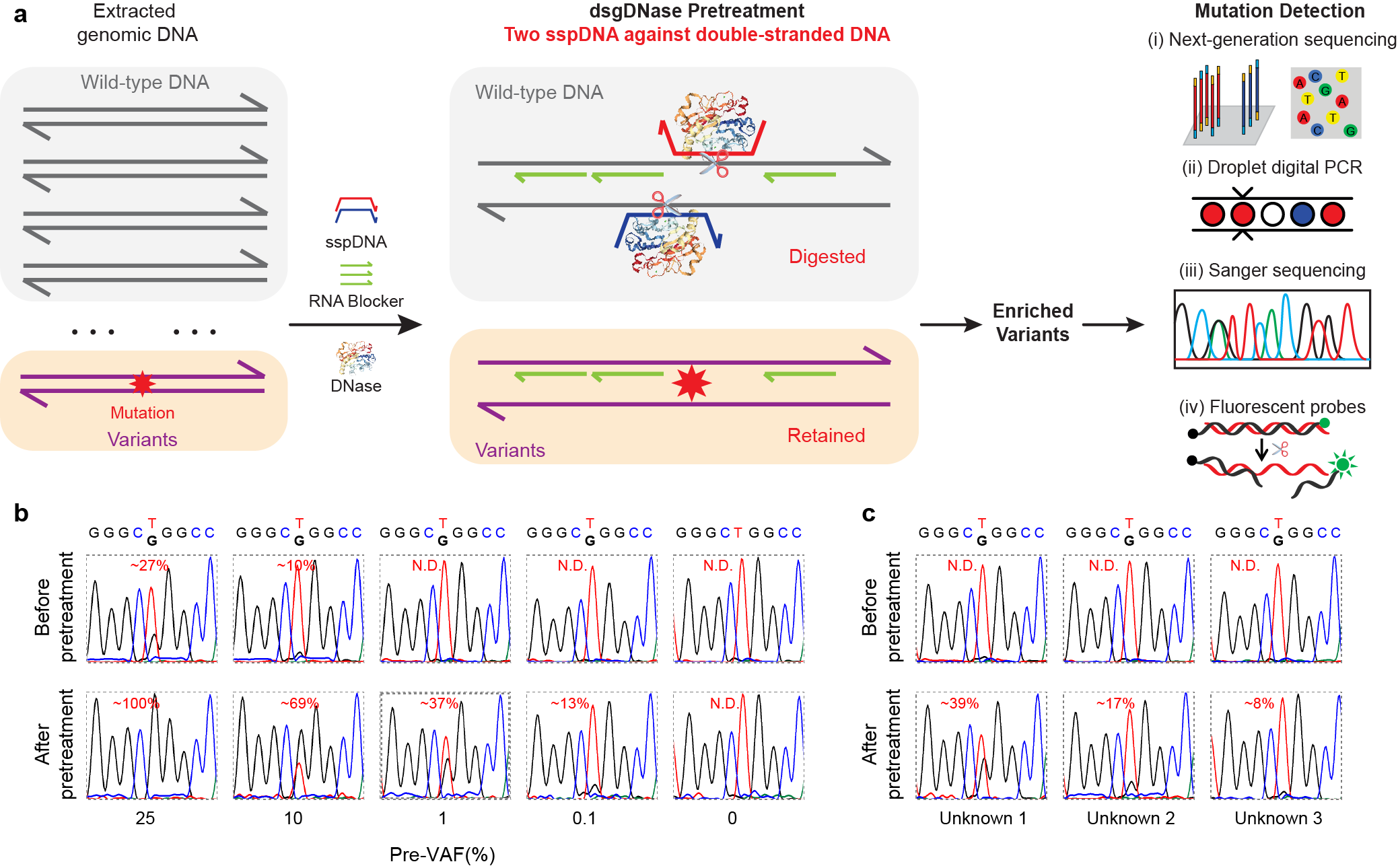
图3. (a)双sspDNA引导的sgDNase用于基因组DNA中低频突变的富集。(b) 不同起始VAF的EGFR L858R突变经双sspDNA引导的sgDNase处理后的VAF测定结果。(c)三个未知样品经双sspDNA引导的sgDNase处理后EGFR L858R突变的VAF测定结果
研究团队进一步设计了成对的sspDNA,用于对基因组双链DNA的直接剪切处理(图3)。初始VAFs分别为1%~10%、0.1%~1%和<0.1%的三个未知样品经过双sspDNA引导的sgDNase处理后,通过Sanger测序测得VAFs分别为39%、17%和8%。测定结果得到了ddPCR法的确证。
sgDNase分子剪切工具在临床样本的基因突变检测中展现出了良好的应用效果(图4)。例如:在用于非小细胞肺癌(NSCLC)和结直肠癌患者的血液样本和组织样本中EGFR L858R突变的检测时,直接测序只能测出两例组织样本为阳性,血液样本都未测到阳性。经过sgDNase处理后,有两例血液样本和另一例组织样本被检出为阳性。这些阳性结果得到了NGS 深度测序结果的确证。对结直肠癌患者的样本进行KRAS G12/G13 突变检测时,经过sgDNase处理后,不仅有效提升了在血液样本中检出的能力,而且帮助测出KRAS G13D+G13S双阳性的样本。在静脉畸形患者样本的检测中,经sgDNase处理后,有多例阳性样本被检出。
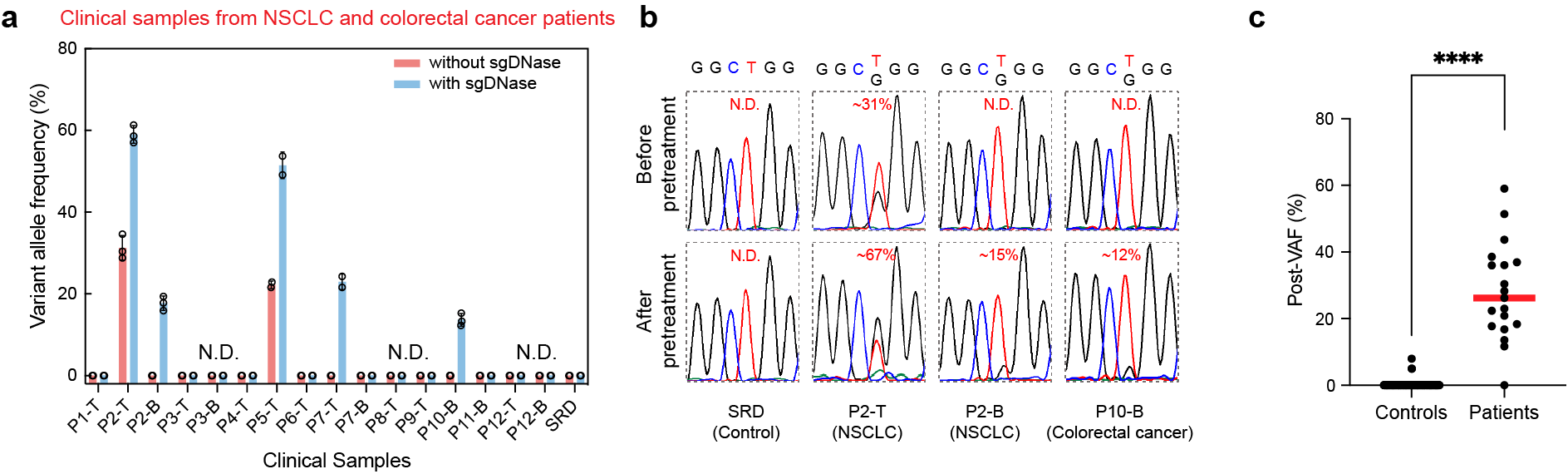
图4. (a)sgDNase用于8位NSCLC 患者(P1—P8)和4位结直肠癌患者(P9—P12)血液样本(B)和组织样本(T)中EGFR L858R突变检测的结果。(b)sgDNase处理前后部分样本中EGFR L858R 突变的测序结果。(c)sgDNase处理后癌症患者与阴性对照样本的VAFs 对比
作为一种新颖的序列特异性DNA分子剪切工具,sgDNase克服了同类方法存在的反应温度条件苛刻、序列设计复杂、定量能力差、辅助设备/试剂昂贵以及普适性不佳等问题,不仅剪切过程反应温和、序列通用性好,而且可达到单碱基分辨的水平。sgDNase与Sanger测序法联用可实现VAF低至0.01%—0.1%的基因突变的快速低成本筛查;与ddPCR法联用可实现VAF低至0.1ppm的基因突变的超灵敏快速定量检测;与常规测序深度的NGS法联用可实现多个突变位点的高通量高灵敏多重检测。sgDNase既可直接用于提升临床检验中肿瘤早筛、复发监测和微小残留检测等的检出能力,也可灵活设计,进一步拓展用于其它基因损伤分析或外源基因的追踪等,在基因检测相关领域有广泛的应用前景。
北京大学化学与分子工程学院已毕业博士生陈维(2011本/2015博)、已毕业本科生徐海齐(2016级)和在读博士生戴沈镔(2017本/2021博)为该工作的共同第一作者,赵美萍、华中科技大学同济医学院肖先金教授(2016年博士毕业于北大化学与分子工程学院)和吴曈勃教授(2017年博士毕业于北大化学与分子工程学院)为共同通讯作者。北京大学第一医院皮肤科林志淼教授和首都医科大学附属北京儿童医院(国家儿童医学中心)张斌博士对研究工作给予了大力支持。该项目得到国家自然科学基金委、北京分子科学国家研究中心的资助与支持。