
-

生物通官微
陪你抓住生命科技
跳动的脉搏
清华大学Nature发文:染色质去乙酰化动态修饰调控新突破
【字体: 大 中 小 】 时间:2023年07月21日 来源:清华大学
编辑推荐:
研究者们通过化学生物学手段构建复合修饰核小体,利用单颗粒冷冻电镜技术解析了酿酒酵母Rpd3S复合物在自由状态和H3K36me3核小体结合状态下的分子结构模型,并结合去乙酰化酶活分析以及酵母遗传学等功能实验,系统全面的对Rpd3S复合物的组装模式、底物识别催化和修饰介导调控等过程进行了分子机制解剖,揭示出甲基化指引的染色质去乙酰化动态和多样化模型。
2023年7月19日,Nature杂志在线发表生命中心李海涛课题组携手生命中心闫创业课题组、上海交通大学李兵课题组合作完成的题为“ Diverse modes of H3K36me3-guided nucleosomal deacetylation by Rpd3S ”(H3K36me3指引下的多模式Rpd3S核小体去乙酰化)的研究长文。研究者们通过化学生物学手段构建复合修饰核小体,利用单颗粒冷冻电镜技术解析了酿酒酵母Rpd3S复合物在自由状态和H3K36me3核小体结合状态下的分子结构模型,并结合去乙酰化酶活分析以及酵母遗传学等功能实验,系统全面的对Rpd3S复合物的组装模式、底物识别催化和修饰介导调控等过程进行了分子机制解剖,揭示出甲基化指引的染色质去乙酰化动态和多样化模型。表观遗传调控是确保生物表型复杂性和多样性的关键机制,本研究凸显了表观遗传调控的精妙复杂性,完美展示出大自然通过形成多亚基大复合物来实现高级调控能力的精巧分子设计。

依赖于上下游修饰的动态组蛋白修饰调控是基因转录过程中的重要表观遗传机制。例如,组蛋白H3第4位赖氨酸的三甲基化修饰(H3K4me3)和H3第36位赖氨酸的三甲基化修饰(H3K36me3)是区分转录起始位点和转录编码区域的标志【1, 2】。在酵母中,组蛋白甲基转移酶Set2可以被RNA聚合酶II直接募集,并特异性甲基化H3K36位点。组蛋白去乙酰化酶(HDAC)Rpd3S复合物在转录编码区域被H3K36me3修饰所招募,并通过其去乙酰化酶活性抑制异常启动子的转录【3, 4】。因此,Set2-Rpd3S调控通路介导了H3K36甲基化和组蛋白去乙酰化的偶联从而维持基因的正常转录延伸并防止异常转录发生。
Rpd3最初被认为是一种全局基因调控因子和共抑制因子,1996年首次报道其作为HDAC发挥作用【5, 6】。这一发现,与同年组蛋白乙酰转移酶GCN5转录共激活功能的突破性发现,奠定了现代表观遗传学新范式。在酵母中,Rpd3与Sin3、Ume1、Rco1和Eaf3进行组装形成0.6-MDa的Rpd3S复合物,通过Eaf3亚基的CHD结构域识别H3K36me3和Rco1的PHD结构域识别未修饰的组蛋白H3 N-端尾部(H3K4un)来定位与调控【7-9】。然而长久以来,Rpd3S复合物催化修饰核小体底物的分子识别模式及其去除组蛋白H3、H4各位点乙酰化修饰的特异性催化机理都缺乏深入的研究。
李海涛合作团队通过样品制备条件的优化,利用冷冻电子显微镜技术(cryo-EM)解析出Rpd3S复合物的3.2 Å分辨率的电镜结构。研究成果展示出Rpd3S复合物的独特分子结构,其中双拷贝的Eaf3和Rco1蛋白形成异源二聚体与Rpd3和Sin3不对称地结合形成一个催化核心复合物,而核心催化酶Rpd3受到其他亚基共同的复杂调控。同时两分子Eaf3-Rco1异源二聚体呈现出不对称组装方式,类似于两个手臂介导Rpd3S复合物的酶活催化。与此同时,李海涛研究团队结合复合物分子模型和ITC等生化技术重新定义了Rco1两个PHD结构域的识别互作机理:PHD1参与识别H3K4un并介导复合物的催化与定位;PHD2主要作为组装模块确保复合物组装的稳定性,但缺失组蛋白修饰的识别能力(图1)。
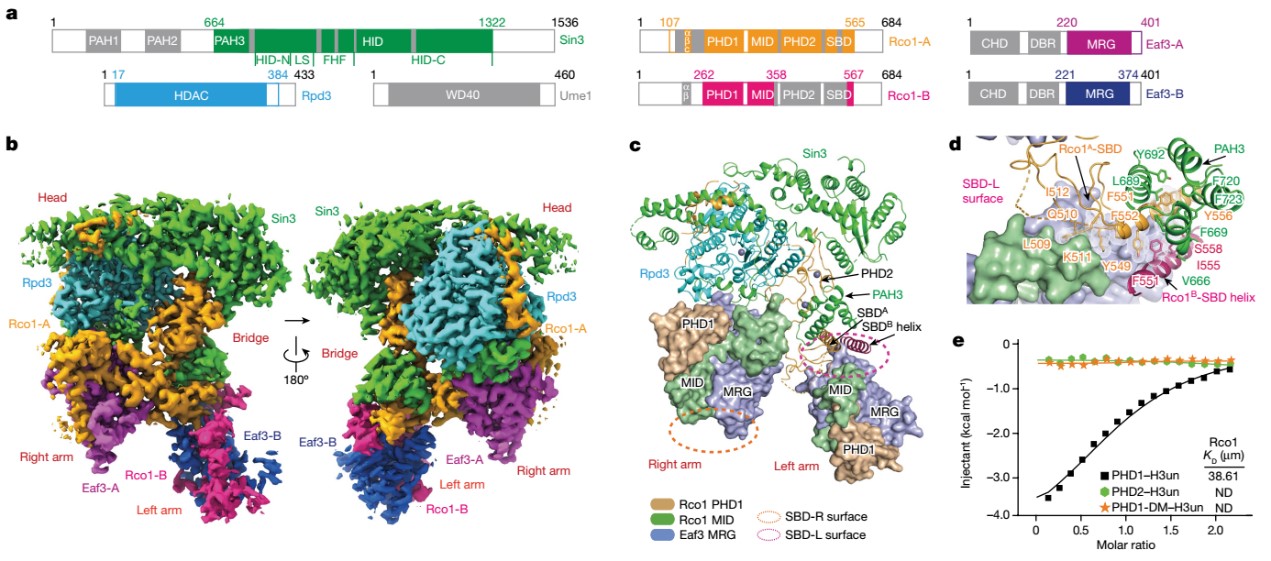
图1. Rpd3S复合物的整体结构以及亚基间的相互作用与调控
随后李海涛研究团队利用修饰多肽分段合成和化学连接方式合成了多种修饰组蛋白,包括H3K36me3、H3K9acK14acK18acK23acK27ac、H3K9acK14acK18acK23acK27acK36me3、H3K4me3K9acK14acK18acK23acK27acK36me3、H3K9RK14acK18acK23acK27acK36me3以及H4K5acK8acK12acK16ac等修饰,并构建出复合修饰核小体底物以用于后续结构生物学和生化酶活研究。
依托人工合成的修饰核小体以及进一步优化样品制备条件,李海涛合作团队成功解析出两种状态的Rpd3S- H3K36me3修饰核小体的结合催化复合物电镜结构,分别命名为“close”状态和“loose”状态,体现出Rpd3S复合物在去乙酰化催化过程中的动态性。其中至关重要的是,两个Eaf3蛋白的CHD结构域分别识别在一个核小体的两个H3K36me3修饰上面,提示染色质双H3K36me3修饰状态的建立与维持是Rpd3S复合物被招募进而行使去乙酰化过程的关键条件(图2)。
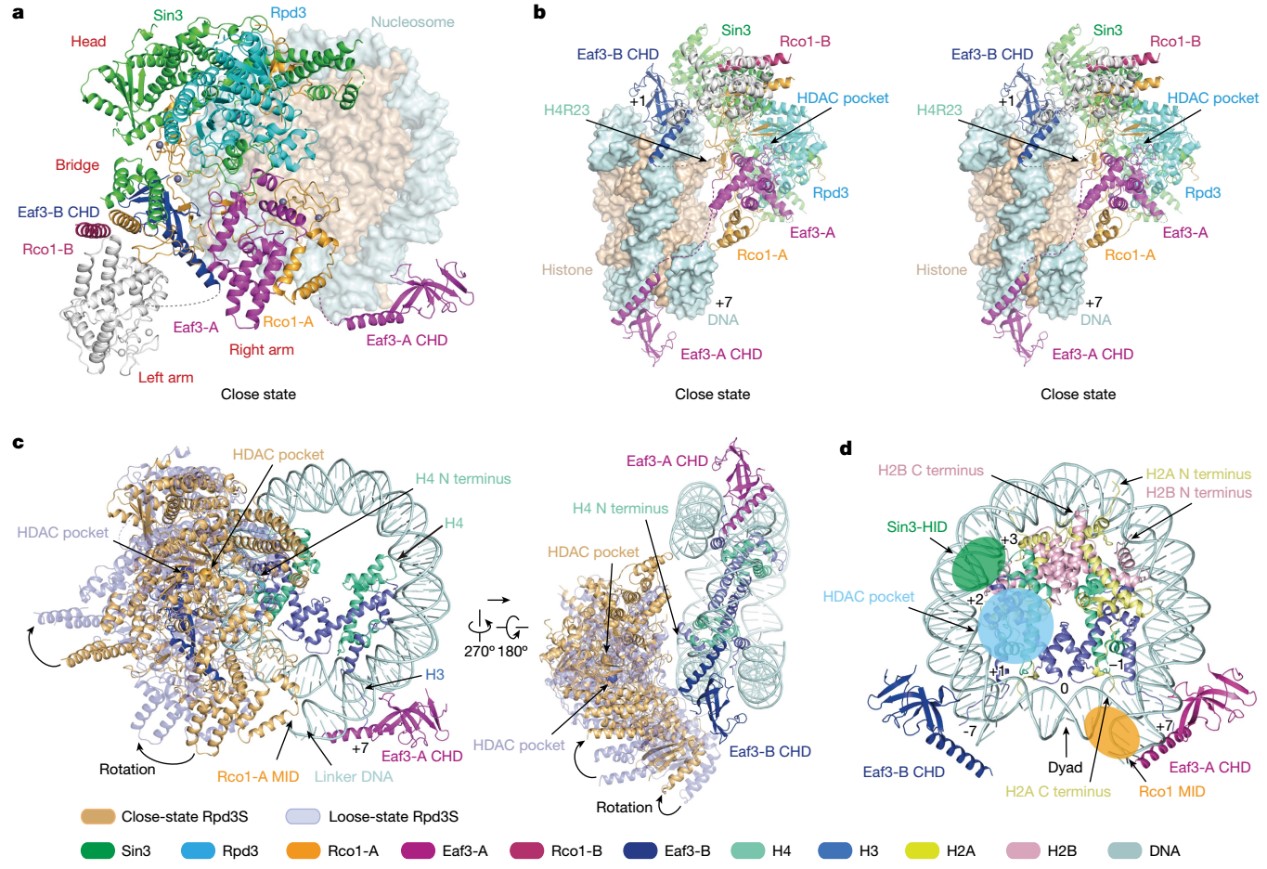
图2. Rpd3S与H3K36me3修饰核小体的互作催化复合物结构
与此同时,复合物结构中,Eaf3、Sin3和Rco1蛋白通过建立与双H3K36me3修饰、核小体DNA以及Linker DNA的多价识别,将Rpd3的催化中心定位在组蛋白H4 N-末端尾部附近进行去乙酰化酶活,这揭示了Rpd3S复合物去除H4 N端乙酰化的分子机理(图3)。
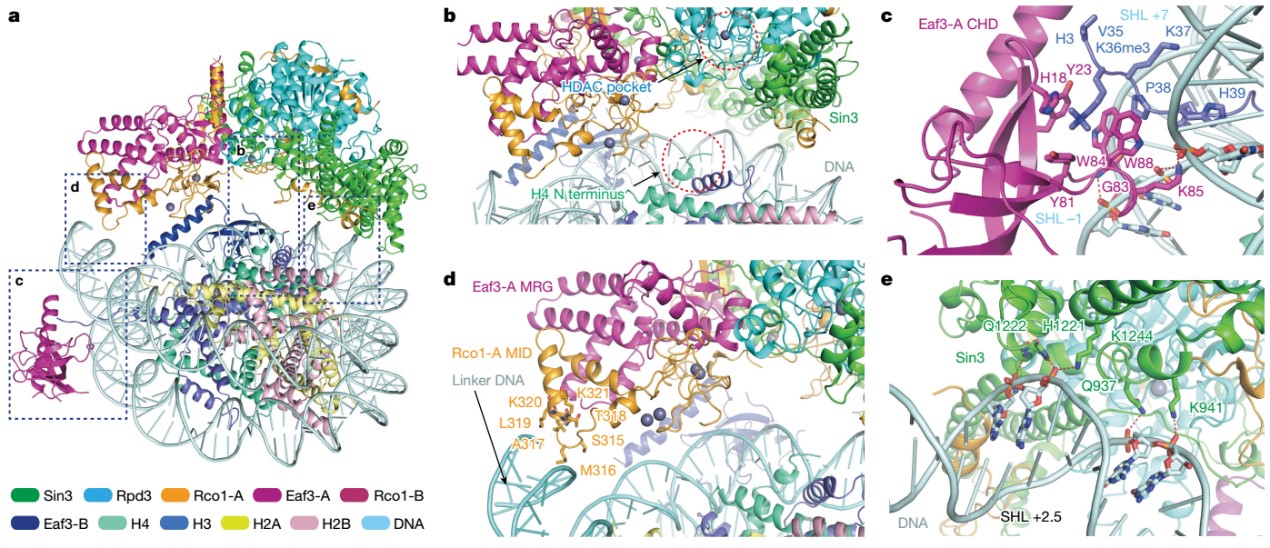
图3. Rpd3S与H3K36me3修饰核小体间的分子互作细节
为进一步探究Rpd3S复合物在多价态结合介导下对修饰核小体去乙酰化的特异性分子机制,李海涛团队合成H4(K5acK8acK12acK16ac),H3(K9acK14acK18acK23acK2 7acK36me3)的修饰组蛋白,并重组多类型修饰核小体作为底物参与酶活鉴定与分析发现。Rpd3S复合物对H3K14ac、H3K23ac以及H4K5ac和H4K12ac具有优先催化的能力。研究同时发现Rpd3S在高效去除H3和H4上的其他各位点的乙酰化修饰的同时,却特异性保留了H3K9位乙酰化修饰(H3K9ac),且加长酶活反应时间并不影响H3K9ac的保留。通过薛定谔软件将H31-16K14ac修饰多肽摆放入Rpd3S结构内部可见,H3K9ac的保留是由于PHD1对于H3K4un的识别从而使H3K9位点远离Rpd3催化中心所致。为进一步探究Rpd3S复合物去除H3 N端乙酰化的分子机理,研究者设计了PHD1结构域的突变,酶活结果证实了PHD1仅参与去除H3 N端乙酰化的分子机理。除此之外,通过特定设计的SBD结构域的突变(7mu)影响了左臂区域包括Eaf3B-CHD结构域的稳定性,酶活实验结果进一步证实了左臂区域对去除H3和H4 N端乙酰化的重要性,尤其针对于H4(图4)。
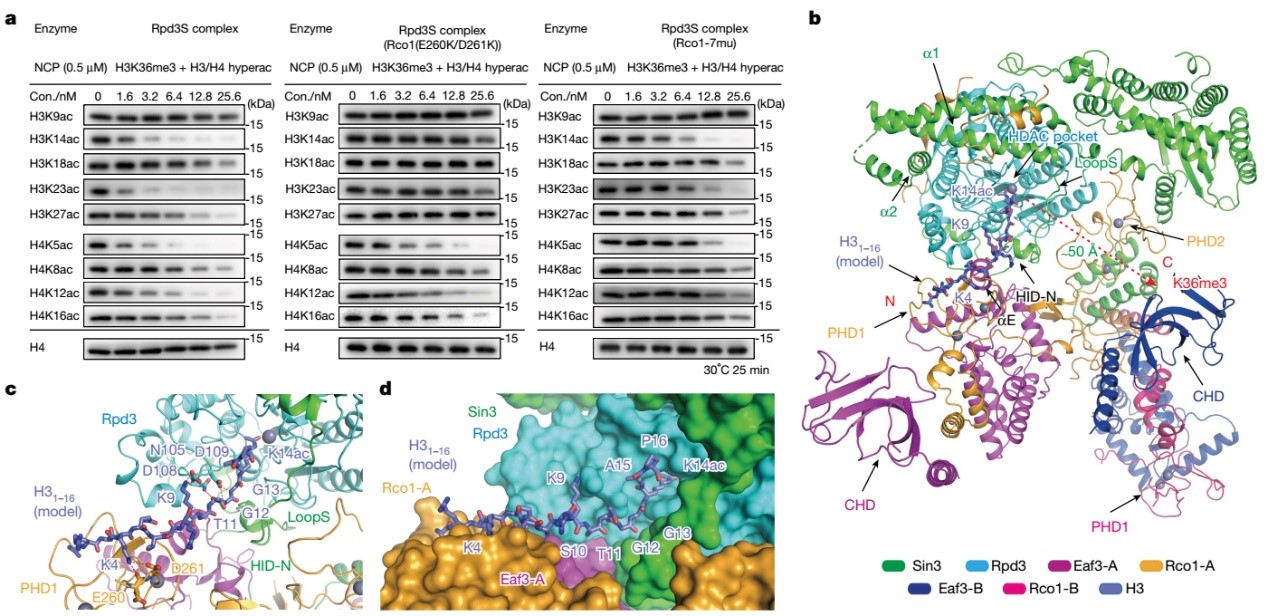
图4. Rpd3S复合物识别结构域间协同调控去乙酰化活性
然而复合物体外酶活实验并不能直接反应复合物体内的调控模式,故李海涛课题组研究人员进一步利用STE11-HIS3酵母报告系统去探究野生型Rpd3S和突变株在酵母表型上的影响。PHD1和SBD结构域的突变都能导致酵母异常转录的发生,且酵母整体的组蛋白H3和H4的乙酰化水平都不同程度上受到了影响,且与体外突变体的酶活实验结果完美吻合。综上,研究人员基于分子结构、生化实验和功能研究结果,提出了Rpd3S复合物去乙酰化的动态调控模型:Rpd3S在单个核小体上利用双分子的CHD结构域识别双组蛋白N端H3K36me3修饰从而将Rpd3的催化中心定位在组蛋白H4 N端尾部附近,从而实现H4高效的去乙酰化。另一种调控模式下,在一个组蛋白H3尾部附近,Rpd3S复合物通过Rco1-A 的PHD1结构域对H3K4un的识别和Eaf3-B CHD结构域对H3K36me3的识别协同实现H3高效的去乙酰化(图5)。
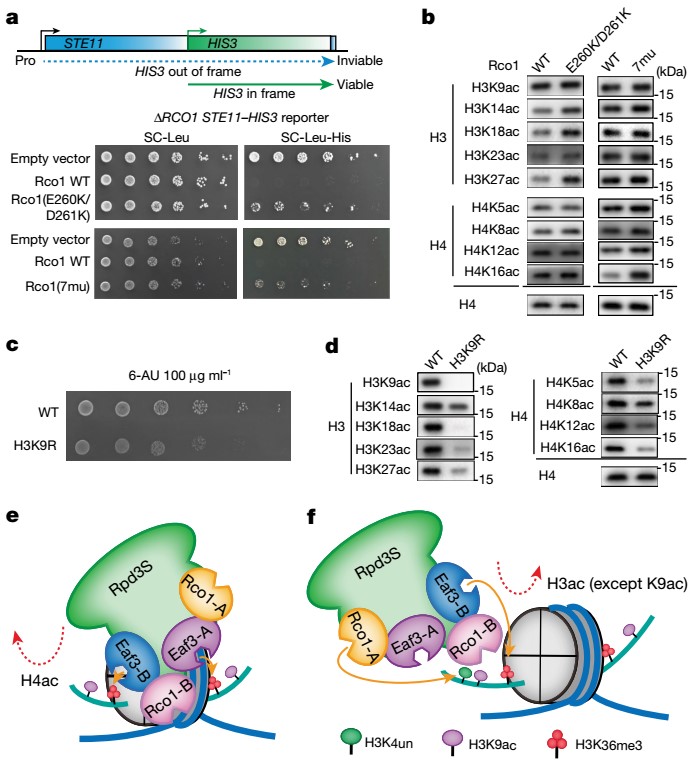
图5. Rpd3S复合物体内功能研究和催化模型
由于对去乙酰化过程中H3K9ac修饰被特异性保留的意外发现,李海涛研究团队通过引入体内H3K9位点突变(H3K9R)以影响H3K9ac乙酰化的建立,可以看到酵母体现出明显的生长抑制性表型。对H3和H4的整体乙酰化修饰水平进行进一步系统研究,研究者发现H3K9ac修饰的缺失进一步影响了近乎H3和H4所有其他位点乙酰化水平的建立,这暗示H3K9ac的存在对后续其他位点的乙酰化建立至关重要。鉴于乙酰化转移酶NuA3和NuA4都存在H3K9ac和H3K36me3的识别结构域,研究人员进一步提出了修饰重建模型:Rpd3S催化反应后,被保留的H3K9ac修饰连同H3K36me3修饰可以作为NuA3-NuA4招募和高乙酰化H3和H4重建的“种子”标记。Rpd3S和NuA3-NuA4作为“eraser–writer”反作用调控酶组合,在转录过程中动态调控基因表达(图6)。
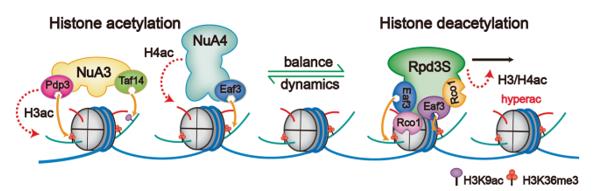
图6. “Rpd3S-NuA3/NuA4”在转录过程中动态调控染色质乙酰化水平模型
在此项研究中,清华大学医学院李海涛课题组博士后管海鹏和已毕业博士生王沛为并列第一作者。清华大学医学院2020级博士生张沛主要参与了本研究,上海交通大学医学院李兵教授、阮纯博士,以及郑州大学医学科学院郑向东特聘教授提供了专业指导和帮助。生命中心PI李海涛和闫创业为共同通讯作者。该工作得到了生命科学联合中心、国家自然科学基金委、国家重点研发计划的资助。
原文链接:
https://www.nature.com/articles/s41586-023-06349-1
参考文献:
1. Krogan, N. J. et al. Methylation of histone H3 by Set2 in Saccharomyces cerevisiae is linked to transcriptional elongation by RNA polymerase II. Mol Cell Biol 23, 4207-4218, doi:10.1128/mcb.23.12.4207-4218.2003 (2003).
2. Kizer, K. O. et al. A novel domain in Set2 mediates RNA polymerase II interaction and couples histone H3 K36 methylation with transcript elongation. Mol Cell Biol 25, 3305-3316, doi:10.1128/MCB.25.8.3305-3316.2005 (2005).
3. Li, B. et al. Infrequently transcribed long genes depend on the Set2/Rpd3S pathway for accurate transcription. Genes Dev 21, 1422-1430, doi:10.1101/gad.1539307 (2007).
4. Carrozza, M. J. et al. Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription. Cell 123, 581-592, doi:10.1016/j.cell.2005.10.023 (2005).
5. Vidal, M. & Gaber, R. F. RPD3 encodes a second factor required to achieve maximum positive and negative transcriptional states in Saccharomyces cerevisiae. Mol Cell Biol 11, 6317-6327, doi:10.1128/mcb.11.12.6317-6327.1991 (1991).
6. Taunton, J., Hassig, C. A. & Schreiber, S. L. A mammalian histone deacetylase related to the yeast transcriptional regulator Rpd3p. Science 272, 408-411 (1996).
7. Li, B. et al. Combined action of PHD and chromo domains directs the Rpd3S HDAC to transcribed chromatin. Science 316, 1050-1054, doi:10.1126/science.1139004 (2007).
8. Ruan, C., Lee, C. H., Cui, H., Li, S. & Li, B. Nucleosome contact triggers conformational changes of Rpd3S driving high-affinity H3K36me nucleosome engagement. Cell Rep 10, 204-215, doi:10.1016/j.celrep.2014.12.027 (2015).
9. Ruan, C., Cui, H. C., Lee, C. H., Li, S. & Li, B. Homodimeric PHD Domain-containing Rco1 Subunit Constitutes a Critical Interaction Hub within the Rpd3S Histone Deacetylase Complex. J Biol Chem 291, 5428-5438 (2016).