
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基因组所王怡雯课题组开发新型批次效应去除方法
【字体: 大 中 小 】 时间:2023年07月21日 来源:中国农科院基因组所
编辑推荐:
近日,《生物信息学简报(Briefings in Bioinformatics)》 在线发表了基因组所王怡雯课题组联合墨尔本大学的研究论文,题为“PLSDA-batch:a multivariate framework to correct for batch effects in microbiome data(PLSDA-batch:校正微生物组数据中批次效应的多变量框架)”
近日,《生物信息学简报(Briefings in Bioinformatics)》 在线发表了基因组所王怡雯课题组联合墨尔本大学的研究论文,题为“PLSDA-batch:a multivariate framework to correct for batch effects in microbiome data(PLSDA-batch:校正微生物组数据中批次效应的多变量框架)”。该研究开发了一种基于偏最小二乘判别分析(PLSDA)的多元非参数批次效应去除方法:PLSDA-batch,该方法可用于校正微生物组数据中由非实验因素导致的结果差异(批次效应)。

研究微生物组成和表型(包括人类疾病)之间的联系是微生物组研究的主要目标,例如,肠道微生物群落的破坏与多种疾病和亚健康状态有关,从炎症性肠病、糖尿病、到肥胖和营养不良。然而,由于微生物群落是高度动态的,因此微生物组数据极易受到批次效应的影响,批次效应通常掩盖了研究人员所感兴趣的生物学效应。现有的批次效应校正方法主要是为基因表达量数据开发的,没有考虑到微生物组数据的固有特征,包括零膨胀、过度离散和变量之间的相关性。即使存在一些针对微生物组数据的去除批次效应的方法,也都存在许多使用场景和条件上的限制。
因此,研究人员开发了一种以多元方式去除批次效应,同时保留实验组差异的方法,称之为PLSDA-batch(下载地址:https://github.com/EvaYiwenWang/PLSDAbatch)。在此基础上,还提出了两种延伸方法:(1)加权 PLSDA-batch(wPLSDA-batch)应对不平衡的实验设计,(2)稀疏PLSDA-batch(sPLSDA-batch)应对模型过拟合现象。同时在模拟数据集和实际数据集上验证了其与目前几种常用的方法removeBatchEffect、ComBat和SVA相比,具有很强的竞争力,特别是在去除设计不平衡的实验中的批次效应上。

图1 | PLSDA-batch的框架
为了验证方法的有效性,研究人员先在模拟数据集上测试了PLSDA-batch,并与常用的removeBatchEffect、ComBat和SVA这三种方法进行了比较;然后在三个实际数据集上,与removeBatchEffect、ComBat这两种方法进行了对比。
通过模拟平衡和不平衡的实验数据,使用pRDA、R(2)、准确性指数等评估手段,与非加权的PLSDA-batch相比,加权的版本对于不平衡的实验设计是必要的。虽然PLSDA-batch与其他批次效应校正方法相比,保留的实验方差比例相似或略小,但在实验设计不平衡的情况下,PLSDA-batch获得了更高的F1得分和AUC。当模拟数据中没有同时具有实验效应和批次效应的变量时,sPLSDA-batch和PLSDA-batch校正后的数据接近基准数据。然而,当模拟数据含有同时包括这两种效应的变量时,sPLSDA-batch的效果略差于PLSDA-batch。此外,研究人员的结果还表明,SVA有过拟合数据的倾向,而ComBat无法完全消除批次差异,removeBatchEffect无法保留足够的实验效应以准确识别变量。
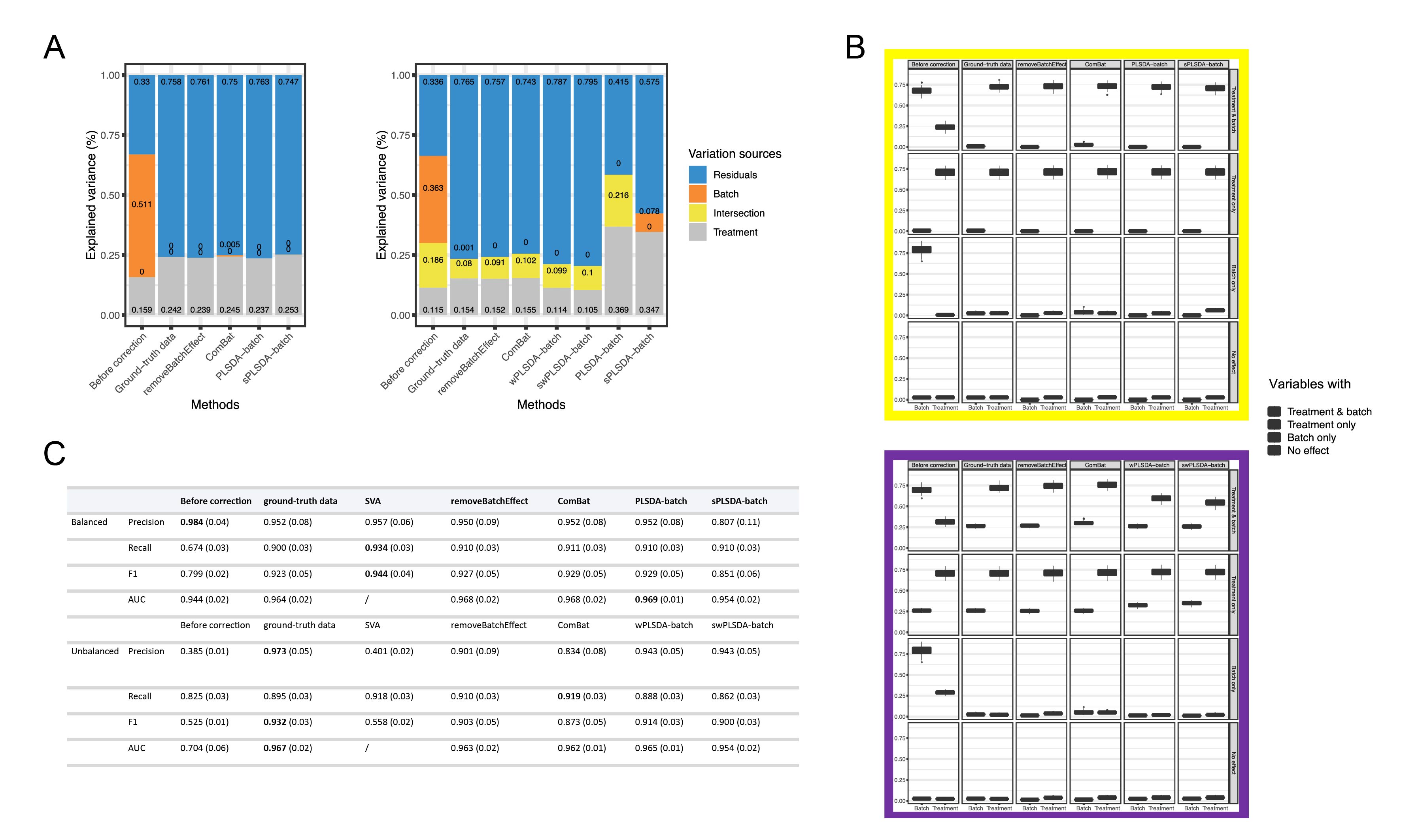
图2 | 模拟数据中PLSDA-batch与其他方法的各项检测指标结果
研究分析了三个采用16s rRNA测序获得的微生物组数据集(OTU级别),分别为海绵组织(sponge,sponge A. aerophoba)数据、厌氧消化(AD,Anaerobic Digestion)数据、高脂肪高糖饮食(HFHS,High Fat High Sugar diet)数据。在分析之前,计数数据均通过了中心对数比转换。在sponge和AD数据中,PLSDA-batch和sPLSDA-batch的表现相似。这两个数据集都具有较强的批次效应,研究人员使用的所有测试指标都表明PLSDA-batch和sPLSDA-batch优于ComBat,后者没有充分地去除批次差异(sponge和AD数据中),且没有充分地保留实验组差异(AD数据中)。并且与本方法相比,使用removeBatchEffect校正的数据能保留的实验方差比例普遍较小。此外,该方法,对于批次效应较弱的HFHS数据,相较于PLSDA-batch,用sPLSDA-batch校正后的数据丢失了少量的实验方差,因为它为了避免过度拟合,估算实验组分时精度没有那么高,表明sPLSDA-batch并不适合弱批次效应。
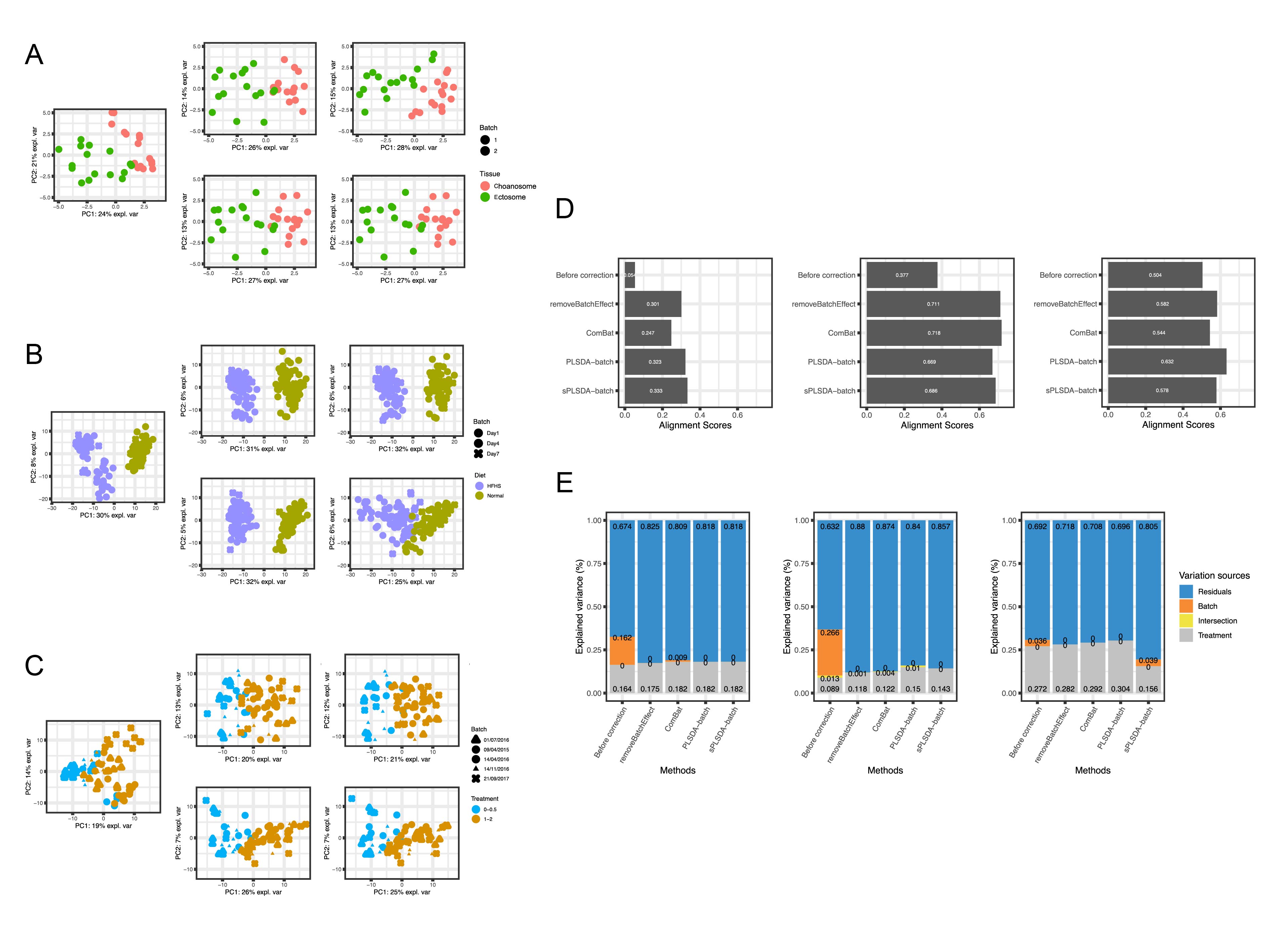
图3 | 实际数据中PLSDA-batch与其他方法的各项检测指标结果
基因组所副研究员王怡雯为该论文的第一作者,墨尔本大学数学与统计学院Kim-Anh Lê Cao教授为通讯作者。
该研究得到了中国国家留学基金委,中国博士后科学基金,中国国家自然科学基金的资助。
原文链接:https://academic.oup.com/bib/article/24/2/bbac622/6991121