编辑推荐:
相关研究论文以“Sequence-based drug design as a concept in computational drug design”为题于2023年7月15日在Nature Communications在线发表
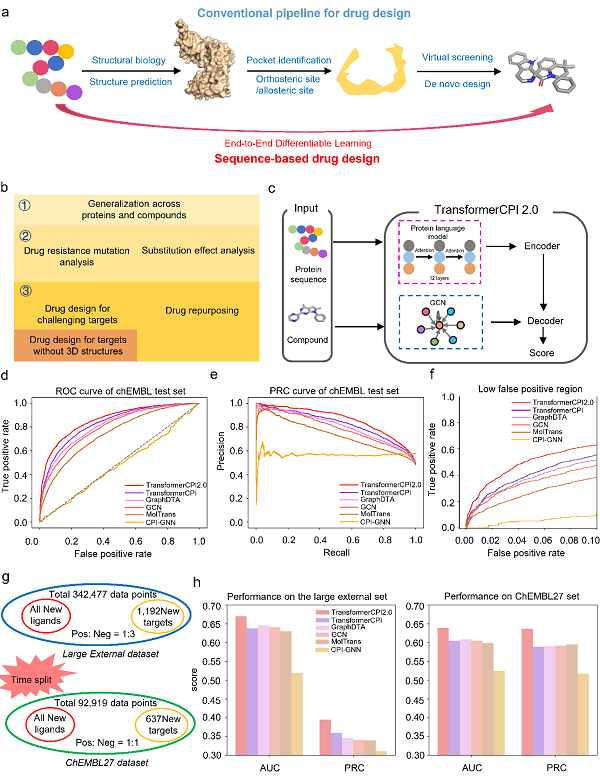
自上世纪90年代以来,基于蛋白质结构的药物 设计(SBDD)一直是创新药物发现的主流方法 ,在针对具有明确靶标的疾病治疗方面取得了重大进步。这种方法一般涉及多个步骤的复杂流程,包括建立蛋白质的三维(3D)结构,识别潜在的配体结合位点,并通过虚拟筛选或全新设计发现活性化合物等等。SBDD流程中的每个步骤都有其局限性,例如许多蛋白质并没有高分辨率的结构。虽然近年来的蛋白质结构预测技术如AlphaFold和RoseTTAFold有所突破,但并非所有预测的结构都适合进行基于结构的药物设计 。精确预测配体结合口袋是一个挑战,因为这些局部结构往往违反“蛋白质折叠规则”,配体的结合也会诱导氨基酸构象发生变化。此外,由于变构效应的机制多样性,对具有多个结构域的新靶标如何确定配体结合位点也存在困难。最后,虚拟筛选可能会产生假阳性,误差和错误的不断累积会导致最后的严重的假阳性问题。
为了解决这些问题,中国科学院上海 药物研究所郑明月团队提出了一种基于序列的药物设计新概念。这种“从序列到药物”(sequence-to-drug)的思路采用端到端的可微学习,直接从蛋白质序列出发进行药物设计发现,而无需经过上述多个中间步骤的复杂流程(图1)。端到端的可微深度学习已经在计算机视觉和自然语言处理等领域产生了革命性的影响,它用可微基元(differentiable primitives)取代了复杂流程的所有组件,从输入端到输出端进行联合优化。AlphaFold2在蛋白质结构预测的成功也很大程度上依赖于端到端可微学习的思想。这个概念具有吸引力地方在于它可以以一种自洽和数据高效的方式执行整个学习建模过程,可能避免了复杂流程中的错误积累。相关研究论文以“Sequence-based drug design as a concept in computational drug design”为题于2023年7月15日在Nature Communications 在线发表。
尽管已经有深度学习研究报道可以基于蛋白质序列信息预测与化合物的相互作用,但是还没有一种模型完全验证了“从序列到药物”的药物设计概念。在这项工作中,研究团队设计了TransformerCPI2.0作为概念验证的基础模型。该模型利用Transformer神经网络架构和预训练蛋白质语言模型,在蛋白质和化学多样性空间中都展示出了泛化能力。对TransformerCPI2.0进行可解释分析,结果表明模型可以通过注意力机制从序列信息中有效学习蛋白质与配体分子间相互作用的抽象知识,而不仅是简单的记忆训练数据中的分布偏差。
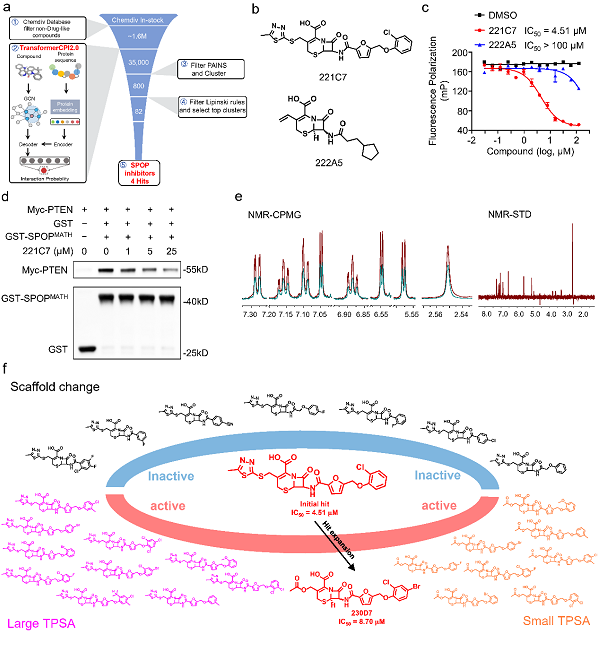
为了进一步检验基于蛋白质序列药物设计的可行性,研究团队还基于TransformerCPI2.0预测结果开展了湿实验的验证研究,针对多种具有挑战性的药物靶标成功发现活性化合物,包括:靶向E3泛素连接酶接头蛋白SPOP(配体结合位点为难以靶向的PPI作用界面)的新骨架抑制剂(图2);靶向E3泛素连接酶RNF130(无蛋白晶体结构、无已知活性化合物)的结合分子。此外,通过逆向应用TransformerCPI2.0对已上市药物进行蛋白质组范围的靶标筛选,成功发现雷贝拉唑抗肿瘤作用的潜在靶标ADP-核糖基化因子ARF1。这些新发现的活性分子和靶标蛋白都是模型训练过程未见的,表明TransformerCPI2.0可以推广到新的蛋白质和化学空间,显示了良好的泛化能力。计算分析和实验结果支持基于序列药物设计的可行性,基于这一概念的新方法 有望成为未来理性药物设计流程的重要组成部分。
这一工作通过计算机、信息学、化学和生物学等多学科融合交叉,为创新药物发现提供了新的视角。 基于序列的方法可以作为基于结构药物设计的替代方法,在无法获得高质量蛋白质3D结构的场景中发挥作用,也可与其他虚拟筛选和高通量体外筛选技术结合,加速药物发现进程。最近,超大规模按需定制化合物库(make-on-demand library)技术快速发展,已覆盖了数亿到数十亿种化学物质的多样性空间,如何开发计算效率更高的虚拟筛选方法成为领域的研究热点。“从序列到药物”的端到端学习策略可以有效的简化建模和筛选流程,可以更加高效的探索未知化学和生物学空间,为原创药物的发现提供新的出发点和切入点。
上海药物所研究生陈立凡,南京中医药大学联合培养研究生樊子生、常婕,博士后杨瑞瑞和侯辉为本文共同第一作者。上海药物所郑明月研究员、张素林副研究员为本文共同通讯作者。本研究得到了国家自然科学基金、临港实验室、国家重点研发专项、中科院青促会和上海市自然科学基金项目的基金资助。
图1. 基于序列的药物设计方法TransformerCPI2.0。a,基于结构药物设计与基于序列药物设计的流程对比。b,“从序列到药物”概念验证的三个阶段,包括:①评价跨蛋白质和化学空间的泛化能力(计算验证);②通过耐药性突变和取代基活性悬崖效应的预测检验模型是否学习到蛋白质-配体相互作用知识(可解释性分析);③开展具有挑战性的虚拟筛选和药物重定向预测(实验验证)。c,TransformerCPI2.0的架构。
图2. 利用TransformerCPI2.0基于蛋白质序列发现新骨架SPOP抑制剂。
(供稿部门:郑明月课题组)
闂備胶鎳撻悘姘跺箲娴h櫣鐭堥柨鐕傛嫹
濠电偞鍨堕幐鎼侇敄閸緷褰掑炊閳规儳浜鹃柣鐔煎亰濡插湱鈧鎸哥€涒晝鈧潧銈搁弫鍌炴倷椤掍焦鐦庨梺璇插缁嬫帡宕濋幒妤€绀夐柣鏃傚帶杩濇繝鐢靛Т濞茬娀宕戦幘鎰佹僵鐎规洖娲ㄩ悾铏圭磽閸屾瑧顦︽俊顐g矒瀹曟洟顢旈崨顖f祫闂佹寧绻傞悧鎾澄熺€n喗鐓欐繛鑼额嚙楠炴﹢鏌曢崶銊ュ摵鐎殿噮鍓熼獮宥夘敊閻e本娈搁梻浣藉亹閻℃棃宕归搹顐f珷闁秆勵殕椤ュ牓鏌涢幘鑼槮濞寸媭鍨堕弻鏇㈠幢濡ゅ﹤鍓遍柣銏╁灡婢瑰棗危閹版澘顫呴柣娆屽亾婵炲眰鍊曢湁闁挎繂妫欑粈瀣煃瑜滈崜姘┍閾忚宕查柛鎰ㄦ櫇椤╃兘鏌ㄥ┑鍡欏ⅵ婵☆垰顑夐弻娑㈠箳閹寸儐妫¢梺璇叉唉婵倗绮氶柆宥呯妞ゆ挾濮烽鎺楁⒑鐠団€虫灁闁告柨楠搁埢鎾诲箣閿旇棄娈ュ銈嗙墬缁矂鍩涢弽顓熺厱婵炲棙鍔曢悘鈺傤殽閻愬弶鍠橀柟顖氱Ч瀵噣宕掑Δ浣规珒
10x Genomics闂備礁鎼崐鐟邦熆濮椻偓楠炴牠鈥斿〒濯爄um HD 闁诲孩顔栭崰鎺楀磻閹剧粯鐓曟慨妯煎帶閻忕姷鈧娲滈崰鎾舵閹烘骞㈡慨姗嗗墮婵啴姊洪崨濠傜瑨婵☆偅绮嶉妵鏃堝箹娴g懓浠㈤梺鎼炲劗閺呮粓鎮鹃柆宥嗙厱闊洤顑呮慨鈧┑鐐存綑濡粓濡甸幇鏉垮嵆闁绘ḿ鏁搁悡浣虹磽娴e憡婀版俊鐐舵铻為柛褎顨呯粈鍡涙煕閳╁啞缂氶柍褜鍏涚划娆撳极瀹ュ鏅搁柨鐕傛嫹
婵犵數鍋涘Λ搴ㄥ垂閼测晜宕查悗锝庡亞閳绘棃鎮楅敐搴″箺缂佷胶娅墂ist闂備線娼уΛ妤呮晝閿濆洨绠斿鑸靛姇濡ɑ銇勯幘璺轰粶缂傚秳绶氶弻娑㈠冀閵娧冣拡濠电偛鐗婇崢顥窱SPR缂傚倷鐒︾粙鎺楁儎椤栫偛鐒垫い鎺嗗亾妞わ缚鍗抽幃褔宕妷銈嗗媰闂侀€炲苯澧村┑鈥愁嚟閳ь剨缍嗛崜姘跺汲閳哄懏鍊垫繛鎴炵懃婵啴鏌涢弮鎾村
闂備礁鎲¢〃鍡椕哄⿰鍛灊闊洦绋掗崵鍕煟閹邦剦鍤熼柕鍫熸尦楠炴牠寮堕幋鐘殿唶闂佸憡鐟ュΛ婵嗩潖婵犳艾惟闁靛绲煎ù鐑芥煟閻樿京鍔嶇憸鏉垮暣閹儵鏁撻敓锟� - 婵犵數鍎戠徊钘夌暦椤掑嫬鐭楅柛鈩冡缚椤╂煡鏌涢埄鍐惧毀闁圭儤鎸鹃々鐑藉箹鏉堝墽绉甸柛搴㈠灥閳藉骞橀姘濠电偞鍨堕幖鈺傜濠婂啰鏆﹂柣鏃囨绾惧ジ鏌涢埄鍐闁告梹甯¢幃妤呭捶椤撶偘妲愰梺缁樼⊕閻熝囧箯鐎n喖绠查柟浼存涧閹線姊洪崨濠傜濠⒀勵殜瀵娊鎮㈤悡搴n唹濡炪倖鏌ㄩ悘婵堢玻濞戙垺鐓欓悹銊ヮ槸閸婂鎮烽姀銈嗙厱婵炲棙锚閻忋儲銇勯銏╁剶鐎规洜濞€瀵粙顢栭锝呮诞鐎殿喗鎮傞弫鎾绘晸閿燂拷
濠电偞鍨堕幐鎼侇敄閸緷褰掑炊椤掆偓杩濇繝鐢靛Т鐎氼噣鎯屾惔銊︾厾鐎规洖娲ゆ禒婊堟煕閻愬瓨灏﹂柟钘夊€婚埀顒婄秵閸撴岸顢旈妶澶嬪仯闁规壋鏅涙俊铏圭磼閵娧冾暭闁瑰嘲鎳庨オ浼村礃閵娧€鍋撴繝姘厸閻庯綆鍋勬慨鍫ユ煛瀹€鈧崰搴ㄥ煝閺冨牆鍗抽柣妯挎珪濮e嫰鏌f惔銏⑩姇闁告梹甯″畷婵嬫偄閻撳宫銉╂煥閻曞倹瀚�



 闁诲孩顔栭崰妤€煤閻旂鈧倿顢曢妶鍡椾粧闁诲繒鍋犻崑鎰版偡瑜版帗鍤傜紒鍙夋尦閹虫洘绂掔€n偄娈忓┑顔矫畷顒佷繆閽樺)鏃堟儌缁嬫娲繝銏n潐鐢偟妲愰幒鎳崇喖骞愭惔锝嗩吇濠电偛顕刊瀵哥礊閳ь剟姊婚崘顓熺《缂佽鲸甯″畷鐔碱敃椤愩垹韦闂備浇妗ㄩ懗鍓佹崲濠靛牏绀婇悗锝庡墯閸犲棝鏌ㄥ┑鍡樺櫣闁哄鍋撻梻浣告惈閸婁粙锝炴径鎰┾偓渚€鏁撻敓锟�>>
闁诲孩顔栭崰妤€煤閻旂鈧倿顢曢妶鍡椾粧闁诲繒鍋犻崑鎰版偡瑜版帗鍤傜紒鍙夋尦閹虫洘绂掔€n偄娈忓┑顔矫畷顒佷繆閽樺)鏃堟儌缁嬫娲繝銏n潐鐢偟妲愰幒鎳崇喖骞愭惔锝嗩吇濠电偛顕刊瀵哥礊閳ь剟姊婚崘顓熺《缂佽鲸甯″畷鐔碱敃椤愩垹韦闂備浇妗ㄩ懗鍓佹崲濠靛牏绀婇悗锝庡墯閸犲棝鏌ㄥ┑鍡樺櫣闁哄鍋撻梻浣告惈閸婁粙锝炴径鎰┾偓渚€鏁撻敓锟�>>  闂備線娼уΛ妤呮嚐椤栫偛桅闁搞儮鏅欓悞濠囩叓閸ャ劎銆掗柣鎰功缁辨帞鎷嬮搹顐ゃ偘闂佺ǹ顑嗙粙鎺旂矙婢跺鍎熼柍銉ュ级濡﹪鏌熼崗鍏煎唉妞ゃ儲鎹囬、姗€骞栨担鐟颁哗闂佺硶鍓濋崝蹇涘磻閹炬剚鍚嬮柛鈩冾殘濡嫰鏌f惔銊︽锭闁告艾顑夐獮鎴︽晲婢跺﹦顔呴梺闈涱樈閸ㄥ磭绮堟径鎰厽闁归偊鍠楅崵鈧梺鎼炲€ら崳锝呯暦閿熺姴绀冮柟缁樺笩椤斿姊虹粙璺ㄧ缂佸鍏樺畷褰掑磼濞戝磭鐭楅梺绯曞墲椤ㄥ懐寰婇崸妤佺厽闁靛繈鍊栧☉褔鏌i埄鍐噰闁诡啫鍥ч唶闁绘棃顥撴禒鎰版煟鎼粹剝璐¢柛瀣缚缁辩偟鈧綆鈧垹缍婇弻銊р偓锝庘偓顖ゆ嫹>>
闂備線娼уΛ妤呮嚐椤栫偛桅闁搞儮鏅欓悞濠囩叓閸ャ劎銆掗柣鎰功缁辨帞鎷嬮搹顐ゃ偘闂佺ǹ顑嗙粙鎺旂矙婢跺鍎熼柍銉ュ级濡﹪鏌熼崗鍏煎唉妞ゃ儲鎹囬、姗€骞栨担鐟颁哗闂佺硶鍓濋崝蹇涘磻閹炬剚鍚嬮柛鈩冾殘濡嫰鏌f惔銊︽锭闁告艾顑夐獮鎴︽晲婢跺﹦顔呴梺闈涱樈閸ㄥ磭绮堟径鎰厽闁归偊鍠楅崵鈧梺鎼炲€ら崳锝呯暦閿熺姴绀冮柟缁樺笩椤斿姊虹粙璺ㄧ缂佸鍏樺畷褰掑磼濞戝磭鐭楅梺绯曞墲椤ㄥ懐寰婇崸妤佺厽闁靛繈鍊栧☉褔鏌i埄鍐噰闁诡啫鍥ч唶闁绘棃顥撴禒鎰版煟鎼粹剝璐¢柛瀣缚缁辩偟鈧綆鈧垹缍婇弻銊р偓锝庘偓顖ゆ嫹>>  闂備浇顫夊鍧楀储妤e喛缍栨繝闈涱儏绾偓闂佸搫娲ㄦ慨鐑芥儗鎼淬劍鐓犵€规洖娲ゆ禒锔剧磼娴e摜鎽犻柟顖涙閺佹捇鏁撻敓锟�-婵犵數鍎戠徊钘夌暦椤掑嫬鐭楅柛鈩冡缚椤╂煡鏌涢埄鍐惧毀闁圭儤鎸绘禍銈嗙箾閸℃ɑ灏柍褜鍓欑粔鎾綖閵忋倖鏅查柛娑卞枦閸氼偊姊虹悰鈥充壕闂佽崵鍠愬妯肩礊娓氣偓閺岀喓鈧稒锚閻忥絾绻濇繝鍐ㄧ仴妞ゆ洩缍侀崺鈧い鎺戝€峰▽顏堟煛閸モ晛顎屾繛鑲╁█閹鈽夐崘宸喘闂佺粯鐗炲▍锝囧垝濮樿泛绀堝ù锝呮贡閺嗙娀姊洪悷鎵邯闁稿鎹囬弻锟犲醇椤掍礁顏�>>
闂備浇顫夊鍧楀储妤e喛缍栨繝闈涱儏绾偓闂佸搫娲ㄦ慨鐑芥儗鎼淬劍鐓犵€规洖娲ゆ禒锔剧磼娴e摜鎽犻柟顖涙閺佹捇鏁撻敓锟�-婵犵數鍎戠徊钘夌暦椤掑嫬鐭楅柛鈩冡缚椤╂煡鏌涢埄鍐惧毀闁圭儤鎸绘禍銈嗙箾閸℃ɑ灏柍褜鍓欑粔鎾綖閵忋倖鏅查柛娑卞枦閸氼偊姊虹悰鈥充壕闂佽崵鍠愬妯肩礊娓氣偓閺岀喓鈧稒锚閻忥絾绻濇繝鍐ㄧ仴妞ゆ洩缍侀崺鈧い鎺戝€峰▽顏堟煛閸モ晛顎屾繛鑲╁█閹鈽夐崘宸喘闂佺粯鐗炲▍锝囧垝濮樿泛绀堝ù锝呮贡閺嗙娀姊洪悷鎵邯闁稿鎹囬弻锟犲醇椤掍礁顏�>>  濠电偞鍨堕幐濠氭偋濠婂牆鐤炬い鎰堕檮婵吋淇婇妶鍌氫壕闂佽鍠楀Σ鏄瞖rmo Fisher闂佽崵濮嶉崟顓熷垱缂備緡鍣崹閬嵥囨导瀛樺€烽柟娈垮枤閹虫劙鏌h箛鎾剁婵﹤顭烽敐鐐哄冀椤撶偟顔呴梺閫炲苯澧存鐐茬箰閳藉饪伴埀顒傚垝椤eld Application Scientist闂備線娼уΛ鏃€鎯斿〒涔簁eting Develop缂傚倷鐒︾粙鎴λ囬搹顐ゎ洸閻庢稒蓱婵鈧箍鍎辩换鎺旂矆婢舵劖鍋i柛銉у厴濡绢噣鏌涙惔锛勭闁归濞€閺屻劎鈧綆鍓欏銊╂⒑濞茶绨绘い鎴濇喘瀵偊骞樼紒妯轰罕闂佸啿鎼幊澶愬磻閹炬剚娼╂い鎰剁悼濠у嫰姊鸿ぐ鎺撴锭缂佸顕划鈺呭醇閺囩偞宓嶉梺缁樺灦宀e潡宕烘径鎰厽闁冲搫锕ゅ▍娆戠磼閹插瀚�>>
濠电偞鍨堕幐濠氭偋濠婂牆鐤炬い鎰堕檮婵吋淇婇妶鍌氫壕闂佽鍠楀Σ鏄瞖rmo Fisher闂佽崵濮嶉崟顓熷垱缂備緡鍣崹閬嵥囨导瀛樺€烽柟娈垮枤閹虫劙鏌h箛鎾剁婵﹤顭烽敐鐐哄冀椤撶偟顔呴梺閫炲苯澧存鐐茬箰閳藉饪伴埀顒傚垝椤eld Application Scientist闂備線娼уΛ鏃€鎯斿〒涔簁eting Develop缂傚倷鐒︾粙鎴λ囬搹顐ゎ洸閻庢稒蓱婵鈧箍鍎辩换鎺旂矆婢舵劖鍋i柛銉у厴濡绢噣鏌涙惔锛勭闁归濞€閺屻劎鈧綆鍓欏銊╂⒑濞茶绨绘い鎴濇喘瀵偊骞樼紒妯轰罕闂佸啿鎼幊澶愬磻閹炬剚娼╂い鎰剁悼濠у嫰姊鸿ぐ鎺撴锭缂佸顕划鈺呭醇閺囩偞宓嶉梺缁樺灦宀e潡宕烘径鎰厽闁冲搫锕ゅ▍娆戠磼閹插瀚�>>  闂備礁鎲¢崙褰掑礈濠靛鏁嬮柍鍝勫€规禍銈夋煕閵夋垵瀚禍閬嶆⒑閹稿海銆掗柛鐘虫礈缁梻鈧稒蓱娴溿倝鏌ㄩ悤鍌涘14濠电姰鍨归悘鍫ュ疾濞戙垺鍋柛鏇ㄥ墻濞堢晫鈧厜鍋撻柍褜鍓熼幃浼村Ψ閳轰胶鐓戦梺鎸庣箓濞诧箓鎮鹃柆宥嗙厾闁告瑥顦辩粻姘舵煕閹炬潙顥嬬紒杈ㄥ浮瀹曞崬顫濋鈧慨鍐⒑閹稿骸鍠曢柟鍑ゆ嫹>>
闂備礁鎲¢崙褰掑礈濠靛鏁嬮柍鍝勫€规禍銈夋煕閵夋垵瀚禍閬嶆⒑閹稿海銆掗柛鐘虫礈缁梻鈧稒蓱娴溿倝鏌ㄩ悤鍌涘14濠电姰鍨归悘鍫ュ疾濞戙垺鍋柛鏇ㄥ墻濞堢晫鈧厜鍋撻柍褜鍓熼幃浼村Ψ閳轰胶鐓戦梺鎸庣箓濞诧箓鎮鹃柆宥嗙厾闁告瑥顦辩粻姘舵煕閹炬潙顥嬬紒杈ㄥ浮瀹曞崬顫濋鈧慨鍐⒑閹稿骸鍠曢柟鍑ゆ嫹>>  生物通微信公众号
生物通微信公众号

