
-

生物通官微
陪你抓住生命科技
跳动的脉搏
真菌隐性次级代谢产物结构多样性形成机制新进展
【字体: 大 中 小 】 时间:2023年03月09日 来源:中国科学院微生物研究所
编辑推荐:
近日,中国科学院微生物研究所真菌学国家重点实验室尹文兵研究团队在 Angewandte Chemie 发表文章
近日,中国科学院微生物研究所真菌学国家重点实验室尹文兵研究团队在Angewandte Chemie发表文章。团队通过解析pretrichodermamide A的生物合成途径,揭示了非沉默生物合成途径中隐性多硫代二酮哌嗪类化合物(ETP)类次级代谢产物结构多样性的形成机制。
ETPs是一类由真菌产生的次级代谢产物,其中一个亚类pretrichodermamides含有独特的α, b'-二硫桥、1, 2-噁嗪环和复杂的多环结构(A–F环,图1),复杂的结构加大了其有机合成的难度,严重制约了pretrichodermamide的药用开发。因此,研究pretrichodermamides的生物合成途径是理解该类次级代谢产物结构复杂性和多样性的关键手段。
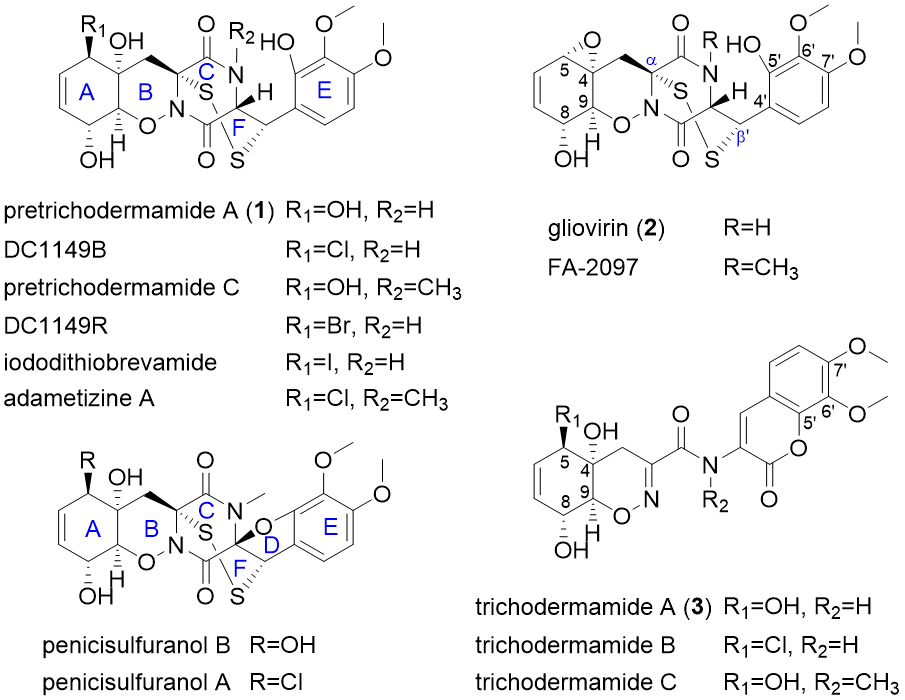
图1. 真菌中具有α, b-二硫桥的ETPs
前期研究中,尹文兵团队在炭团木霉中鉴定了pretrichodermamide A和其生物合成基因簇(Chemical Science, 2021, 12, 4132-4138)。本项研究则通过基因敲除和体外蛋白反应解析α, b'-二硫桥形成后的pretrichodermamide A生物合成步骤(图2),结果发现两个细胞色素P450氧化酶TdaB和TdaQ分别催化C8, C9过氧化和N10羟基化,再经由自发环化形成独特的1, 2-噁嗪环(B环),这一发现为1, 2-噁嗪环的酶法合成提供了新元件;P450氧化酶TdaI、甲基转移酶TdaO和TdaH依次作用,完成E环的修饰;还原酶TdaD将呋喃环(D环)还原;P450氧化酶TdaG催化环己二烯结构(A环)的氧化。
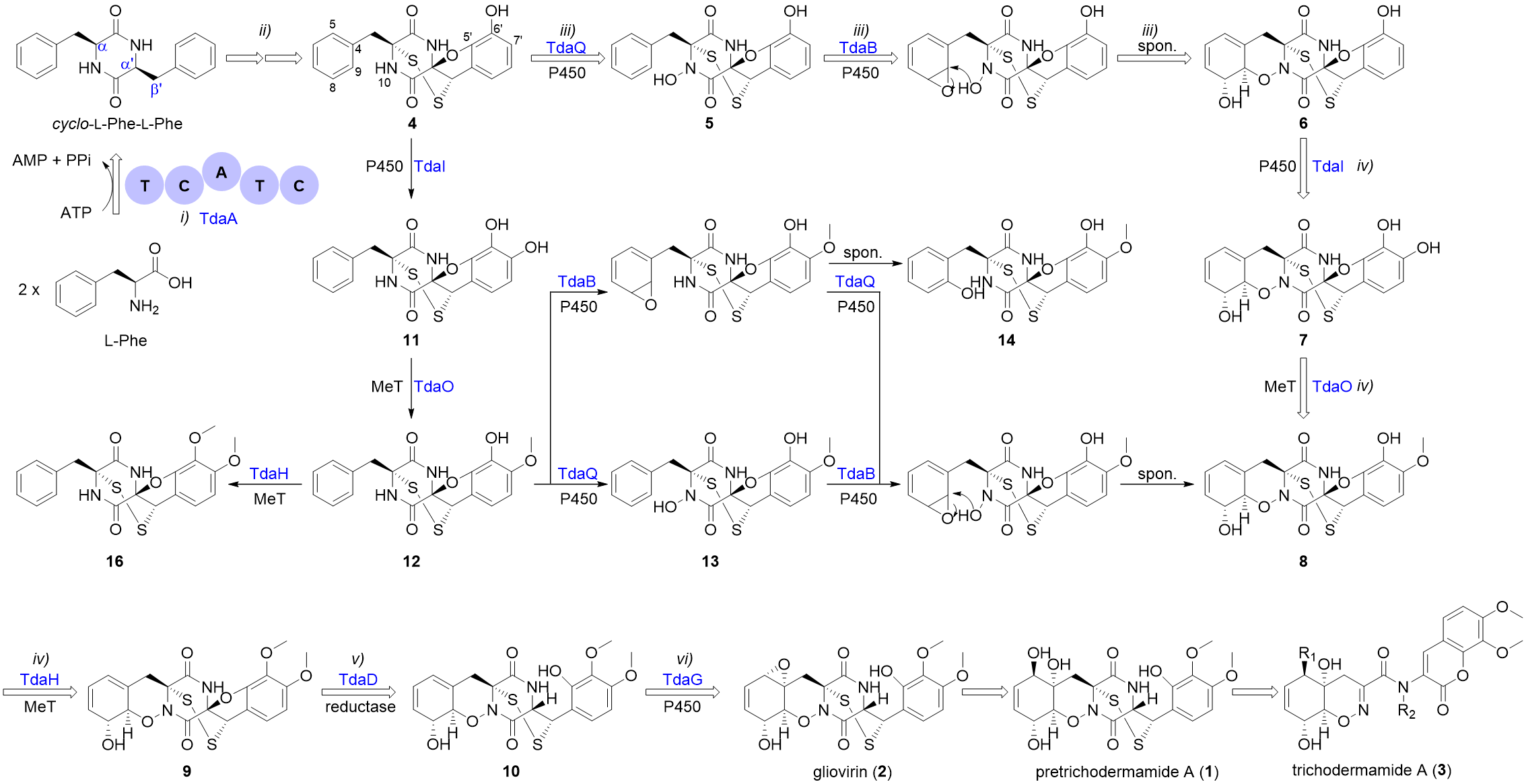
图2. Pretrichodermamide A的生物合成途径
作者在基因敲除突变株中还发现大量隐秘的ETPs,其中20个通过NMR进行了结构鉴定。比如,tdaI敲除后,其前体6可以被TdaG、TdaD和TdaH分别催化,经由三条不同的旁支途径,形成6a–6g、15、15c以及15g(图3),这些结果说明tda基因簇中不同的修饰酶虽然均具有良好的底物选择宽泛性,但它们催化的位点和反应却都是专一的。本研究以TdaG、TdaD和TdaH为例,通过不同底物喂养和体外蛋白反应,证实了它们底物选择的宽泛性。
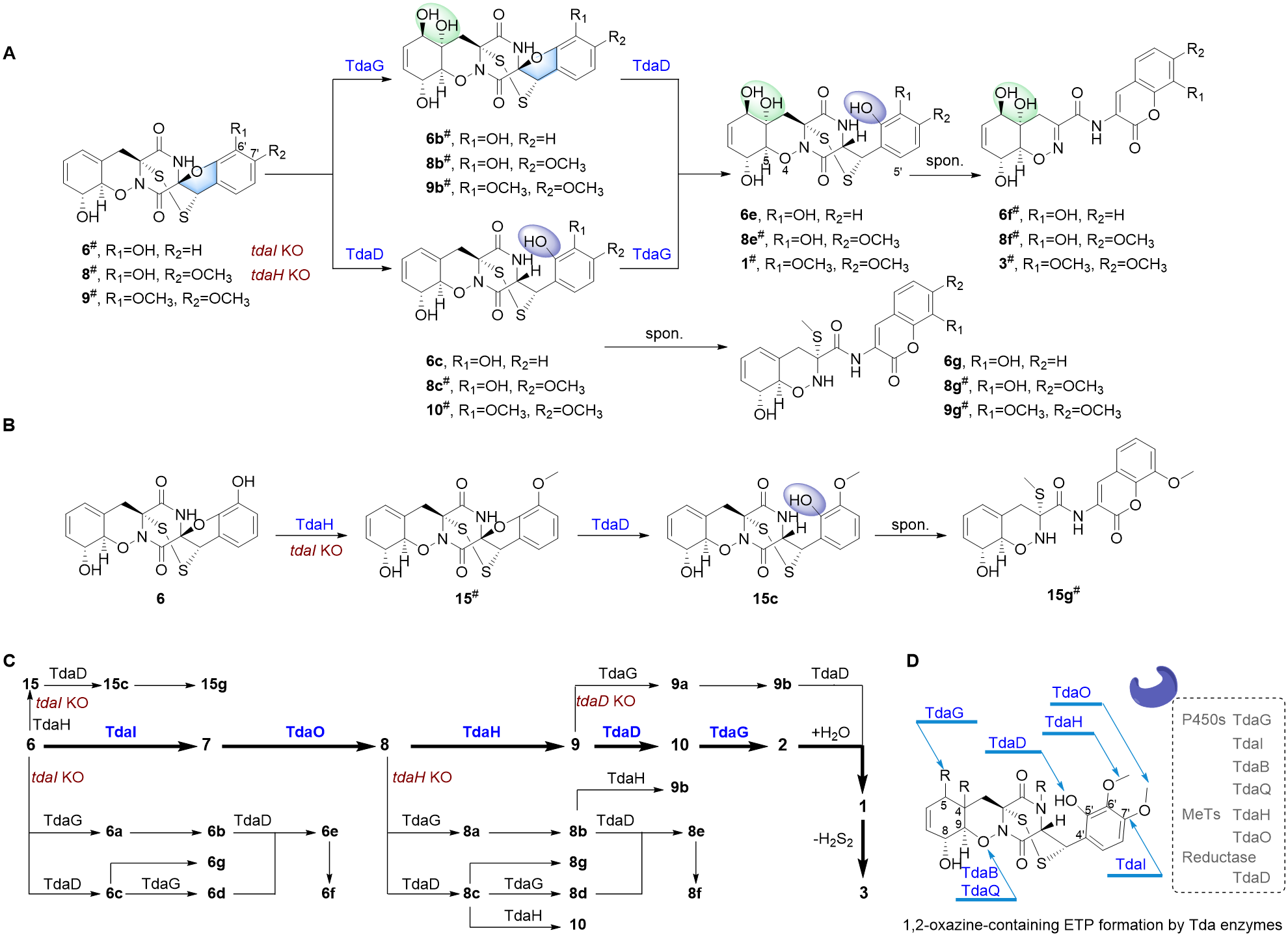
图3. Pretrichodermamide A生物合成途径中ETPs结构多样性形成机制
本研究通过解析α, b'-二硫桥形成后的pretrichodermamide A复杂生物合成步骤,鉴定了7个关键的修饰酶,发现超过20个隐秘的ETPs,揭示了该类次级代谢产物结构多样性的形成机制。又通过这些修饰酶的催化特性,实现ETPs的可预测生物合成,为其定向挖掘及多样性创制提供了新思路。
研究得到国家重点研发计划、国家自然科学基金委、中国科学院战略生物资源服务网络计划生物资源衍生库项目和博士后科学基金的资助。尹文兵研究员为本文通讯作者,特别研究助理范洁博士和冉火苗博士为共同第一作者,德国马尔堡大学李书明教授和中国医学科学院药物研究所胡友财研究员对本研究给予了大力支持。