
-

生物通官微
陪你抓住生命科技
跳动的脉搏
又见面了,Watson和Crick,我是Hoogsteen ——三螺旋介导的三元相互作用
【字体: 大 中 小 】 时间:2022年06月22日 来源:清华园生命学院
编辑推荐:
自然界的生物大分子的组装与去组装过程往往是受到多种元件来调控的,正因为复杂精确的调控,有限种类的单体分子才可以组织成复杂的形态而实现多种功能。典型的例子就是细胞骨架系统,仅由简单的微管蛋白和肌动蛋白单体就可以组装成复杂的微管和微丝网络,该网络对维持细胞结构和功能具有重要作用。因此实现响应特定刺激的自组装过程一直是研究人员热衷的方向,而如何在人工合成的系统中实现类似于自然界的刺激响应系统一直也是...
自然界的生物大分子的组装与去组装过程往往是受到多种元件来调控的,正因为复杂精确的调控,有限种类的单体分子才可以组织成复杂的形态而实现多种功能。典型的例子就是细胞骨架系统,仅由简单的微管蛋白和肌动蛋白单体就可以组装成复杂的微管和微丝网络,该网络对维持细胞结构和功能具有重要作用。因此实现响应特定刺激的自组装过程一直是研究人员热衷的方向,而如何在人工合成的系统中实现类似于自然界的刺激响应系统一直也是具有挑战的话题。在自组装的领域中,DNA纳米技术尤其是DNA折纸术创造出了各种各样具有高度可编程性和复杂度的核酸结构,经过数十年的发展,人们已经可以利用DNA折纸技术实现一些响应性自组装的过程。在本研究中,本文作者以三螺旋为基础设计三元相互作用,其中第三元作为刺激信号元素来引发自组装过程从而实现响应特定刺激的自组装。
具体来说,本文作者基于DNA三螺旋结构来构建三元作用体系,三螺旋中三条链通过寡聚嘌呤和寡聚嘧啶链之间的Watson-Crick碱基互补配对和两条寡聚嘌呤之间的反平行Hoogsteen碱基配对形成复合物。与之前报道的多种二元作用不同,这个三元作用体系由三个独立的元件(A,B和C)组成,我们分别用三螺旋的三条链来代表。如图1所示, C链作为独立于整个体系之外的刺激信号来介导A链和B链之间的相互作用。
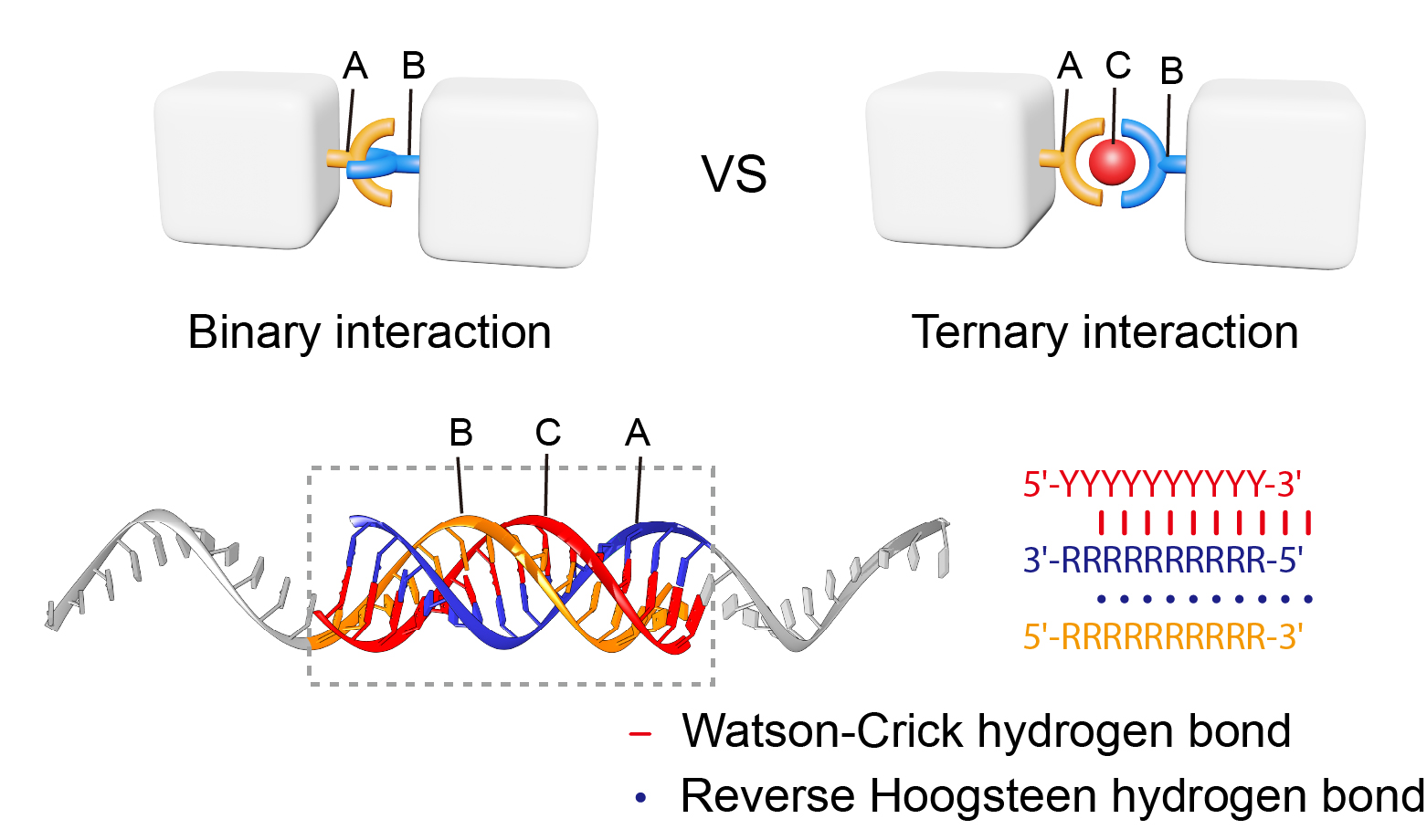
图1 基于DNA三螺旋的三元作用力。
验证想法的可行性,作者首先在简单的DNA结构中进行验证并优化出最佳的DNA三螺旋形成条件,接着又将优化的三螺旋结构用于介导DNA折纸结构的高级结构组装。通过对DNA折纸结构单体价态的调控,作者先是得到了长度逐渐增加的纳米纤维结构,当价态进一步提高之后,作者又观察到了一些其他的高级结构,如二维分枝结构和三维堆叠结构(图2)。

图2 基于DNA折纸结构的三元相互作用。通过改变作用位点的数目实现DNA折纸结构价态的精确调控,组装出越来越长的纳米纤维结构。当价态进一步增加又可以得到更多的高级结构。
作者进一步设计了基于三螺旋结构的正交反应体系,两组独立的三螺旋序列被设计出来并通过荧光标记和结构光成像的方法证实了该正交反应体系可以对特定的信号输入产生对应的输出信号(图3)。这也是明显优于此前报道过的基于pH刺激响应的三螺旋介导DNA折纸结构组装策略的地方,在pH响应系统中,很难实现对多种信号的特异性响应。

图3 基于三螺旋的选择性信号系统。在不同的信号输入下形成不同的结果,借助荧光标记和结构光成像,可以实现结果的可视化。没有任何输入的情况下,两种结构都保持单体,只能观察到分散的红色和绿色光点;在任意一种输入下,可以观察到一种颜色的短棒状结构和另外一种点状结构;当两种输入都存在时,可以看到两种颜色的棒状结构。
为了扩展该三元作用体系的应用,作者还成功的将该方法用于介导细菌的自组装,这也是首次报道的三螺旋介导的细菌相互作用。作者先通过代谢标记的方法使得细菌表面带有叠氮基团并借助点击化学的方法在细菌表面修饰上所需的DNA。最后在第三条链的作用下,细菌出现了明显的聚集性行为,通过共聚焦荧光显微的方法,成功表征了细菌的聚集行为(图4)。
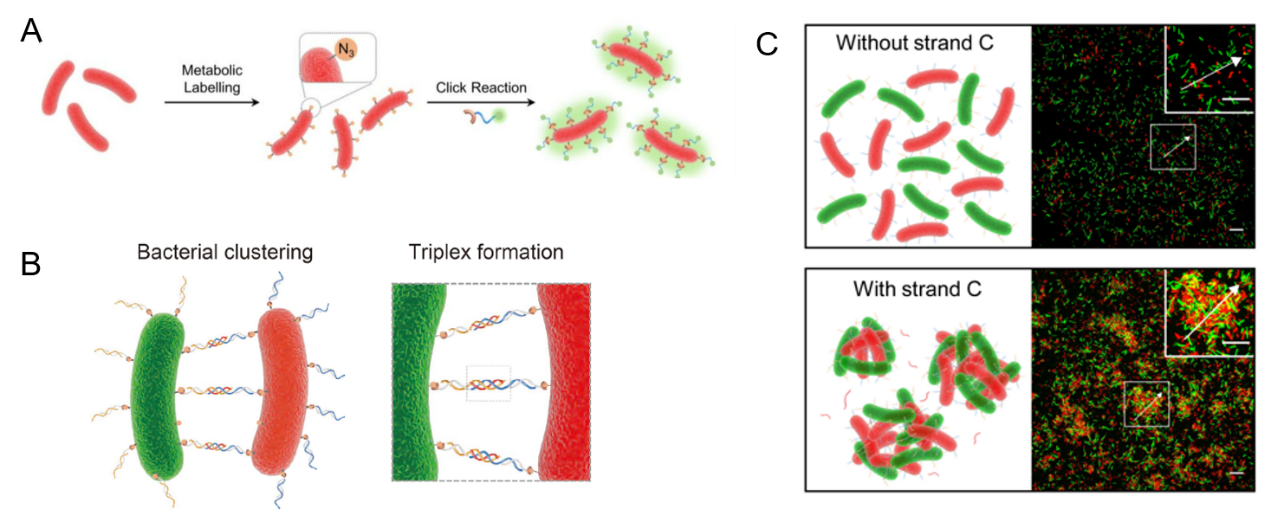
图4 DNA三螺旋介导的细菌组装过程。(A)通过代谢标记和点击化学的方式在细菌表面标记DNA。(B)三螺旋介导的细菌自组装示意图。(C)三螺旋介导的细菌自组装共聚焦显微镜图像结果
总的来说,本研究提出了新的三元作用的形式并借助DNA三螺旋实现了三元相互作用,作者成功应用三元作用调控DNA纳米结构和细菌的自组装过程,证实了该作用形式从纳米尺度到微米尺度都具有适用性。该研究是刺激响应性自组装的一个有意义的新概念尝试,对复杂精准的天然和人工自组装体系的机理阐释有启示意义,特别来说,也对人工细胞交互的动态调控也提供了一个实践路线,将有可能进一步被开发成为抗菌的新思路。
该成果由清华大学生命学院魏迪明分子设计课题组(MADlab)和湖南大学化学化工学院邢航课题组合作完成,论文题目为“使用可编程的DNA三元相互作用实现合成结构的可控自组装”(Controllable assembly of synthetic constructs with programmable ternary DNA interaction),于2022年6月17日发表于《核酸研究》(Nucleic Acid Research)上。清华大学生命学院2019级博士生崔黄陈,2017级博士生张天晴,湖南大学化学化工学院博士生孔玉涵为本文的共同第一作者,张天晴和清华大学生命学院魏迪明副教授,湖南大学湖南大学化学化工学院邢航教授共同为本文的通讯作者。该研究得到科技部,国家自然科学基金委、清华-北大生命科学联合中心、清华大学结构生物学高精尖中心等基金资助。另外值得一提的,此课题是先前分子设计课题组(MADlab)博士生张天晴提出的三螺旋作为核酸纳米结构组织方式研究的进一步展开。