
-

生物通官微
陪你抓住生命科技
跳动的脉搏
微生物所在难培养候选门纳米菌TM7类群挖掘培养与表面寄生机制研究中取得重要进展
【字体: 大 中 小 】 时间:2022年12月06日 来源:中国科学院微生物研究所
编辑推荐:
鎮ㄤ娇鐢ㄧ殑娴忚鍣ㄤ笉鏀寔鎴栨病鏈夊惎鐢╦avascript, 璇峰惎鐢╦avascript鍚庡啀璁块棶! [video:1] 该研究发表在 Proceedings of the National Academy of Sciences 上,题目为 “Type IV Pili Trigger Episymbiotic Association of Saccharibacteria with Its Bacterial Host” ( https://www.pnas.org/doi/10.1073/pnas.2215990119 )
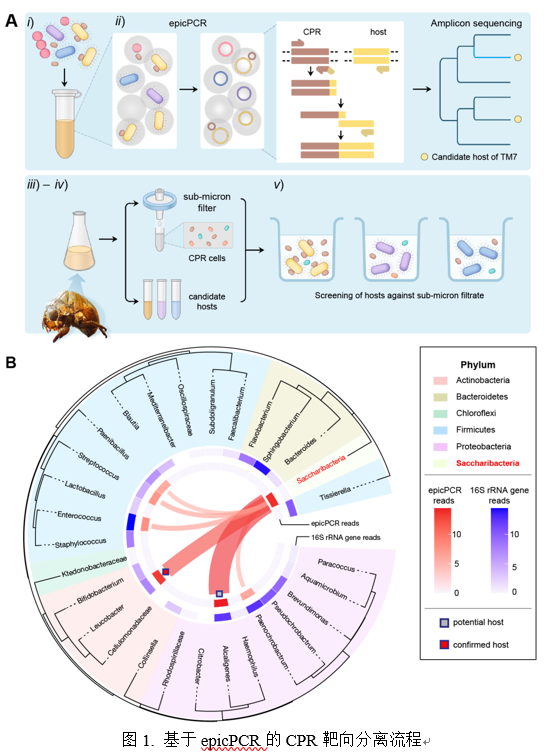
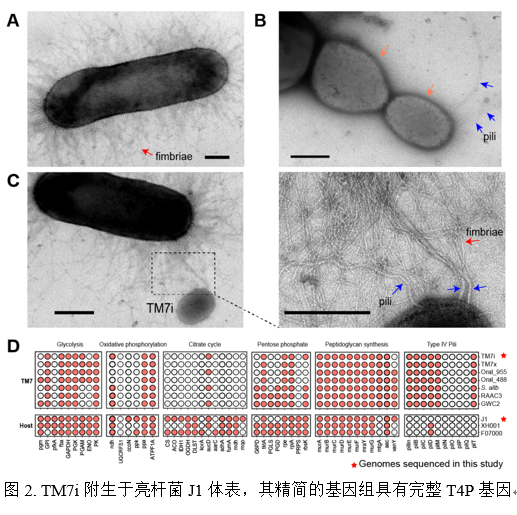
研究发现TM7i细菌专性地寄生于J1的体表,而TM7i基因组内保守的T4P则对其运动与宿主侵染至关重要。利用超高分辨率的成像揭示了CPR细菌是如何与宿主菌发生互作,并首次描述了TM7i完整的寄生生活史。
[video:2]
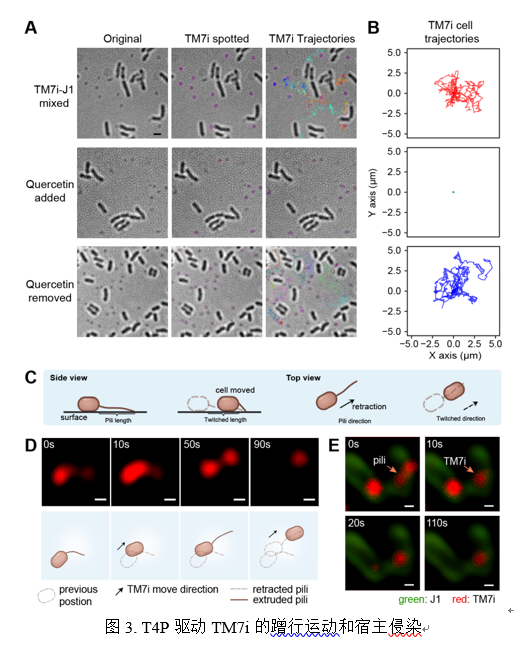
基于显微成像和定量PCR的结果还发现槲皮素可以种群水平上与宿主J1共生期间剂量依赖性地降低了TM7i的生长,表明T4P在TM7i识别和黏附宿主J1方面起着至关重要的作用。
[video:1]
该研究发表在Proceedings of the National Academy of Sciences上,题目为 “Type IV Pili Trigger Episymbiotic Association of Saccharibacteria with Its Bacterial Host”(https://www.pnas.org/doi/10.1073/pnas.2215990119)。微生物所博士生谢冰亮为第一作者,杜文斌研究员为通讯作者。研究得到了国家重点研发计划“难培养和微量病原体靶向培养技术研究”项目(2021YFC2301000)、国家自然科学基金委重大研究计划“水圈微生物驱动地球元素循环的机制”项目(91951103,92251302)等支持。