| Nature:对2500余例血浆样本进行cfRNA建库,成功预测疾病发生的风险 |
| |
随着NGS技术的普及和推广,很多科研人都已经比较了解甚至做过转录组建库测序实验。但,转录组建库真的简单吗?
很多这样或者那样的问题:
|
·我的样本是FFPE样本,但保存时间有点久,质量很差,降解特别严重,能建库吗?
·我的样本是血浆样本,RNA提取量真的太少了,加上还要去除rRNA,能建库吗?
·我的样本是游离RNA,做外泌体分析,这种特殊样本,这能建库吗?
·我的样本,我的样本。。。。。。
|
| |
想要解决以上问题,首先需要从源头把控,选择合适的核酸提取方案以获得足量RNA用于下游的应用;以及规范样本的取样操作及保存条件,尽量降低RNA降解的风险。
但还是有可能遇到RNA含量低,质量差的问题,该如何应对?
选择合适的文库构建试剂盒来优化文库构建环节不失为一种有效地补救措施,一般要关注以下三点:
|
(1)灵敏度:是否支持微量样本起始,有效检测基因数
(2)兼容度:是否支持不同品质的RNA样本,甚至是高度降解的样本
(3)简便性:整体流程是否操作时间短,简便易上手
|
| 上文Nature研究论文中所使用的Takara Bio 的RNA建库试剂盒SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian(Pico v2)就满足上述需求,可以针对微量的、质量差的样本,构建转录组文库的建库试剂盒。同时,Takara SMARTer® Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian(Pico v3),在Pico v2的基础上,反转录步骤增加8 nt UMI(分子标签),因此可以灵敏地识别PCR重复片段、校正PCR扩增错误并降低偏好性,从而进行正确的样本数据回溯,提供更可靠的RNA-Seq分析! |
| |
 |
| |
|
| ● 可应用于微量样本 |
RNA含量低,风险在于可能无法有效构建文库。这两个试剂盒可以从以下两个维度支持微量样本构建RNA-Seq文库!
其一,可以做到皮克级total RNA起始(范围支持250 pg-10 ng 人、大/小鼠total RNA)构建高品质文库!,即使珍贵的微量临床样本,也无需过于担心样本量不足!其二,不用先去除rRNA!虽然在制备文库前从总RNA中去除核糖体来源的RNA有利于降低测序成本,提高数据mapping率。但微量样品起始时则有可能会造成纯化后的样品太少而不能制备合格的测序文库。这两个试剂盒,采用的方法则是先合成cDNA测序文库,之后特异识别并去除来源于核糖体RNA的cDNA片段。简单方便的同时,也省去了单独购买rRNA去除产品的烦恼和费用! |
 |
| |
| ● 可应用于低品质样本 |
RIN值反映的是RNA的完整性,数值越大表明RNA质量越好,完整性越高。RIN≥8,表明RNA完整性好,无明显降解。RIN越低则说明RNA降解越严重,其构建的文库风险在于有效数据低,mapping率低,duplication高。
这两个试剂盒均支持分析质量非常差(RIN3-10,DV200≥25%)的FFPE、LCM、以及cfRNA等来源的样本!以随机引物起始,捕获全部mRNA和lncRNA在内的信息! |
| |
| 来看三个实际的应用案例: |
A. FFPE样本:FFPE样本的RNA-Seq分析,能为Biomarker筛查、回顾性研究、疾病机制探讨提供可能。然而,由于处理、保存等条件的限制,FFPE样本中不易留存高品质RNA。同时,提取RNA前不当的前处理方案,不合适的扩增方法也不易获得足够量的核酸用于NGS分析。
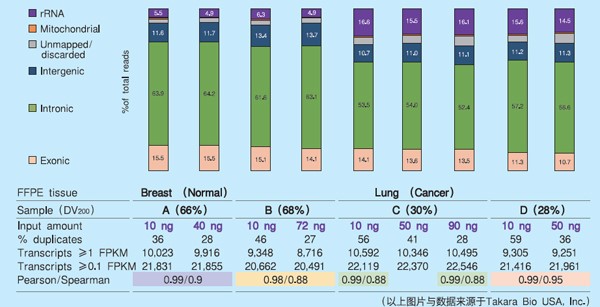
实验范例:取4个不同降解程度的FFPE-RNA样本(DV200在28%-68%之间),使用SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian制备文库,然后进行NGS分析。从数据中我们可以看出,①无论RNA起始量高低,或降解是否严重,Picov2均能获得约10,000的转录本检出数(FPKM≥1),说明Picov2具备足够高的灵敏度;②同一样本不同起始量之间数据的相关性系数达到0.99,说明Picov2所制备的文库具备高度的可重复性 |
 |
| |
B. cfRNA样本:存在于人体循环系统中一些内源性或外源性微量RNA片段,包括mRNA、miRNA和lncRNA等。由于在体外易被RNA酶降解,cfRNA完整性常常较低;加之提取的cfRNA常处于微量范畴,为科研工作者NGS分析这类样本增加难度。
文献范例:如本文开头所说,研究团队对来自8个独立队列的1840名孕妇的2539例血浆样本进行了cfRNA分析,就能在临床确诊几个月前预测孕期出现先兆子痫的风险。对1908例cfRNA图谱进行机器学习和建模,并对474例样本进行测试,其准确性与妊娠中期的超声检查相似且优于妊娠晚期超声检查。在案例中,使用SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian对cfRNA样本进行文库构建,并进行了靶向富集测序。由此说明:Pico v2非常适合cfRNA样本的可靠分析!
参考文献:Rasmussen M,Reddy M,Nolan R,et al. RNA profiles reveal signatures of future health and disease in pregnancy[J]. Nature, 2022, 601(7893). |
| |
C. LCM(激光捕获显微切割)样本:LCM是利用激光在显微镜下切割分离出单个细胞或纯化单一类型细胞群的一种技术。切割部分可用于进一步的分子生物学方法,如qPCR、NGS、蛋白质组学等。
文献范例: 研究人员对收集到的1,082份BCa FFPE标本,采用分层随机化方法分批处理标本。然后通过LCM获得切片用于后续实验。之后筛选出DV200>15%的样品用于后续NGS实验所用的样本。使用SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian Kit制备total RNA-Seq文库,并在Illumina NovaSeq 6000测序(PE 200),每个样本获得约1×109 reads。
参考文献:Paul ED, et al. Multiplexed RNA-FISH-guided Laser Capture Microdissection RNA Sequencing Improves Breast Cancer Molecular Subtyping, Prognostic Classification, and Predicts Response to Antibody Drug Conjugates. Preprint. medRxiv. 2023; 2023.12.05.23299341. |
| |
| 以上数据结果表明:面对不同类型的获得的微量亦或是低品质样本,Pico v2和Pico v3都可以胜任文库构建的任务! |
| |
| 看到这里,您可能打算买一个试试,但先别着急! |
近期,Takara对产品进行了升级,推出了Takara SMART-Seq® Total RNA Pico Input (ZapR™ Mammalian) 和SMART-Seq® Total RNA Pico Input with UMIs (ZapR™ Mammalian),在保留前一代可靠性能的基础上,有效提升rRNA去除效率,以更好地剔除不需要的reads信息。
(注:这两个试剂盒分别是Pico v2和Pico v3对应的升级版本,不再包含index) |
| |
| ● 实验范例:新旧版本的对比实验 |
| 选择250 pg的人脑RNA样本起始,使用SMART-Seq Total RNA Pico Input with UMIs制备文库。之后分别采用ZapR Mammalian rRNA Depletion Kit试剂盒中的模块处理(+)和未处理(-),然后通过PCR扩增进行富集或者不进行处理。同时,使用前一代版本产品SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian对250 pg的人脑RNA样本制备文库。最后使用CogentAP 对3x106PE reads进行数据分析。如下图所示,与前一代版本产品相比,SMART-Seq Total RNA Pico Input with UMIs (ZapR Mammalian)制备的文库,rRNA相关reads百分比明显降低,外显子reads百分比增加! |
 |
| |
| ● 实验范例:构建FFPE样本高品质文库 |
| 使用SMART-Seq® Total RNA Pico Input with UMIs (ZapR™ Mammalian) 制备10 ng FFPE RNA文库 (RIN=3, DV200=77%)。使用CogentAP进行数据分析,使用3x106 PE reads进行数据分析。如下图显示,即使以降解FFPE样本起始,可有效去除rRNA来源cDNA,并获得可靠的测序数据结果。 |
 |
| 而且针对以上提到的建库试剂盒,近期均有促销活动,感兴趣的小伙伴可以看看哦! |
| |
|
| |
 |
| |



