
-

生物通官微
陪你抓住生命科技
跳动的脉搏
AlphaFold和AI蛋白质折叠革命后的下一步
【字体: 大 中 小 】 时间:2022年04月14日 来源:nature
十多年来,分子生物学家马丁·贝克(Martin Beck)和他的同事一直在试图拼出世界上最难的拼图之一:人类细胞中最大的分子机器的详细模型。这个庞然大物,被称为核孔复合体,控制着分子进出细胞核的流动,而细胞核正是基因组所在的位置。每个细胞中都有数百个这样的复合物。每一种都由超过1000种蛋白质组成,它们一起在穿过核膜的孔周围形成环。
这1000块拼图是由30多个蛋白质组成的,它们以各种方式交织在一起。让谜题更难的是,这些积木的实验确定的3D形状是许多物种收集的结构的混合体,所以并不总是能很好地结合在一起。拼图盒子上的图片——核孔复合体的低分辨率3D视图——缺乏足够的细节,无法知道有多少碎片精确地组合在一起。
2016年,贝克领导的一个团队在德国法兰克福的马克斯·普朗克生物物理研究所(MPIB)报告了一个模型1,该模型覆盖了约30%的核孔复合体和约一半的30个构建块,称为Nup蛋白。
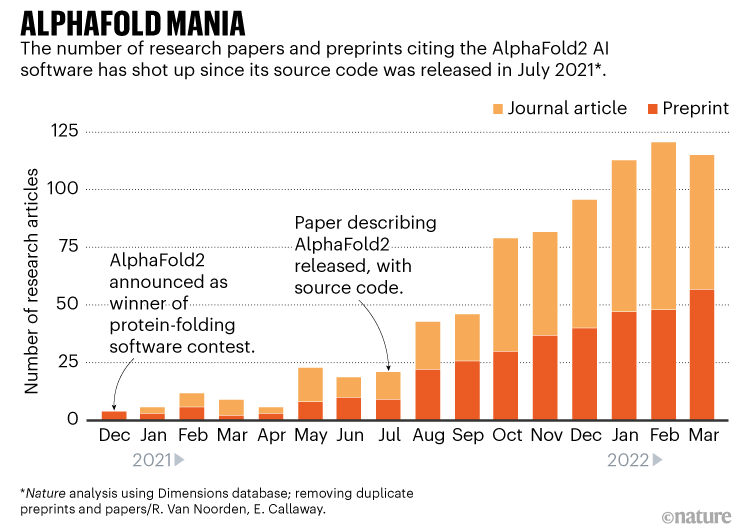
2021年7月,总部位于伦敦的DeepMind公司(谷歌母公司Alphabet的一部分)公开了一款名为AlphaFold2的人工智能工具。该软件可以根据基因序列预测蛋白质的3D形状,在很大程度上具有非常精确的准确性。这改变了贝克的工作,也改变了成千上万其他生物学家的研究。
通过AlphaFold,贝克和MPIB的分子生物学家Agnieszka Obarska-Kosinska和一个由生物化学家Gerhard Hummer领导的小组以及德国汉堡的欧洲分子生物学实验室(EMBL)结构建模师Jan Kosinski领导的团队,可以更准确地预测人类Nup蛋白的形状。通过对AlphaFold的一项调整,他们成功地在2021年10月发布了一个模型,该模型覆盖了60%的复合物。它揭示了复合物是如何稳定原子核中的孔洞的,同时也暗示了复合物是如何控制进出的物质的。
DeepMind的人工智能可以预测大量蛋白质的结构
在过去的半年里,AlphaFold热潮席卷了生命科学领域。“我参加的每一次会议,人们都在说,‘为什么不使用AlphaFold?’,”伦敦大学学院的计算生物学家Christine Orengo说。
在某些情况下,人工智能为科学家节省了时间;在另一些国家,它使以前难以想象或极其不切实际的研究成为可能。它有局限性,一些科学家发现它的预测对他们的工作来说太不可靠了。但实验的步伐是疯狂的。即使是那些开发软件的人,也在努力跟上它在从药物发现、蛋白质设计到复杂生命起源等领域的应用。“
AlphaFold在2020年12月的一场名为“蛋白质结构预测关键评估”(Critical Assessment of Protein Structure Prediction, CASP)的竞赛中占据了主导地位,引起了轰动。这项竞赛每两年举行一次,衡量生物学中最大挑战之一的进展:仅从蛋白质的氨基酸序列来确定蛋白质的3D形状。用X射线晶体学或低温电子显微镜(cryo-electron microscopy, cryo-EM)等实验方法,以建立蛋白质形状的图像。
第二版,AlphaFold2的预测与经验结构相当。
目前还不清楚DeepMind何时会将该软件或其预测广泛发布,因此研究人员使用了来自DeepMind AlphaFold团队的负责人John Jumper一次公开演讲的信息,以及他们自己的见解,开发了他们自己的人工智能工具,名为RoseTTAFold。
然后在2021年7月15日,出现了描述RoseTTAFold和AlphaFold2的论文,以及免费提供的开源代码和专家运行自己版本的工具所需的其他信息。一周后,DeepMind宣布,它已经使用AlphaFold预测了几乎每一种由人类制造的蛋白质的结构,以及其他20种被广泛研究的生物体的整个“蛋白质组”,如小鼠和大肠杆菌——总共超过365000个结构。DeepMind还将这些信息公开发布到英国辛克斯顿EMBL欧洲生物信息学研究所(EMBL–EBI)维护的数据库中。自那以后,该数据库已膨胀到近100万个结构。
E. Porta-Pardo et al. PLoS Comput. Biol. 18, e1009818 (2022).
今年,DeepMind计划发布总计超过1亿个结构预测。这几乎是所有已知蛋白质的一半,是蛋白质数据库(PDB)结构库中实验确定的蛋白质数量的数百倍。
AlphaFold部署了深度学习神经网络:受大脑神经线路启发的计算架构,以识别数据中的模式。它已经在PDB和其他数据库中通过实验确定了成千上万的蛋白质结构和序列。面对一个新的序列,它首先在数据库中寻找相关的序列,这些序列可以识别出倾向于一起进化的氨基酸,这表明它们在3D空间中接近。现有相关蛋白质的结构为估算新序列中氨基酸对之间的距离提供了另一种方法。
AlphaFold在试图为氨基酸的3D位置建模时,会从这些平行轨迹中反复迭代线索,并不断更新其估算值。专家表示,AlphaFold之所以如此出色,似乎是因为它在机器学习研究中应用了新思想,特别是它使用了一种名为“注意力”的人工智能机制,来确定在任何时候,哪些氨基酸连接对它的任务最重要。
DeepMind的蛋白质结构人工智能即将走入大众
该网络依赖于相关蛋白质序列的信息,这意味着AlphaFold有一些局限性。它不是用来预测突变对蛋白质形状的影响的,比如那些导致疾病的突变。它也没有被训练去确定蛋白质在其他相互作用的蛋白质或分子(如药物)存在时是如何改变形状的。但是它的模型带有分数,用来衡量网络对蛋白质中每个氨基酸单元的预测的信心。研究人员正在调整AlphaFold的代码,以扩大其功能。
据DeepMind称,到目前为止,已有超过40万人使用了EMBL-EBI的AlphaFold数据库。还有一些AlphaFold的“超级用户”:研究人员在自己的服务器上安装了该软件,或者使用基于云的AlphaFold版本来预测EMBL-EBI数据库之外的结构,或者设想该工具的新用途。
生物学家已经对AlphaFold解决结构问题的能力印象深刻。丹麦奥胡斯大学(Aarhus University)结构生物学家托马斯·博森(Thomas Boesen)说:“根据我目前所看到的情况,我非常信任AlphaFold。”该软件已经成功地预测了Boesen中心已经确定但尚未公布的蛋白质的形状。他说:“这是我对自己的肯定。”他正在使用AlphaFold来模拟细菌蛋白质的结构,这些蛋白质促进冰的形成,可能有助于云中冰的冷却效果,因为生物学家还不能通过实验完全确定这种结构。
斯德哥尔摩大学的蛋白质生物信息学家Arne Elofsson说,只要一个蛋白质卷曲成一个明确的3D形状——并不是所有的蛋白质都是这样——AlphaFold的预测就很难被击败。“这是一键式的解决方案,可能是你所能得到的最好的模式。”
AlphaFold不太自信的地方是,“它很擅长告诉你它什么时候不起作用”。这种本质上紊乱的区域——约占人类蛋白质组的三分之一——可能只有当另一种分子(如信号伴侣)出现时才会被明确定义。
伦敦癌症研究所(Institute of Cancer Research)的计算生物学家诺曼·戴维(Norman Davey)表示,AlphaFold识别疾病的能力,改变了他研究这些区域属性的工作。他说:“我们没有做任何努力,预测的质量立刻大幅提高。”
AlphaFold向EMBL-EBI数据库中倾倒的蛋白质结构也立即投入使用。Orengo的团队正在寻找新的蛋白质种类(没有经过实验验证),并且已经发现了成百上千种可能的新蛋白质家族,这扩大了科学家对蛋白质的外观和功能的认识。在另一项努力中,该团队正在搜索从海洋和废水中提取的DNA序列数据库,以试图识别新的食用塑料的酶。利用AlphaFold快速接近数千种蛋白质的结构,研究人员希望更好地了解酶是如何进化来分解塑料的,并有可能改进它们。
马萨诸塞州剑桥市哈佛大学的进化生物学家Sergey Ovchinnikov说,将任何蛋白质编码基因序列转换成可靠结构的能力对于进化研究来说应该是特别强大的。研究人员通过比较基因序列来确定生物及其基因在不同物种间的关系。对于远亲基因,比较可能无法找到进化上的亲缘关系,因为序列变化太大了。但是通过比较蛋白质结构——蛋白质结构往往比基因序列变化得慢——研究人员可能会发现被忽视的古老关系。苏黎世瑞士联邦理工学院的计算生物学家佩德罗·贝尔特劳说:“这为研究蛋白质进化和生命起源提供了一个惊人的机会。”
为了验证这一想法,由首尔国立大学计算生物学家马丁·斯坦格领导的一个团队和他的同事使用了他们开发的一种名为Foldseek的工具,在AlphaFold数据库中寻找导致COVID-19的病毒SARS-CoV-2的RNA复制酶的亲属。这项研究发现了之前未被确认的可能的古代近亲:真核生物中的蛋白质——包括黏液霉菌——在其3D结构上类似于被称为逆转录酶的酶,这种酶是HIV等病毒用来将RNA复制到DNA的,尽管在基因序列水平上几乎没有相似之处。
对于那些想要确定特定蛋白质的详细结构的科学家来说,AlphaFold预测并不一定是一个立即的解决方案。相反,它提供了一个可以通过实验验证或改进的初始近似值——这本身就有助于理解实验数据。例如,来自X射线结晶学的原始数据显示为衍射X射线的图案。通常,科学家需要对蛋白质的结构进行初步猜测,才能解释这些模式。英国剑桥大学的结构生物学家兰迪·里德(Randy Read)说,以前,他们经常从PDB中的相关蛋白质中拼凑信息,或者使用实验方法。他的实验室专门研究这些方法。现在,AlphaFold的预测使得大多数X射线图像不再需要这种方法,他的实验室正在努力在实验模型中更好地利用AlphaFold。“我们已经完全重新调整了研究的重点。”
人工智能为蛋白质折叠预测提供了动力
他和其他研究人员使用AlphaFold从X射线数据中确定晶体结构,如果没有一个足够的初始模型,这些数据是无法解释的。“人们正在解决多年来一直没有解决的结构,”Claudia Millán Nebot说,她曾在里德实验室做博士后,现在在剑桥的分析公司SciBite工作。她希望看到大量新的蛋白质结构提交给PDB,这在很大程度上是AlphaFold的结果。
对于专门从事低温电子显微镜的实验室来说,情况也是如此。低温电子显微镜可以捕捉快速冷冻蛋白质的图片。位于教堂山的北卡罗莱纳大学的结构生物学家和药理学家Bryan Roth说,在某些情况下,AlphaFold的模型准确地预测了被称为G蛋白偶联受体(GPCRs)的蛋白质的独特特征——这是重要的药物靶标——而其他计算工具却错了。他说:“这似乎对生成第一个模型非常有利,然后我们用一些实验数据对其进行改进。这节省了我们一些时间。”
但Roth补充说,AlphaFold并不总是那么准确。他说,在他的实验室已经解决但尚未发表的几十个GPCR结构中,“大约有一半的情况下,AlphaFold结构相当好,而另一半情况下,它们或多或少对我们的目的没有用处。”他说,在某些情况下,AlphaFold对预测进行了高度自信的标注,但实验结构表明它是错误的。即使软件做对了,它也不能模拟出蛋白质与药物或其他小分子(配体)结合时的样子,而这些小分子会极大地改变蛋白质的结构。这样的警告让罗斯想知道AlphaFold在药物研发方面到底有多大用处。
在药物发现过程中,使用计算对接软件来筛选数十亿个小分子,以找到一些可能与蛋白质结合的分子——这表明它们可以制造有用的药物,这种做法越来越普遍。
他们的工作局限于那些AlphaFold的预测与实验结构相吻合的蛋白质。但即使在这些情况下,对接软件也为实验结构和AlphaFold的服用提供了不同的药物,这表明微小的差异也可能产生影响。“这并不意味着我们找不到新的配体,我们只是找到了不同的配体。”他们的团队现在正在合成利用AlphaFold结构识别出的潜在药物,并在实验室中测试它们的活性。
制药公司和生物技术公司的研究人员对AlphaFold在药物研发方面的潜力感到兴奋。2021年11月,DeepMind推出了自己的子公司同构实验室(IsoMorphic Labs),旨在将AlphaFold和其他人工智能工具应用于药物发现。但该公司几乎没有透露其他有关计划的信息。
凯伦·阿金山亚(Karen Akinsanya)是某药物研发公司的负责人,该公司总部位于纽约市,也发布化学模拟软件。她说,她和同事们已经在将AlphaFold结构应用于虚拟筛选和候选药物的化合物设计方面取得了一些成功,包括用于GPCRs。她发现,就像实验结构一样,需要额外的软件来获得氨基酸侧链的细节或者单个氢原子可能所在的位置。一旦完成,AlphaFold结构已经被证明足以指导药物发现——在某些情况下。
“很难说‘这是灵丹妙药’;因为你可以对一个结构做得很好——令人惊讶和兴奋的好——所以它非常适用于所有的结构。显然不是,”Akinsanya说。她和她的同事们发现,AlphaFold的准确性预测并没有显示出一个结构是否会对以后的药物筛选有用。她说,AlphaFold结构永远不会完全取代药物发现中的实验性结构。但他们可能会通过补充实验方法来加快这个过程。
DeepMind取消了对AlphaFold用于商业应用的关键限制。当该公司在2021年7月发布AlphaFold的代码时,它规定运行AlphaFold神经网络所需的参数(或权重)只能用于非商业用途。AlphaFold神经网络是根据数十万蛋白质结构和序列训练网络的最终结果。Akinsanya表示,这对一些行业来说是一个瓶颈,当DeepMind改变策略时,引发了一波“兴奋的浪潮”。(RoseTTAFold也有类似的限制,它的开发者之一Ovchinnikov说。但下一个版本将是完全开源的。)
人工智能工具不仅仅改变了科学家对蛋白质外观的判断。一些研究人员正在使用它们来制造全新的蛋白质。“深度学习正在彻底改变我们团队中蛋白质设计的方式,”华盛顿大学西雅图分校的生物化学家大卫·贝克(David Baker)说,他是蛋白质设计和结构预测领域的领军人物。他的团队和计算化学家Minkyung Baek领导了开发RoseTTAFold的工作。
贝克的团队让AlphaFold和RoseTTAFold“产生幻觉”新的蛋白质。研究人员已经改变了人工智能代码,这样,给定氨基酸的随机序列,软件将优化它们,直到它们类似于神经网络识别的蛋白质。
2021年12月,贝克和他的同事报告称,细菌中表达了129种这种幻觉蛋白,并发现大约五分之一的蛋白质折叠成与预测形状相似的形状。贝克说:“这是第一次证明你可以利用这些网络设计蛋白质。”他的团队现在正在使用这种方法来设计蛋白质,通过指定负责所需功能的氨基酸,让人工智能想象出其他的功能,比如催化特定的化学反应。

四个蛋白质“幻觉”的例子。在每一种情况下,AlphaFold都会呈现一个随机的氨基酸序列,预测结构,并改变序列,直到软件自信地预测它会折叠成一个定义明确的3D形状的蛋白质。颜色显示预测的信心(从红色表示非常低的信心,从黄色和浅蓝色到深蓝色表示非常高的信心)。为了清晰,初始帧被放慢。
当DeepMind发布其AlphaFold代码时,Ovchinnikov希望更好地了解该工具的工作原理。几天之内,他和计算生物学的同事,建立了一个名为ColabFold的网站,允许任何人向AlphaFold或RoseTTAFold提交蛋白质序列,并获得结构预测。Ovchinnikov设想,他和其他科学家将使用ColabFold来尝试“打破”AlphaFold,例如,通过提供有关目标蛋白质序列进化亲属的错误信息。通过这样做,Ovchinnikov希望他能够确定网络是如何学会如此好地预测结构的。
事实证明,大多数使用ColabFold的研究人员只是想得到一种蛋白质结构。但也有人将其用作平台,修改AlphaFold的输入,以应对新的应用。“我没想到会有这么多不同类型的黑客,”Jumper说。
到目前为止,最流行的方法是使用这个工具来研究由多个相互作用的——通常是相互缠绕的——肽链组成的蛋白质复合物。就像核孔复合体一样,细胞中的许多蛋白质在与多个蛋白质亚基形成复合物时获得它们的功能。
AlphaFold的设计目的是预测单肽链的形状,它的训练完全由这类蛋白质组成。但这个网络似乎已经了解到复杂的东西是如何组合在一起的。AlphaFold的代码公布几天后,东京大学(University of Tokyo)的蛋白质生物信息学家Yoshitaka Moriwaki在推特上表示,如果用一长串连接序列将两个蛋白质序列拼接在一起,它就可以准确预测它们之间的相互作用。白很快分享了另一种预测复合体的方法,这是从开发RoseTTAFold中收集到的。
ColabFold后来加入了预测复合物的能力。2021年10月,DeepMind发布了一款名为AlphaFold-Multimer8的更新,与之前的版本不同,它专门针对蛋白质复合物进行训练。Jumper的团队将其应用于PDB中的数千个复合物,发现它可以预测约70%的已知蛋白质-蛋白质相互作用。
这些工具已经在帮助研究人员发现潜在的新的蛋白质伴侣。Elofsson的团队利用AlphaFold软件预测了65000对人类蛋白质的结构,这些蛋白质根据实验数据被怀疑会相互作用。贝克领导的一个研究小组使用AlphaFold和RoseTTAFold来模拟几乎每一对酵母编码的蛋白质之间的相互作用,确定了100多个以前未知的复合物。Elofsson说,这样的屏幕只是一个起点。它们在预测一些蛋白质配对方面做得很好,尤其是那些稳定的蛋白质配对,但很难识别更多的短暂的相互作用。Elofsson表示:“看起来不错并不意味着它就是正确的。你需要一些实验数据来证明你是正确的。”
核孔复合体的工作是预测和实验数据如何协同工作的一个很好的例子,Kosinski说,“这并不像我们把所有30种蛋白质,扔到AlphaFold中,然后得到结构。”为了将预测的蛋白质结构组合在一起,研究小组使用了核孔复合体的3D图像,这些图像是用一种称为冷冻电子断层扫描的冷冻电子显微镜捕获的。在一个例子中,可以确定蛋白质接近程度的实验发现,复合体的两个组成部分之间出现了令人惊讶的相互作用,AlphaFold的模型随后证实了这一点。该团队目前绘制的核孔复合体图是实验和模拟的起点,这些实验和模拟研究了核孔复合体是如何发挥作用的——以及它在疾病中是如何失效的。
对于AlphaFold取得的所有进展,科学家们表示,重要的是要清楚它的局限性——尤其是因为研究人员并不擅长预测蛋白质结构。
将AlphaFold应用于破坏蛋白质自然结构的各种突变(包括与早期乳腺癌有关的突变)的尝试证实,该软件不具备预测蛋白质新突变的后果的能力,因为没有可检测的与进化相关的序列。
AlphaFold团队目前正在考虑如何设计神经网络来应对新的突变。Jumper预计,这将需要该网络更好地预测蛋白质从展开状态到折叠状态的过程。纽约市哥伦比亚大学的计算生物学家Mohammed AlQuraishi说,这可能需要一种仅依靠已知的蛋白质物理学知识来预测结构的软件。他说:“我们感兴趣的一件事是,在不使用进化信息的情况下,从单个序列进行预测。这确实是一个悬而未决的关键问题。”
AlphaFold的设计初衷也是为了预测一个单一的结构,尽管它已经被黑客攻击,可以预测出多个结构。但许多蛋白质具有多种构象,这对它们的功能很重要。“AlphaFold无法真正处理那些可以采用不同构象的不同结构的蛋白质,”Schueler-Furman说。这些预测是针对孤立的结构,而许多蛋白质与配体(如DNA和RNA)、脂肪分子和矿物质(如铁)一起发挥作用。Elofsson说:“我们仍然缺少配体,我们还缺少蛋白质的其他一切。”
开发下一代神经网络将是一个巨大的挑战,AlQuraishi说。AlphaFold依赖于数十年的研究,这些研究生成了网络可以学习的实验性蛋白质结构。这么大的数据量目前还无法捕捉蛋白质的动态变化,也无法捕捉蛋白质可能与之相互作用的数万亿个小分子的形状。当蛋白质与其他分子相互作用时,PDB包含蛋白质的结构,但这只是捕捉到化学多样性的一小部分,Jumper补充道。
研究人员认为,他们需要时间来确定如何最好地使用AlphaFold和相关的人工智能工具。AlQuraishi看到了早期电视节目的相似之处,当时一些节目只是由广播电台播报新闻。“我认为,我们将发现结构的新应用,这是我们尚未想到的。”
AlphaFold革命的结局是什么,谁都说不准。“事情变化得太快了,”贝克说。“即使在明年,我们也将看到使用这些工具取得真正重大的突破。”EMBL-EBI的计算生物学家珍妮特·桑顿(Janet Thornton)认为,AlphaFold最大的影响之一可能只是说服生物学家更开放地接受计算和理论方法的见解。她说:“对我来说,这场革命就是心态的改变。”
AlphaFold革命激发了科辛斯基的远大梦想。他设想,受AlphaFold启发的工具不仅可以用来模拟单个蛋白质和复合物,还可以模拟整个细胞器,甚至是单个蛋白质分子水平的细胞。“这是我们未来几十年要追随的梦想。”
 闁诲孩顔栭崰妤€煤閻旂鈧倿顢曢妶鍡椾粧闁诲繒鍋犻崑鎰版偡瑜版帗鍤傜紒鍙夋尦閹虫洘绂掔€n偄娈忓┑顔矫畷顒佷繆閽樺)鏃堟儌缁嬫娲繝銏n潐鐢偟妲愰幒鎳崇喖骞愭惔锝嗩吇濠电偛顕刊瀵哥礊閳ь剟姊婚崘顓熺《缂佽鲸甯″畷鐔碱敃椤愩垹韦闂備浇妗ㄩ懗鍓佹崲濠靛牏绀婇悗锝庡墯閸犲棝鏌ㄥ┑鍡樺櫣闁哄鍋撻梻浣告惈閸婁粙锝炴径鎰┾偓渚€鏁撻敓锟�>>
闁诲孩顔栭崰妤€煤閻旂鈧倿顢曢妶鍡椾粧闁诲繒鍋犻崑鎰版偡瑜版帗鍤傜紒鍙夋尦閹虫洘绂掔€n偄娈忓┑顔矫畷顒佷繆閽樺)鏃堟儌缁嬫娲繝銏n潐鐢偟妲愰幒鎳崇喖骞愭惔锝嗩吇濠电偛顕刊瀵哥礊閳ь剟姊婚崘顓熺《缂佽鲸甯″畷鐔碱敃椤愩垹韦闂備浇妗ㄩ懗鍓佹崲濠靛牏绀婇悗锝庡墯閸犲棝鏌ㄥ┑鍡樺櫣闁哄鍋撻梻浣告惈閸婁粙锝炴径鎰┾偓渚€鏁撻敓锟�>>  闂備線娼уΛ妤呮嚐椤栫偛桅闁搞儮鏅欓悞濠囩叓閸ャ劎銆掗柣鎰功缁辨帞鎷嬮搹顐ゃ偘闂佺ǹ顑嗙粙鎺旂矙婢跺鍎熼柍銉ュ级濡﹪鏌熼崗鍏煎唉妞ゃ儲鎹囬、姗€骞栨担鐟颁哗闂佺硶鍓濋崝蹇涘磻閹炬剚鍚嬮柛鈩冾殘濡嫰鏌f惔銊︽锭闁告艾顑夐獮鎴︽晲婢跺﹦顔呴梺闈涱樈閸ㄥ磭绮堟径鎰厽闁归偊鍠楅崵鈧梺鎼炲€ら崳锝呯暦閿熺姴绀冮柟缁樺笩椤斿姊虹粙璺ㄧ缂佸鍏樺畷褰掑磼濞戝磭鐭楅梺绯曞墲椤ㄥ懐寰婇崸妤佺厽闁靛繈鍊栧☉褔鏌i埄鍐噰闁诡啫鍥ч唶闁绘棃顥撴禒鎰版煟鎼粹剝璐¢柛瀣缚缁辩偟鈧綆鈧垹缍婇弻銊р偓锝庘偓顖ゆ嫹>>
闂備線娼уΛ妤呮嚐椤栫偛桅闁搞儮鏅欓悞濠囩叓閸ャ劎銆掗柣鎰功缁辨帞鎷嬮搹顐ゃ偘闂佺ǹ顑嗙粙鎺旂矙婢跺鍎熼柍銉ュ级濡﹪鏌熼崗鍏煎唉妞ゃ儲鎹囬、姗€骞栨担鐟颁哗闂佺硶鍓濋崝蹇涘磻閹炬剚鍚嬮柛鈩冾殘濡嫰鏌f惔銊︽锭闁告艾顑夐獮鎴︽晲婢跺﹦顔呴梺闈涱樈閸ㄥ磭绮堟径鎰厽闁归偊鍠楅崵鈧梺鎼炲€ら崳锝呯暦閿熺姴绀冮柟缁樺笩椤斿姊虹粙璺ㄧ缂佸鍏樺畷褰掑磼濞戝磭鐭楅梺绯曞墲椤ㄥ懐寰婇崸妤佺厽闁靛繈鍊栧☉褔鏌i埄鍐噰闁诡啫鍥ч唶闁绘棃顥撴禒鎰版煟鎼粹剝璐¢柛瀣缚缁辩偟鈧綆鈧垹缍婇弻銊р偓锝庘偓顖ゆ嫹>>  闂備浇顫夊鍧楀储妤e喛缍栨繝闈涱儏绾偓闂佸搫娲ㄦ慨鐑芥儗鎼淬劍鐓犵€规洖娲ゆ禒锔剧磼娴e摜鎽犻柟顖涙閺佹捇鏁撻敓锟�-婵犵數鍎戠徊钘夌暦椤掑嫬鐭楅柛鈩冡缚椤╂煡鏌涢埄鍐惧毀闁圭儤鎸绘禍銈嗙箾閸℃ɑ灏柍褜鍓欑粔鎾綖閵忋倖鏅查柛娑卞枦閸氼偊姊虹悰鈥充壕闂佽崵鍠愬妯肩礊娓氣偓閺岀喓鈧稒锚閻忥絾绻濇繝鍐ㄧ仴妞ゆ洩缍侀崺鈧い鎺戝€峰▽顏堟煛閸モ晛顎屾繛鑲╁█閹鈽夐崘宸喘闂佺粯鐗炲▍锝囧垝濮樿泛绀堝ù锝呮贡閺嗙娀姊洪悷鎵邯闁稿鎹囬弻锟犲醇椤掍礁顏�>>
闂備浇顫夊鍧楀储妤e喛缍栨繝闈涱儏绾偓闂佸搫娲ㄦ慨鐑芥儗鎼淬劍鐓犵€规洖娲ゆ禒锔剧磼娴e摜鎽犻柟顖涙閺佹捇鏁撻敓锟�-婵犵數鍎戠徊钘夌暦椤掑嫬鐭楅柛鈩冡缚椤╂煡鏌涢埄鍐惧毀闁圭儤鎸绘禍銈嗙箾閸℃ɑ灏柍褜鍓欑粔鎾綖閵忋倖鏅查柛娑卞枦閸氼偊姊虹悰鈥充壕闂佽崵鍠愬妯肩礊娓氣偓閺岀喓鈧稒锚閻忥絾绻濇繝鍐ㄧ仴妞ゆ洩缍侀崺鈧い鎺戝€峰▽顏堟煛閸モ晛顎屾繛鑲╁█閹鈽夐崘宸喘闂佺粯鐗炲▍锝囧垝濮樿泛绀堝ù锝呮贡閺嗙娀姊洪悷鎵邯闁稿鎹囬弻锟犲醇椤掍礁顏�>>  濠电偞鍨堕幐濠氭偋濠婂牆鐤炬い鎰堕檮婵吋淇婇妶鍌氫壕闂佽鍠楀Σ鏄瞖rmo Fisher闂佽崵濮嶉崟顓熷垱缂備緡鍣崹閬嵥囨导瀛樺€烽柟娈垮枤閹虫劙鏌h箛鎾剁婵﹤顭烽敐鐐哄冀椤撶偟顔呴梺閫炲苯澧存鐐茬箰閳藉饪伴埀顒傚垝椤eld Application Scientist闂備線娼уΛ鏃€鎯斿〒涔簁eting Develop缂傚倷鐒︾粙鎴λ囬搹顐ゎ洸閻庢稒蓱婵鈧箍鍎辩换鎺旂矆婢舵劖鍋i柛銉у厴濡绢噣鏌涙惔锛勭闁归濞€閺屻劎鈧綆鍓欏銊╂⒑濞茶绨绘い鎴濇喘瀵偊骞樼紒妯轰罕闂佸啿鎼幊澶愬磻閹炬剚娼╂い鎰剁悼濠у嫰姊鸿ぐ鎺撴锭缂佸顕划鈺呭醇閺囩偞宓嶉梺缁樺灦宀e潡宕烘径鎰厽闁冲搫锕ゅ▍娆戠磼閹插瀚�>>
濠电偞鍨堕幐濠氭偋濠婂牆鐤炬い鎰堕檮婵吋淇婇妶鍌氫壕闂佽鍠楀Σ鏄瞖rmo Fisher闂佽崵濮嶉崟顓熷垱缂備緡鍣崹閬嵥囨导瀛樺€烽柟娈垮枤閹虫劙鏌h箛鎾剁婵﹤顭烽敐鐐哄冀椤撶偟顔呴梺閫炲苯澧存鐐茬箰閳藉饪伴埀顒傚垝椤eld Application Scientist闂備線娼уΛ鏃€鎯斿〒涔簁eting Develop缂傚倷鐒︾粙鎴λ囬搹顐ゎ洸閻庢稒蓱婵鈧箍鍎辩换鎺旂矆婢舵劖鍋i柛銉у厴濡绢噣鏌涙惔锛勭闁归濞€閺屻劎鈧綆鍓欏銊╂⒑濞茶绨绘い鎴濇喘瀵偊骞樼紒妯轰罕闂佸啿鎼幊澶愬磻閹炬剚娼╂い鎰剁悼濠у嫰姊鸿ぐ鎺撴锭缂佸顕划鈺呭醇閺囩偞宓嶉梺缁樺灦宀e潡宕烘径鎰厽闁冲搫锕ゅ▍娆戠磼閹插瀚�>>  闂備礁鎲¢崙褰掑礈濠靛鏁嬮柍鍝勫€规禍銈夋煕閵夋垵瀚禍閬嶆⒑閹稿海銆掗柛鐘虫礈缁梻鈧稒蓱娴溿倝鏌ㄩ悤鍌涘14濠电姰鍨归悘鍫ュ疾濞戙垺鍋柛鏇ㄥ墻濞堢晫鈧厜鍋撻柍褜鍓熼幃浼村Ψ閳轰胶鐓戦梺鎸庣箓濞诧箓鎮鹃柆宥嗙厾闁告瑥顦辩粻姘舵煕閹炬潙顥嬬紒杈ㄥ浮瀹曞崬顫濋鈧慨鍐⒑閹稿骸鍠曢柟鍑ゆ嫹>>
闂備礁鎲¢崙褰掑礈濠靛鏁嬮柍鍝勫€规禍銈夋煕閵夋垵瀚禍閬嶆⒑閹稿海銆掗柛鐘虫礈缁梻鈧稒蓱娴溿倝鏌ㄩ悤鍌涘14濠电姰鍨归悘鍫ュ疾濞戙垺鍋柛鏇ㄥ墻濞堢晫鈧厜鍋撻柍褜鍓熼幃浼村Ψ閳轰胶鐓戦梺鎸庣箓濞诧箓鎮鹃柆宥嗙厾闁告瑥顦辩粻姘舵煕閹炬潙顥嬬紒杈ㄥ浮瀹曞崬顫濋鈧慨鍐⒑閹稿骸鍠曢柟鍑ゆ嫹>>  生物通微信公众号
生物通微信公众号
知名企业招聘
今日动态 | 人才市场 | 新技术专栏 | 中国科学人 | 云展台 | BioHot | 云讲堂直播 | 会展中心 | 特价专栏 | 技术快讯 | 免费试用
版权所有 生物通
Copyright© eBiotrade.com, All Rights Reserved
联系信箱:
粤ICP备09063491号
