
-

生物通官微
陪你抓住生命科技
跳动的脉搏
棉花遗传改良团队构建首个棉花纤维高分辨率动态3D基因组结构图…
【字体: 大 中 小 】 时间:2022年02月09日 来源:华中农业大学植物科学技术学院
编辑推荐:
相关研究成果以“Dynamic 3D genome architecture of cotton fiber reveals subgenome-coordinated chromatin topology for 4-staged single-cell differentiation”为题发表在国际学术期刊Genome Biology上
南湖新闻网讯(通讯员 裴柳玲)近日,我校棉花遗传改良团队首次构建了棉花纤维的高分辨率三维基因组结构图谱,揭示了亚基因组协作调控异源四倍体棉花纤维发育的拓扑结构基础,对棉花功能基因组研究具有重要推动作用。同时,棉花纤维作为纯净的植物单细胞类型,该研究为解析其他植物单细胞分化的转录调控机制提供了参考。相关研究成果以“Dynamic 3D genome architecture of cotton fiber reveals subgenome-coordinated chromatin topology for 4-staged single-cell differentiation”为题发表在国际学术期刊Genome Biology上。
植物细胞分化伴随着基因转录调控网络重塑,该过程受到表观遗传修饰和染色质结构等影响。棉花纤维是胚珠表皮上高度分化的单细胞结构,其分化发育过程一般经历约50天,分为四个相对重叠阶段:起始分化、伸长、次生细胞壁合成和脱水成熟期。因此,棉花纤维可作为研究植物细胞分化的模式系统。该团队前期的研究发现,随着纤维发育,CHH (H = A、T 或 C) DNA 甲基化水平逐渐增加,伴随着常染色质向异染色质的转换(Nucleic Acids Research, 2016)。随着三维(3D)基因组学技术的发展,该团队利用棉花叶片进行比较三维基因组研究,发现了基因组多倍化和基因组大小演化中染色质高级结构的重组规律(Nature Plants, 2018; Molecular Biology and Evolution, 2021)。
受制于棉花纤维细胞的生理特殊性,普通的Hi-C等实验效果不理想,纤维的三维基因组研究进展缓慢。本研究通过对原位Hi-C技术进行改进,极大地提高了Hi-C实验数据的信噪比。研究中利用海岛棉3-79开花后0天(DPA)、5天(DPA)、10天(DPA)、20天(DPA)的胚珠或纤维样品——分别代表纤维起始分化、伸长(早期伸长和快速伸长)和次生壁合成阶段,进行纤维的三维基因组学研究,旨在解析纤维发育中的染色质拓扑结构改变与基因转录调控之间的关系。
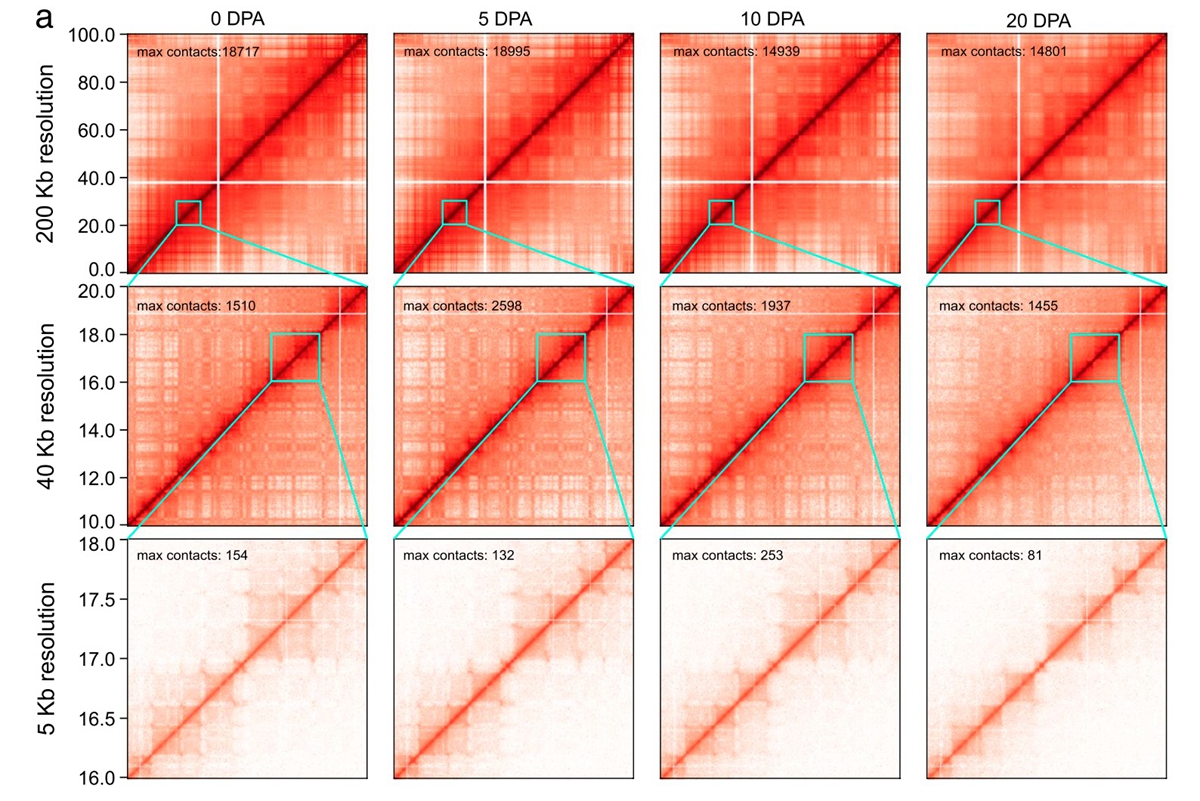
图1. 不同分辨率下棉花纤维的三维基因组结构图谱
研究发现,随着纤维发育,活跃表达的基因变少,同时活性染色质修饰减少,并且At和Dt两个亚基因组对异源四倍体纤维的发育存在协同贡献。在纤维发育过程中,不同的分辨率下纤维细胞核内呈现了不同的染色质高级结构(图1);染色质区室从活跃到不活跃的转换伴随着纤维发育相关基因的沉默。研究鉴定了10571个拓扑关联结构域 (TAD),其中25.6%在不同发育阶段中特异存在,75.23%在两个亚基因组之间发生分割或融合,而这种亚基因组间同源TAD的结构变化与转座子的插入/消除有关。值得注意的是,部分远程 TAD-TAD 相互作用组成的复杂 TAD 团(clique)在纤维发育后期逐渐解散,这是后期染色质高级结构变化的一个突出特征。染色质环介导的转录调控网络在纤维发育过程中发生显著变化,而且在两个亚基因组之间也存在明显差异,这可能与同源基因的偏向性表达相关(图2)。这项研究揭示了四倍体棉花纤维发育中两个亚基因组的时空不对称染色质结构,并为植物细胞阶段性分化的调控机制提供了新的见解。
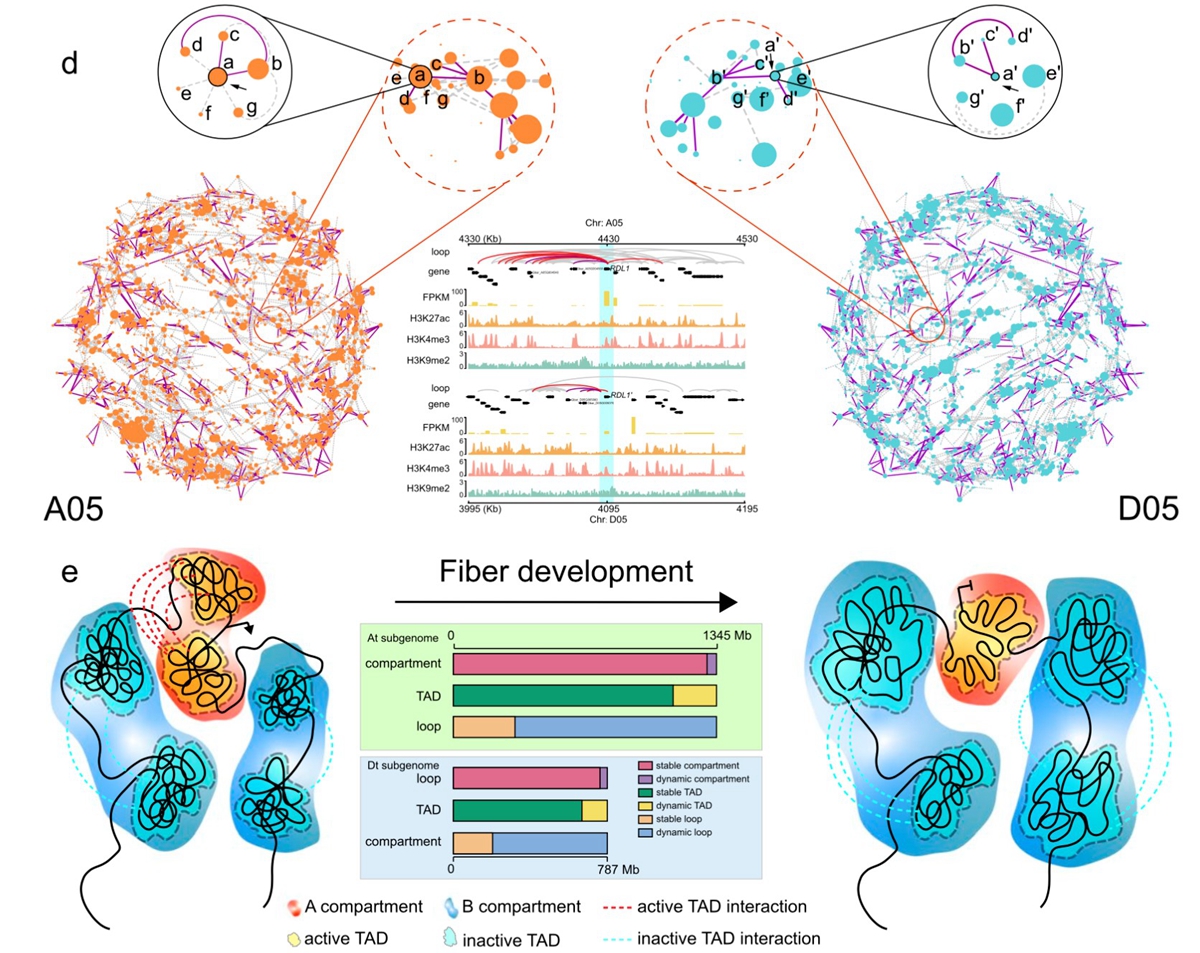
图2. 染色质环介导At和Dt亚组间差异性的调控网络
作物遗传改良国家重点实验室、湖北洪山实验室博士生裴柳玲和黄鲜晖为论文共同第一作者,王茂军教授为通讯作者,张献龙教授参与了课题设计。该研究得到了国家自然科学基金和中央高校基本科研业务费专项资金的资助。
审核人:王茂军
【英文摘要】
Background
Despite remarkable advances in our knowledge of epigenetically mediated transcriptional programming of cell differentiation in plants, little is known about chromatin topology and its functional implications in this process.
Results
To interrogate its significance, we establish the dynamic three-dimensional (3D) genome architecture of the allotetraploid cotton fiber, representing a typical single cell undergoing staged development in plants. We show that the subgenome-relayed switching of the chromatin compartment from active to inactive is coupled with the silencing of developmentally repressed genes, pinpointing subgenome-coordinated contribution to fiber development. We identify 10,571 topologically associating domain-like (TAD-like) structures, of which 25.6% are specifically organized in different stages and 75.23% are subject to partition or fusion between two subgenomes. Notably, dissolution of intricate TAD-like structure cliques showing long-range interactions represents a prominent characteristic at the later developmental stage. Dynamic chromatin loops are found to mediate the rewiring of gene regulatory networks that exhibit a significant difference between the two subgenomes, implicating expression bias of homologous genes.
Conclusions
This study sheds light on the spatial-temporal asymmetric chromatin structures of two subgenomes in the cotton fiber and offers a new insight into the regulatory orchestration of cell differentiation in plants.